Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista Pan-Amazônica de Saúde
versão impressa ISSN 2176-6215versão On-line ISSN 2176-6223
Rev Pan-Amaz Saude v.1 n.3 Ananindeua set. 2010
http://dx.doi.org/10.5123/S2176-62232010000300004
ARTÍCULO ORIGINAL
Epidemiología molecular de los serotipos 2 y 3 del virus dengue, aislados en Brasil de 1991 a 2008
Ana Cecília Ribeiro CruzI; Ricardo GallerII; Eliana Vieira Pinto da SilvaI; Mayra de Oliveira e SilvaI; Adriana Ribeiro CarneiroI; Elizabeth Salbé Travassos da RosaI; Helena Baldez VasconcelosI; Eric Luiz Rodrigues de SáIII; Pedro Fernando da Costa VasconcelosI
ISeção de
Arbovirologia e Febres Hemorrágicas, Instituto Evandro Chagas/SVS/MS,
Ananindeua, Pará, Brasil
IIInstituto de Tecnologia em Imunobiológicos Bio-Manguinhos,
Fundação Oswaldo Cruz, Rio de Janeiro, Rio de Janeiro, Brasil
IIIFundação de Medicina Tropical do Tocantins,
Araguaina, Tocantins, Brasil
Endereço para correspondência
Correspondence
Dirección para correspondencia
Título original: Molecular epidemiology of dengue virus serotypes 2 and 3 isolated in Brazil from 1991 to 2008. Traducido por: Lota Moncada
RESUMEN
El virus dengue (DENV1-4) causa el dengue clásico y la fiebre hemorrágica del dengue / síndrome de choque del dengue (FHD/SCD) en regiones tropicales y subtropicales. El objetivo de este estudio fue el de evaluar los genotipos circulantes de DENV2 y DENV3 obtenidos en distintas regiones geográficas en el período de 1991 a 2008. Los análisis filogenéticos de DENV2 demostraron que el genotipo III (Sudeste de Asia/América), a pesar de las diversas alteraciones nucleotídicas y de aminoácidos, fue el único que circuló durante los últimos 19 años. Desde su introducción en el estudio, en el año 2000, todas las muestras aisladas de DENV3 analizadas, fueron agrupadas en el genotipo III (subcontinente indio). No se encontraron evidencias de que el DENV3 pertenezca a otros genotipos investigados.
Palabras clave: Virus del Dengue; Fiebre Hemorrágica Dengue; Flavivirus.
INTRODUCCIÓN
El dengue clásico (DC) y la fiebre hemorrágica del dengue/Síndrome de choque por dengue (FHD/SCD) son importantes problemas de salud en las regiones tropicales y subtropicales. El número creciente de pacientes con manifestaciones clínicas graves y la expansión de áreas epidémicas ha sido propicio a que surgieran muchas investigaciones acerca de su agente causador, el virus del dengue (DENV), que es el más difundido e importante virus transmitido por mosquitos, en términos de morbilidad y mortalidad1,2,3. Una pandemia global de dengue, que tuvo inicio durante la Segunda Guerra Mundial, se extendió por todo el planeta y alcanzó, predominantemente, a los países tropicales. De acuerdo con la OMS4, la DC causa aproximadamente 5 millones de internaciones de niños y, como mínimo, el FHD/SCD causa 50 mil muertes anuales.
Los serotipos 1 a 4 del DENV (DENV1 a DENV4) pertenecen al género Flavivirus, de la familia Flaviviridae. El genoma de los flavivirus se caracteriza por tener un filamento único de ARN, de sentido positivo y mide aproximadamente 11 Kb de largo, en donde es producida una única poliproteína. Esta poliproteína sufre proteólisis por proteasa celular y viral, de modo a generar 10 proteínas virales, y su orden es 5'-C-prM(M)-E-NS1-NS2A-NS2B-NS3-NS4A-NS4B-NS5-3'5,6.
Las infecciones por el DENV pueden manifestarse con cuadros que varían de asintomáticos a subclínicos, pasando de DC a FHD/CSD, los que pueden observarse durante un brote de dengue. Sin embargo, las infecciones por DENV generalmente llevan a dos síndromes bien conocidas y definidas: DC y FHD. El mecanismo por el cual el huésped infectado por DENV desarrolla la enfermedad, benigna o severa, todavía no ha sido elucidado. El propuesto mecanismo dependiente de anticuerpo, que precipita la exacerbación clínica, sugiere que los anticuerpos circulantes derivados de una infección primaria adhieren a un virus heterólogo en una infección secundaria, lo que facilita la penetración de partículas del virus en células mononucleares7,8. Otros estudios demostraron que l os mediadores del sistema inmunológico del huésped, como las citocinas y complementos, pueden tener una participación directa en la patogénesis de la extravasación de plasma, una característica típica del FHD9,10,11,12.
En Brasil, el DENV2 fue aislado por primera vez en 1989, de un caso importado de Uganda, África. Antes de este caso, los serotipos DENV1 y DENV4 ya habían causado una epidemia en Boa Vista, Estado de Roraima, Brasil13. En 1986, el DENV1 causó epidemias en Rio de Janeiro y en otras ciudades14. En 1990, fue observado el primer brote autóctono de DENV2 en el Estado de Rio de Janeiro. La introducción del serotipo DENV2 resultó en un aumento del número de casos graves de la enfermedad, con el surgimiento de varios casos de FHD/SCD15,16. Además, el serotipo DENV3 fue aislado en el Estado de São Paulo a partir de un caso importado registrado en el año 200017. Un año después de su introducción en Brasil, el DENV3 causó un gran brote de dengue en el Estado de Rio de Janeiro y rápidamente se extendió a varios otros estados de Brasil18.
En este estudio, efectuamos la secuenciación de los genes C, prM/M y E de los serotipos DENV2 y DENV3 en 27 cepas originadas de diferentes áreas geográficas de Brasil y colectadas en distintos períodos entre 1991 y 2008, de modo a investigar la epidemiología molecular de los serotipos que actualmente circulan en el país.
MATERIAL Y MÉTODOS
AISLAMIENTO VIRAL
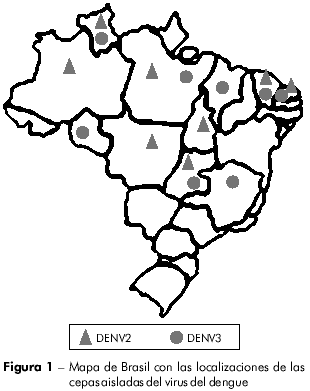
Se obtuvieron veintisiete cepas de DENV (14 de DENV2 y 13 de DENV3) a partir de una colecta de virus realizada por la Sección de Arbovirología y Fiebres Hemorrágicas del Instituto Evandro Chagas (IEC). Los virus fueron aislados de muestras de suero colectadas de pacientes clínicamente diagnosticados como casos de DC, FHD/SCD y encefalitis durante los brotes que ocurrieron entre 1991 y 2008, y de mosquitos capturados en el área endémica (Figura 1). Los virus se cultivaron en células de Aedes albopictus (clone C6/36). Las informaciones relevantes acerca de cada aislado están resumidas en el tabla 1.
EXTRACCIÓN DE ARN Y AMPLIFICACIÓN POR RT-PCR
Las muestras de ARN genómico se extrajeron utilizándose el reactivo Trizol (Invitrogen, EUA), siguiendo las instrucciones del fabricante. Los cDNAs fueron sintetizados y amplificados por RT-PCR en dos etapas, con primers específicos para el DENV2 y el DENV3, que fueron desarrollados para generar productos sobrepuestos correspondientes a la región genómica estructural C/prM/M/E, conforme mencionado anteriormente. Inicia lmente, 0,2-1 µg de ARN viral y 20 µM del primer complementar (antisense) se calentaron a 90o C por 90 s y después se colocaron en hielo. A la mezcla de RT conteniendo una solución tampón de RT 1X (200 mM Tris-HCl [pH 8,4], 500 mM KCl), 0,2 mM de DNTP (deoxinucleótido), 100 mM DTT (ditiotreitol), 40 U de inhibidor de ARNse (Invitrogen, EUA) y 200 U de transcriptasa reversa Superscript II (Invitrogen, USA), se adicionó al ARN precalentado y se ajustó el volumen a 20µL. La reacción RT se realizó a una temperatura de 45o C por 1 h, seguida de un calentamiento a 94o C por 10 min. Los productos de la RT fueron utilizados como moldes para la amplificación por PCR. Las reacciones estaban compuestas por 20µM para cada uno de los primers sense y antisense, tampón de PCR 1X, 1 mM de MgCl2, 0,2 mM de dNTP, 5 U/µL de Platinum Taq Polymerase (Invitrogen) y el volumen final se ajustó para 50 µL con agua libre de ARNse. Las muestras fueron colocadas en un termociclador (Perkin Elmer 9600, EUA) a una temperatura de 94o C por 90 s (para una desnaturación inicial); en seguida, se realizaron 35 ciclos de: 94o C por 30 s, 55o C por 30 s y 72o C por 120 s, y una etapa final de 72o C por 5 min. Los productos de la RT-PCR fueron visualizados en gel de agarosa 1,5% teñido con bromuro de etidio (5 µg/ml).
SECUENCIA NUCLEÓTIDA Y ANÁLISIS SECUENCIALES
Los productos de la RT-PCR fueron purificados con el QIAquick gel extraction kit (Qiagen, Inc., Valencia, CA). El cDNA purificado se utilizó como molde para la secuenciación con el kit Big Dye Terminator 3.0 (Applied Biosystems, Inc., EUA), siguiendo las orientaciones del fabricante. La secuenciación se realizó utilizando el equipo ABI Prism 377 (Applied Biosystems). Las secuencias nucleótidas fueron analizadas y editadas con el software SeqMan (DNASTAR, Lasergene software package). El análisis filogenético se realizó por medio de alineamientos secuenciales progresivos múltiples de pares, utilizando CLUSTAL W (software Megalign; Lasergene software package DNASTAR).
Una secuencia de pares de base abarcando los genes de las proteínas estructurales C-prM-E fue utilizada para comparar los 14 aislados descritos y 22 secuencias anteriormente publicadas de DENV2 colectadas del GenBank (www.ncbi.nlm.nih.gov). Fueron utilizadas un total de 1.977 bases de los genes M y E para realizar la comparación de los 13 aislados con las 27 secuencias previamente publicadas de DENV3. La secuencia de DENV2 (cepa Jamaica, número de acceso M20558) fue usada como un outgroup.
Los análisis filogenéticos se ejecutaron con valores bootstrap de mil seudo réplicas, por el método de neighbor-joining (NJ) Kimura-2-parameter (software MEGA 2.1)19,20,21.
RESULTADOS
ANÁLISIS DE DENV2
Determinamos la secuencia nucleótida completa del genoma estructural de 14 aislados de DENV2 de pacientes con diferentes niveles de gravedad de la enfermedad (Tabla 1). Las diferencias de números de nucleótidos en la comparación entre los virus estudiados y la cepa Jamaica (Jam83) del DENV2, revelaron una gran semejanza (89,0 a 99,9%) de secuencias nucleótidas entre nuestros aislados. La semejanza media entre los subtipos de DENV2 fue de 89,5 y 99,8% para las secuencias de nucleótidos y aminoácidos, respectivamente.
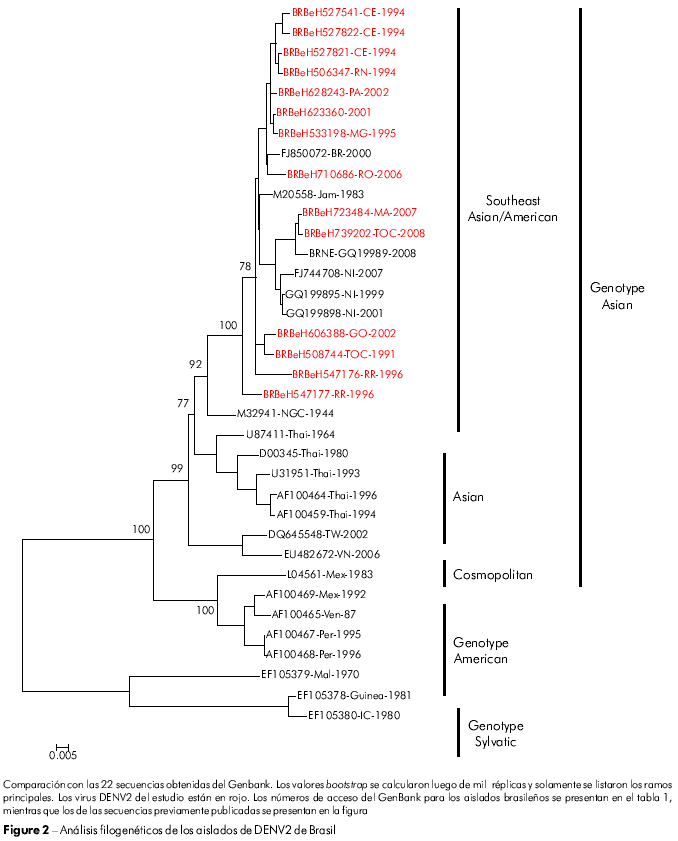
La clasificación de los genotipos adoptada fue la propuesta por Rodriguez-Roche et al22 y Vasilaks e Weaver23, que utilizó el genotipo salvaje como outgroup. El árbol filogenético elaborado para las secuencias nucleótidas C/prM/M/E de nuestros aislados de DENV2 agrupó todos los aislados en el genotipo Jamaica (Figura 2; genotipo III). Además, nuestros aislados de DENV2 fueron relacionados más próximos a la cepa colectada en Brasil (RJ2000; FJ850072) y con una cepa Jamaica de DENV2 (JAM1 983; M20558). El genotipo asiático (III) fue dividido en cuatro linajes diferentes, que representan las cepas provenientes del sudeste de Asia/América, Asia I, Asia II y Asia III.
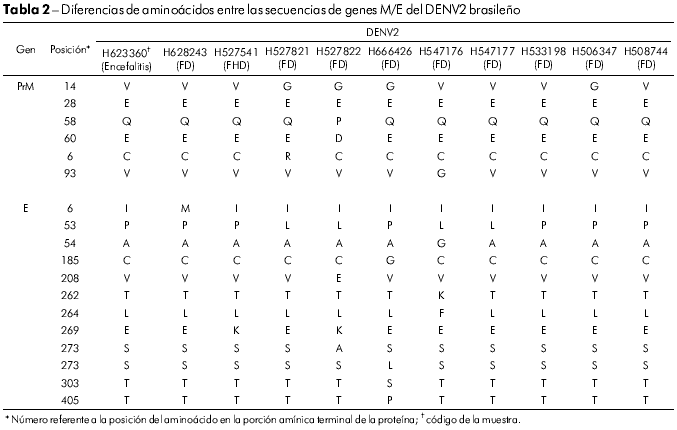
Las secuencias de aminoácidos prM/M/E de los 14 aislados brasileños de DENV2 demostraron que esas proteínas estaban bastante conservadas. Apenas cinco posiciones en los genes prM/M/E variaron en los cinco aislados, siendo que el aminoácido 14 del gen prM, que era una glicina (V → G) en cuatro cepas, fue el que más varió. El aislado BeH527822 acumuló dos cambios en las posiciones prM/M 58 (Q → P) y 60 (E → D), como demostrado en el tabla 2. En el gen E, 12 posiciones de aminoácidos se mostraron variables, siendo que E53 (P → L) y E269 (E → K) se observaron en cuatro y dos aislados, respectivamente. Los aislados más variables fueron BeH527822, BeH666426 y BeH547176, que presentaron cambios en cuatro posiciones. La única que se observó en todos ellos fue la E53 (P → L) ( Tabla 2 ).
ANÁLISIS DE DENV3
La homología de las secuencias nucleótidas entre las cepas del virus DENV3 varió entre 89,7 y 100%. Las secuencias deducidas de aminoácidos en las posiciones prM/M y E en todos los 10 aislados de DENV3, mostraron que sus proteínas se presentaban bastante conservadas, con una tasa de similitud variando entre 93,2% y 100%. La homología secuencial media fue de 94,8% y 96,6% para las secuencias de nu cleótidos y aminoácidos, respectivamente.
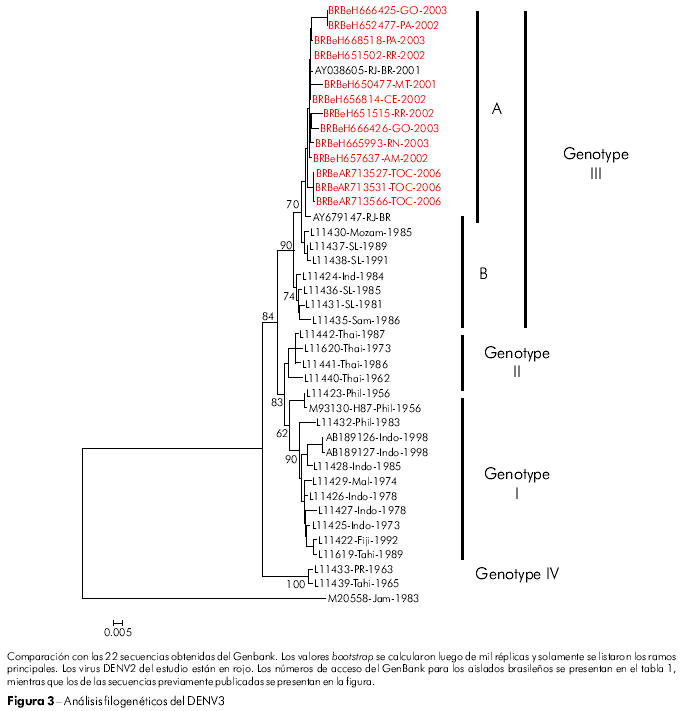
El árbol filogenético desarrollado para los aislados de DENV3 tuvo como base la alineación de secuencias observada en la región E/NS1 (992-2550 nt), utilizándose los criterios de división de los genotipos descritos por Lanciotti24. Todos los aislados de DENV3 fueron identificados como miembros del genotipo III (subcontinente indio) y fueron agrupados en dos subclados (A y B). El subclado A contiene las cepas latinoamericanas y se considera genéticamente más próximo a las cepas del subclado B aisladas en Sri Lanka (L11437, L11438) (Figura 3).
La variabilidad de las secuencias de aminoácidos para los aislados de DENV3 quedó restricta a una posición en el gen M (M86 H → R), mientras que se encontraron variaciones en 6 posiciones en el gen E (Tabla 3). Las posiciones que presentaron más variaciones fueron E6 (l → V), E89 (Q → P) y E246 (Q → P). Los aislados BeH657637 y BeH665993 acumularon cambios en tres de las seis posiciones variables de los aminoácidos (Tabla 3).

DISCUSIÓN
La divergencia entre secuencias nucleótidas (10,5%) para el NV2 obtenida en este estudio sugiere una acumulación de mutaciones en el período de alta circulación de este serotipo del DENV25. De hecho, el DENV2 circula en Brasil hace casi dos décadas; sin embargo, sorprendentemente, ha habido baja incidencia de casos de FHD cuando comparado con otros países. Desde la introducción del DENV3, en 2002, el número de casos de FHD ha aumentado drásticamente, lo que sugiere una alta virulencia de este serotipo26. Estudios anteriores sobre el DENV2 en las Américas había ya demostrado que la emergencia del FHD está relacionada a la introducción de un genotipo asiático en el Caribe27. Nuestros datos confirman que este genotipo del DENV2 ha sido predominante durante los 19 años de circulación del DENV en Brasil.
Según la clasificación propuesta por Rodriguez-Roche et al22 y Vasilaks & Weaver23, el DENV2 se agrupa en tres genotipos, siendo que sus genotipos III y IV circulan en las Américas. El genotipo III circuló en todos os países de las Américas y tuvo como prototipo a la cepa Jamaica, que ha sido asociada a casos de FHD. El genotipo IV (americano) ha sido asociado apenas a cepas de virulencia más baja y a la DC. Es importante observar que este genotipo ha circulado solamente en el subcontinente americano28. En nuestro estudio, ninguna de las secuencias brasileñas analizadas se incluyó en el genotipo IV (americano) del DENV2. En realidad, todas las secuencias fueron agrupadas en el genotipo III (Figura 2).
Los primeros estudios comparando la secuencia nucleótida del gen de 12 aislados de DENV2 no mostraron ninguna correlación entre la gravedad de la enfermedad y la secuencia de nucleótidos o de aminoácidos29. Ni aun estudios más detallados sobre el genoma completo del genotipo del DENV2 del sudeste de Asia han conseguido identificar sitios específicos que determinaran su virulencia30,31. Leitmeyer at al28 realizaron un análisis más consistente de la diversidad genética del DENV2. Este estudio identificó una sustitución del aminoácido en la posición E390 como determinante primario del dengue grave. En el caso del genotipo americano, se descubrió que los casos de DC estaban asociados a la presencia de ácido aspártico (Asp-D), que puede alterar las interacciones con los receptores celulares. Sin embargo, los resultados obtenidos en 14 secuencias de DENV2 en nuestro estudio, identificaron un Asn en la posición E390 en todas las secuencias analizadas, cuando comparadas a las 14 cepas y a las cepas de los genotipos asiático y americano28. Los resultados indican que el DENV2 brasileño analizado tiene el potencial de causar FHD, lo que es consistente con los datos presentados por investigaciones con aislados originados de Venezuela32.
El cambio del aminoácido E390 está localizado en la región carboxílica terminal III (aa 303-395) en la superficie lateral, que debe contener los residuos involucrados en el tropismo y en la virulencia de diferentes flavivirus33,34,35. En el gen E de 14 virus brasileños analizados, se observan 12 cambios y apenas cinco sustituciones de aminoácidos se asociaron de forma significativa a un cambio de carácter y polaridad. Esas posiciones fueron: E208 (Val/V → Glu/E), 262 (Thr/T → Lys/K), 273 (Ser/S → Leu/L), 274 (Ser/S → Ala/A) y 405 (Thr/T → Pro/P). Todas los cambios de aminoácidos se presentan en el tabla 2. Los cambios se observaron en el ectodominio III o fuera del ectodominio de la proteína, como en el residuo 405 de la muestra H666426/Goi4191. Algunos cambios presentados en el tabla 2, como el E262 y 264, que se observan en H547176/ROR1811 y en la cepa viral originaria de Venezuela (AF100466), sugieren que el virus se haya originado en aquel país, confirmando así la existencia de dos formas de introducción del DENV2 en Brasil.
En las dos últimas décadas, el DENV3 ha causado epidemias de FHD en el sudeste de Asia, este de África y América Latina. El primer análisis filogenético de la proteína E de este serotipo del dengue resultó en la identificación de cuatro genotipos distintos24. En general, los genotipos pueden agruparse según su origen geográfico: Américas, Subcontinente indio, Tailandia y sudeste de Asia/Pacífico Sur25.
El genotipo de DENV3 que circuló en las Américas hasta 1989 presentaba un bajo potencial epidémico y fue aislado apenas en pacientes con DC. Entre 1980 y 1990, dos nuevos genotipos fueron introducidos en la región del Pacífico Sur y en las Américas: el genotipo del sudeste de Asia (genotipo I) está asociado a grandes epidemias de FHD en Tahití y en Fiji36, y el genotipo del Subcontinente indio (genotipo III) fue introducido en América Central en la década de 1990 por Nicaragua37,38,39,40.
En 1994, el DENV3 resurgió en las Américas, causando un pequeño brote asociado a la DC en Panamá. El virus se extendió en dirección al norte de América Central hasta Nicaragua y México41,2. Siete años después de su introducción en las Américas, el DENV3 se extendió por América del Sur hasta Venezuela, Paraguay y Brasil, causando grandes epidemias42,43,17,44,45.
Los 13 aislados brasileños de DENV3 de nuestro estudio fueron colectados en diferentes regiones, y, junto a los aislados obtenidos en países vecinos45,46,40, todos se agruparon en el genotipo III (Subcontinente indio). Son genéticamente distintos a la cepa de DENV3 que circuló en las Américas en la década de 1960, que pertenecía al genotipo V47. Es posible que esta variabilidad acumulada sea el resultado de su rápida dispersión y del aumento en su replicación viral. Esta hipótesis está corroborada por los valores bootstrap de aproximadamente 84% mostrados por el árbol filogenético desarrollado según el método neighbor-joining (Figura 3). La variabilidad de la cepa de DENV3 (genotipo III) puede adjudicarse a sus múltiples introducciones en Brasil, lo que propició la clasificación en subgrupos de acuerdo al origen y año de aislamiento de su cepa viral (Figura 3).
La transmisión local del DENV3 fue detectada inicialmente en el Estado de Rio de Janeiro en diciembre de 2000. En el verano de 2001, ocurrió la primera epidemia autóctona en la Ciudad de Rio de Janeiro, con un alto número de casos de FHD. En seguida, hubo diseminación de este genotipo por todo Brasil. Todos los virus aislados pertenecían al genotipo III, semejante al que circulaba en Sri Lanka, corroborando lo que ya había sido descrito en estudios anteriores47,48,49. Con base en la cronología del aislamiento del virus, nuestros resultados demuestran que la dispersión del genotipo III del virus DENV3 en Brasil ocurrió en sentido Sudeste-Norte. Esos hallazgos difieren de los presentados por Figueiredo et al50, que apuntaban a la circulación del genotipo I en el Estado de Minas Gerais, Sudeste de Brasil, y por Araújo et al51, que sugería la formación de un nuevo (V), que agruparía las cepas de DENV3 de Brasil, China y Japón. Las diferencias en los resultados muestran que la circulación de nuevos genotipos pareció estar restricta a una región específica de Brasil o a un cierto período de tempo. Este hecho hace surgir algunas cuestiones importantes: ¿Por qué otras cepas de estos genotipos no pudieron ser identificadas? ¿Esta cepa reserva un potencial de transmisión? Por lo tanto, la emergencia y el surgimiento de otros o nuevos genotipos debe ser más investigada. Estos estudios deben incluir el análisis de las secuencias completas del genoma del virus DENV3 y de su perfil epidemiológico.
CONCLUSIÓN
Nuestro estudio sugiere que la baja variabilidad genética del DENV2 circulante en Brasil puede explicar la baja incidencia de casos graves de la enfermedad registrados hasta el presente momento. Sin embargo, la diseminación masiva del genotipo III del DENV2, que presentó una variabilidad mayor que el DENV3, puede haber ocasionado una acumulación de cambios genéticos asociados al aumento de la virulencia, al estatus inmunológico y a otros factores importantes descritos. Este hecho podría explicar el gran número de casos graves de dengue desde la introducción de DENV2 y DENV3 en Brasil.
AGRADECIMIENTOS
Agradecemos la valiosa contribución de los Drs. Márcio Roberto Teixeira Nunes, Ralph Lainson y Elena Caride, Creuza Lima Carvalho y Maria Natividade.
APOYO FINANCIERO
Esta investigación fue parcialmente financiada por el Consejo Nacional de Desarrollo Científico y Tecnológico, Brasil (CNPq) (becas 300460/2005-8 y 501558/2003-9).
REFERENCIAS
1 Monath TP, Heinz FX. Flaviviruses. In: Fields BN, Knipe DM, Howley PM, editors. Field's Virology. Philadelphia: Lippincott Williams Wilkins; 1996. p. 961-1034.
2 Guzmán MG, Kouri G. Dengue: an update. Lancet Infect Dis. 2002 Jan;2(1):33-42.
3 Kourí G. El dengue, un problema cresciente de salud en las Américas. Rev Panam Salud Publica. 2006;19(3):143-5. [ Links ]
4 World Health Organization. Bulletin of Dengue. 1986
5 Chambers TJ, Hahn CS, Galler R, Rice CM. Flavivirus genome organization, expression, and replication. Annu Rev Microbiol. 1990;44:649-88.
6 Rice CM. Flaviviridae: the viruses and their replication. In: Fields BN, Knipe DM, Howley PM, editors. Fields Virology. Philadelphia: Lippincott Williams Wilkins; 1996. p. 931-60.
7 Halstead SB. Pathogenesis of dengue: challenges to molecular biology. Science. 1988 Jan;239(4839):476-81. [ Links ]
8 Littaua R, Kurane I, Ennis FA. Human IgG Fc receptor II mediates antibody-dependent enhancement of dengue virus infection. J Immunol. 1990 Apr;144(8):3183-6. [ Links ]
9 Kurane I, Ennis FE. Immunity and immunopathology in dengue virus infections. Semin Immunol. 1992 Apr;4(2):121-7. [ Links ]
10 Rothman AL, Ennis FA. Imunnopathogenesis of dengue haemorrhagic fever. Virology. 1999;257:1-6.
11 Chaturvedi UC, Argwal R, Elbishbishi EA, Mustafá AS. Cytokine cascade in dengue haemorrhagic fever: inplications for pathgenesis. FEMS Immunol Med Microbiol. 2000 Jul;28(3):183-8. [ Links ]
12 Rothman AL. Dengue: defining protective versus pathologic immunity. J Clin Invest. 2004 Apr;113(7):946-51.
13 Travassos da Rosa APA, Travassos da Rosa JFS, Pinheiro FP, Vasconcelos PFC. Arboviroses. In: Leão RNQ, editor. Doenças Infecciosas e Parasitárias. Belém: CEJUP; 1997. p. 207-26.
14 Schatzmayr HG, Nohueira RM, Travassos da Rosa AP. An outbreak of dengue virus at Rio de Janeiro – 1986. Mem Inst Oswaldo Cruz. 1986 Apr-Jun;81(2):245-6. [ Links ]
15 Nogueira RM, Miagostovich MP, Lampe E, Souza RW, Zagne SM, Schatzmayr HG. Dengue epidemic in the stage of Rio de Janeiro, Brazil, 1990-1: co-circulation of dengue 1 and dengue 2 serotypes. Epidemiol Infect. 1993 Aug;111(1):163-70. [ Links ]
16 Zagne SM, Alves VG, Nogueira RM, Miagostovich MP, Lampe E, Tavares W. Dengue haemorrhagic fever in the state of Rio de Janeiro, Brazil: a study of 56 confirmed cases. Trans R Soc Trop Med Hyg. 1994 Nov-Dec;88(6):677-9.
17 Rocco IM, Kavakama BB, Santos CL. First isolation of dengue 3 in Brazil from an imported case. Rev Inst Med Trop Sao Paulo. 2001 Jan-Feb;43(1):55-7.
18 Siqueira Jr JB, Martelli CMT, Coelho GE, Simplício ACR, Hatch DL. Dengue and dengue hemorrhagic fever, Brazil, 1981-2002. Emerg Infect Dis. 2005 Jan;11(1):48-53.
19 Kumar S, Tamura K, Nei M. MEGA3: integrated software for molecular evolutionary genetics analysis and sequence alignment. Brief Bioinform. 2004 Jun;5(2):150-63. [ Links ]
20 Saitou N, Nei M. The neighbor-joining method: a new method for reconstructing phylogenetic trees. Mol Biol Evol. 1987 Jul;4(4):406-25. [ Links ]
21 Kimura M. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequence. J Mol Evol. 1980 Dec;16(2):111-20.
22 Rodriguez-Roche R, Alvarez M, Gritsun T, Halstead S, Kouri G, Gould EA, et al. Virus evolution during a severe dengue epidemic in Cuba, 1997. Virology. 2005;334(2):154-9.
23 Vasilakis N, Weaver SC. History and evolution of human dengue emergence. In: Maramorosch K, Shatkin AS, Murphy FA, editors. Advances in Virus Research. California: Academic Press; 2008. p. 1-76.
24 Lanciotti RS, Lewis JG, Gubler D, Trent DW. Molecular evolution and epidemiology of dengue 3 viruses. J Gen Virol. 1994 Jan;75(1):65-75. [ Links ]
25 Rico-Hesse R. Microevolution and virulence of dengue virus. In: Chambers TJ, Monath TP, editors. The Flaviviruses: structure, replication and evolution. California: Academic Press; 2003. p. 316-41.
26 Secretaria de Vigilância em Saúde. Plano Nacional de Combate a Dengue. Brasília: Ministério da Saúde; 2006.
27 Rico-Hesse R, Harrison LM, Salas RA, Tovar D, Nisalak A, Ramos C, et al. Origins of dengue type 2 viruses associated with increased pathogenicity in the Americas. Virology. 1997 Apr;230(2):224-51.
28 Leitmeyer KC, Vaughn DW, Watts DG, Salas R, Chacon IV, Ramos C, et al. Dengue virus structural differences that correlate with pathogenesis. J Virol. 1999 Jun;73(6):4738-47. [ Links ]
29 Block J, Samuel S, Gibbs AJ, Vitarana UT. Variation of the nucleotide and encoded amino acid sequences of the envelope gene from eight dengue 2 viruses. Arch Virol. 1989;105(1-2):39-53. [ Links ]
30 Mangada MN, IgarashI A. Molecular and in vitro analysis of eigth dengue type 2 viruses isolated from patients exhibiting different disease severities. Virology. 1998 May;244(2):458-66.
31 Pandey BD, Igarashi A. Severity-related molecular differences among nineteen strains of dengue type 2 viruses. Microbiol Immunol. 2000 Feb;44(3):179-88. [ Links ]
32 Uzcategui NY, Camacho D, Comach G, Cuello de Uzcategui R, Holmes EC, Gould EA. Molecular epidemiology of dengue type 2 virus in Venezuela: evidence for in situ virus evolution and recombination. J Gen Virol. 2001 Dec;82(12):2945-53. [ Links ]
33 Hasegawa HM, Yoshida T, Shiosaka S, Kobayashi Y. Mutations in the envelope protein of Japanese encephalitis virus affect entry into cultured cells and virulence in mice. Virology. 1992 Nov;191(1):158-65. [ Links ]
34 Jiang WR, Lowe A, Higgs S, Reid S, Gould EA. Single amino acid codon changes detected in louping ill virus antibody-resistant mutants with reduced neurovirulence. J Gen Virol. 1993 May;74(5):931-5. [ Links ]
35 Mandl CW, Guirakhoo F, Holzmann H, Heinz FX, Kunz C. Antigenic structure of the flavivirus envelope protein E at the molecular level, using tick-borne encephalitis virus as a model. J Virol. 1989 Feb;63(2):564-71. [ Links ]
36 Chungue E, Deubel V, Cassar O, Laille M, Martin PMV. Molecular epidemiology of dengue 3 viruses and genetic relatedness among dengue 3 strains isolated from patients with wild or severe form for dengue fever in French Polynesia. J Gen Virol. 1993 Dec;74(12):2765-70.
37 Balmaseda A, Sandoval E, Perez L, Gutierrez CM, Harris E. Application of molecular typing techniques in the 1998 dengue epidemic in Nicaragua. Am J Trop Med Hyg. 1999 Dec;61(6):893-7.
38 Harris E, Roberts TG, Smith L, Selle J, Kramer LD, Valle S, et al. Typing of dengue viruses in clinical specimens and mosquitoes by single-tube multiplex reverse transcriptase PCR. J Clin Microbiol. 1998 Sep;36(9):2634-9. [ Links ]
39 Harris E, Videa E, Perez L, Sandoval E, Tellez Y, Perez ML, et al. Clinical, epidemiologic, and virologic features of dengue in the 1998 epidemic in Nicaragua. Am J Trop Med Hyg. 2000 Jul-Aug;63(1- 2):5-11. [ Links ]
40 Usuku S, Castillo L, Sugimoto C, Noguchi Y, Yogo Y, Kobayashi N. Phylogenetic analysis of dengue 3 viruses prevalent in Guatemala during 1996-1998. Arch Virol. 2001 Jul;146(7):1381-90. DOI:10.1007/s007050170098 [ Links ]
41 Centers for Disease Control and Prevention. Dengue type 3 infection – Nicaragua and Panama, October-November 1994. MMWR Morb Mortal Wkly Rep. 1995 Jan;44(2):21-4. [ Links ]
42 Halstead SB. Dengue in the Americas and Southeast Asia: do they differ? Rev Panam Salud Publica. 2006 Dec;20(6):407-15. [ Links ]
43 Nogueira RMR, Miagostovich MP, Filippis AM, Pereira MA, Schatzmayr HG. Dengue virus type 3 in Rio de Janeiro, Brazil. Mem Inst Oswaldo Cruz. 2001 Oct;96(7):925-6. DOI:10.1590/S0074-02762001000700007 [ Links ]
44 Simone TS, Nogueira RM, Araújo ES, Guimarães FR, Santos FB, Schatzmayr HG, et al. Dengue virus surveillance: the co-circulation of DENV1, DENV2 and DENV3 in the State of Rio de Janeiro, Brazil. Trans R Soc Trop Med Hyg. 2004 Sep;98(9):553-62. [ Links ]
45 Aquino VH, Anatriello E, Gonçalves PF, Silva EV, Vasconcelos PF, Vieira DS, et al. Molecular Epidemiology of dengue type 3 virus in Brazil and Paraguay, 2002-2004. Am J Trop Med Hyg. 2006;75(4):710-5. [ Links ]
46 Alvarez M, Rodriguez-Roche R, Bernardo L, Vázquez S, Morier L, Gonzalez D, et al. Dengue Hemorrhagic Fever caused by sequential dengue 1-3 virus infections over a long time interval: Havana epidemic, 2001-2002. Am J Trop Med Hyg. 2006;75(6):1113-7. [ Links ]
47 Uzcategui NY, Comach G, Camacho D, Salcedo M, Cabello de Quintana M, Jimenez M, et al. Molecular epidemiology of dengue virus type 3 in Venezuela. J Gen Virol. 2003 Jun;84(6):1569-75. [ Links ]
48 Messer WB, Gubler DJ, Harris E, Sivananthan K, Silva AM. Emergence and global spread of a dengue serotype 3, subtype III virus. Emerg Infect Dis. 2003 Jul;9(7):800-9. [ Links ]
49 Nogueira RM, Schatzmayr HG, Filippis AM, Santos FB, Cunha RV, Coelho JO, et al. Dengue virus type 3, Brazil, 2002. Emerg Infect Dis. 2005 Sep;11(9):1376-81. [ Links ]
50 Figueiredo LB, Cecílio AB, Ferreira GP, Drumond BP, Oliveira JG, Bonjardim CA, et al. Dengue virus 3 genotype 1 associated with dengue fever and dengue hemorrhagic fever, Brazil. Emerg Infect Dis. 2008 Feb;14(2):314-6. [ Links ]
51 Araújo JMG, Bello G, Schatzmayr HG, Santos FB, Nogueira RMR. Dengue virus type 3 in Brazil: a phylogentic perspective. Mem Inst Oswaldo Cruz. 2009;104(3):526-9.
 Correspondência
/ Correspondence / Correspondencia:
Correspondência
/ Correspondence / Correspondencia:
Ana Cecília Ribeiro Cruz
Instituto Evandro Chagas,
Seção de Arbovirologia e Febres Hemorrágicas
Rodovia BR 316, km 7, s/nº. Bairro: Levilândia
CEP: 67030-000 Ananindeua-Pará-Brasil
Tel.: 55 (91) 3217-3199 | Fax: 55 (91) 3226-5262
E-mail:anacecilia@iec.pa.gov.br
Recebido em / Received / Recibido en: 31/7/2009
Aceito em / Accepted / Aceito en: 1/2/2010










 texto em
texto em 
 Curriculum ScienTI
Curriculum ScienTI