Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista Pan-Amazônica de Saúde
versão impressa ISSN 2176-6215versão On-line ISSN 2176-6223
Rev Pan-Amaz Saude v.3 n.4 Ananindeua dez. 2012
http://dx.doi.org/10.5123/S2176-62232012000400002
ARTIGO ORIGINAL | ORIGINAL ARTICLE| ARTÍCULO ORIGINAL
Polimorfismo do gene humano NRAMP1, níveis de anticorpos anti-PGL-1 e suscetibilidade para hanseníase em áreas endêmicas do Estado do Pará, Brasil
Polymorphism of human NRAMP1 gene, levels of anti-PGL-1 antibodies and susceptibility to leprosy in endemic areas of Pará State, Brazil
Polimorfismo del gen humano NRAMP1, niveles de anticuerpos anti-PGL-1 y susceptibilidad a la lepra en áreas endémicas del Estado de Pará, Brasil
Maria do Perpétuo Socorro Amador Silvestre; Luana Nepomuceno Gondim Costa Lima; André Brandão de Araújo; Juarez Antônio Simões Quaresma
Laboratório de Hanseníase, Seção de Bacteriologia e Micologia, Instituto Evandro Chagas/SVS/MS, Ananindeua, Pará, Brasil
Endereço para correspondência
Correspondence
Dirección para correspondencia
RESUMO
O gene humano de resistência natural que codifica uma proteína detectada no macrófago - NRAMP1 parece estar envolvido com a influência no padrão de resposta imune à infecção por Mycobacterium leprae. Foi avaliada a associação do polimorfismo deste gene (3'UTR ins/del CAAA) com a hanseníase per se e com os tipos da doença, também relacionada com os níveis de anticorpos anti-PGL-1, representando o padrão de resposta imune humoral (tipo TH2) na população estudada. Um total de 122 pacientes com hanseníase e 110 não doentes, procedentes de municípios endêmicos do Estado do Pará, Brasil, foram genotipados para este polimorfismo e analisados segundo os níveis de anticorpos anti-PGL-1 desta micobactéria. Observou-se associação com a hanseníase per se (p = 0,0087), e o polimorfismo da região 3' não traduzida do gene NRAMP1 com inserção/deleção de quatro pares de bases (CAAA) foi fortemente associado com a forma multibacilar (p = 0,025) comparada ao grupo de contatos não consanguíneos. Heterozigotos e portadores do alelo com a deleção (159 pb) foram mais frequentes entre os casos multibacilares do que nos paucibacilares. Os haplótipos do gene NRAMP1 avaliados neste estudo parecem exercer influência importante na apresentação clínica da hanseníase, determinada também pela positividade ao antígeno PGL-1 do M. leprae, expressão da resposta imune humoral na hanseníase.
Palavras-chave: Genética; Hanseníase; Polimorfismo Genético.
ABSTRACT
The human gene of natural resistance which encodes a protein detected in macrophage - NRAMP1 seems to be involved in the influence on the pattern of immune response to infection by Mycobacterium leprae. It was evaluated the association of the polymorphism of this gene (3'UTR ins/del CAAA) in leprosy per se with types of that disease, also related to the levels of anti-PGL-1 antibodies, representing the pattern of humoral immune response (TH2 type) in this population. A total of 122 leprosy patients and 110 non-patients, coming from endemic municipalities in Pará State, Brazil, were genotyped for this polymorphism and analyzed according to the levels of anti-PGL-1 antibodies of this mycobacterium. An association was observed in leprosy per se (p = 0.0087), and polymorphism of the 3' untranslated region of the NRAMP1 gene with insertion/deletion of four base pairs (CAAA) was strongly associated with the multibacillary form (p = 0.025) compared to the non-consanguineous group of contact. Heterozygous and allele carriers with deletion (159 bp) were more frequent among multibacillary than in the paucibacillary forms. The haplotypes of NRAMP1 gene, in this study, have an important influence on clinical presentation of leprosy, also determined by the positivity to PGL-1 antigen of M. leprae, expression of humoral immune response in leprosy.
Keywords: Genetics; Leprosy; Polymorphism, Genetic.
RESUMEN
El gen humano de resistencia natural que codifica una proteína detectada en el macrófago - NRAMP1 parece estar ligado a la influencia en el patrón de respuesta inmune a la infección por Mycobacterium leprae. Evaluamos la asociación del polimorfismo de este gen (3'UTR ins/del CAAA) con la lepra per se y con los tipos de enfermedad, también relacionados a los niveles de anticuerpos anti-PGL-1, representando un patrón de respuesta inmune humoral (tipo TH2) en la población estudiada. Un total de 122 pacientes con lepra y 110 no enfermos, procedentes de municipios endémicos del Estado de Pará, Brasil, fueron genotipados para este polimorfismo y analizados según los niveles de anticuerpos anti-PGL-1 de esta micobacteria. Observamos una asociación con la lepra per se (p = 0,0087) y el polimorfismo de la región 3' no traducido del gen NRMP1, con inserción/deleción de cuatro pares de bases (CAAA), fuertemente asociado a la forma multibacilar (p = 0,025), comparado con el grupo de contactos no consanguíneos. Los heterocigotos y los portadores del alelo con deleción (159 pb) fueron más frecuentes entre los casos multibacilares que en los paubacilares. Los haplótipos del gen NRAMP1 evaluados en este grupo parecen ejercer una influencia importante en la presentación clínica de la lepra, determinada también por la positividad al antígeno PGL-1 del M. leprae, expresión de la respuesta inmune humoral en la enfermedad de Hansen.
Palabras clave: Genética; Lepra; Polimorfismo Genético.
INTRODUÇÃO
Hanseníase é doença infecciosa crônica, causada pelo bacilo de Hansen, descoberto em 1983, no século XIX. O bacilo possui tropismo por pele, anexos cutâneos e, principalmente, para terminações nervosas da pele e nervos periféricos, o que pode acarretar aos portadores da doença incapacidades físicas irreversíveis quando o diagnóstico é tardio.
A hanseníase ainda representa problema de saúde pública no mundo, com notificação de 219.075 casos novos em 105 países, em 2012, dos quais 127.295 detectados na índia, seguida do Brasil, com 33.955 e da Indonésia, com 20.0231.
Desde 1997 foi priorizado, como indicador epidemiológico para monitoramento da endemia no Brasil, o coeficiente de detecção de casos novos em menores de 15 anos de idade, já que ele identifica focos de transmissão ativa da doença. Essa definição foi recomendada pela Organização Mundial da Saúde a partir de reunião ocorrida em Nova Deli (índia), em 20091.
No ano de 2011, observou-se redução dos coeficientes de detecção em menores de 15 anos de idade e na população em geral do Brasil, porém ainda com taxas elevadas nas Regiões Norte, Nordeste e Centro-Oeste. Nesse mesmo ano, o coeficiente de detecção no Brasil, para cada 100.000 habitantes, foi de 17,6. O Estado do Pará é considerado o primeiro do Brasil em número absoluto de casos, tendo apresentado, em 2012, segundo informações da Coordenação Estadual do Programa de Controle da Hanseníase - PCH/SINAN-Net, 4.076 casos novos, com coeficiente de detecção de 7.431/100.000 habitantes e prevalência de 5.212 casos, com taxa de 7.01 para cada 10.000 habitantes1,2.
A descoberta de instrumentos diagnósticos capazes de detectar infecção por Mycobacterium leprae antes das manifestações clínicas é uma meta importante para os países endêmicos em hanseníase, objetivando o diagnóstico precoce e prevenção das incapacidades físicas3,4.
O diagnóstico e a classificação dos casos de hanseníase, realizados no campo ou na rede básica dos serviços de saúde, é feito basicamente por meio de dados clínicos, bacteriológicos, por meio da baciloscopia do esfregaço cutâneo e, às vezes, histopatológicos, quando estes métodos encontram-se acessíveis.
Não há disponível, no campo ou nos serviços de saúde, teste laboratorial sensível e específico que possa detectar infecção assintomática pelo M. leprae ou, ainda, predizer risco de adoecimento por formas graves da hanseníase e monitorar a evolução do estágio, de infectado para infeccioso, ou seja, para doença em atividade clínica3,4,5,6,7,8,9,10,11.
Estudos para análise da contribuição do fator genético na manutenção da endemia hansênica em algumas áreas do mundo, têm sido desenvolvidos e a utilização de marcadores genéticos moleculares foram empregados até o momento, não só para determinação da influência do componente genético na hanseníase per se, mas também no padrão de resposta imune do hospedeiro à infecção, bem como o complexo mecanismo de efeitos pleitrópicos dos genes candidatos polimórficos, presentes na população de indivíduos infectados e com doença clínica instalada12,13,14,15.
A NRAMP1 (resistência natural associada à proteína macrofágica) define uma família de proteínas extremamente conservada durante a evolução, distribuída em grupos evolutivos que variam da bactéria ao homem, sugerindo participação fundamental em todos os organismos vivos16,17,18.
O gene NRAMP1 localiza-se no cromossomo 2, na região q35, e contém uma fita com 15 éxons e extensão perfazendo tamanho de aproximadamente 14 kb19,20. Em camundongos, uma mutação recessiva no gene NRAMP1, causando a substituição de glicina por ácido aspártico na posição 169 do quarto domínio transmembrânico, resulta em suscetibilidade a alguns patógenos intracelulares, entre eles o Mycobacterium bovis17,18.
Este gene humano NRAMP1 (homólogo murino) é um dos genes associados à resistência/suscetibilidade do hospedeiro à infecção com M. leprae. Este gene codifica uma proteína integral de membrana de 60 Kda, com 12 domínios transmembrânicos, que se localiza em fagolisossomas de macrófagos. A proteína possui vários sítios de fosforilação e alças extracelulares glicosiladas. Há evidências de que sua função seja de canal iônico, transportando íons divalentes pela membrana21,22,23,24,25,26.
O transporte de íons para fora do fagolisossoma é dependente de pH. A diminuição do conteúdo iônico dentro do fagolisossoma, principalmente ferro (Fe2+), manganês (Mn2+) e zinco (Zn2+), controlaria a proliferação de microorganismos intracelulares em fagócitos, visto que os patógenos utilizam estes íons como cofatores para replicação do DNA, produção de importantes enzimas para seu metabolismo e neutralização de componentes tóxicos do fagolisossoma e também expressão de diferentes fatores de virulência12,21.
De acordo com Buu et al20, um polimorfismo de 4 pb resultantes da inserção/deleção de uma trinca de nucleotídeos CAAA de um elemento Alu foi detectada na extremidade 3' em estudo de análise do gene humano NRAMP1. Segundo os autores, o efeito fisiológico desse polimorfismo não está totalmente entendido. Contudo há evidências de elementos regulatórios dentro das 3'UTR de vários genes. Na ausência de efeito fisiológico bem definido, um marcador elevadamente polimórfico descrito pode ser proveitoso em estudos de associação genética, para avaliar o papel do gene humano NRAMP1 na suscetibilidade para doenças como tuberculose, hanseníase e leishmaniose17,20.
O interesse no estudo da associação do polimorfismo do gene NRAMP1 com suscetibilidade para hanseníase é a influência que ele exerce na apresentação antigênica às células TCD4+, não somente por meio da expressão de moléculas de MHC classe II, mas também pela regulação do processo de apresentação de antígenos e, consequentemente, a determinação do padrão de resposta imune do hospedeiro à infecção com M. leprae12,17,18,26.
A hanseníase é considerada uma doença complexa do ponto de vista genético, posto que ela não apresenta padrão de herança mendeliana clássica, em que há perfeita correlação entre genótipo e fenótipo, mas sua ocorrência pode estar sendo controlada por ação conjunta de números variados de genes, associada a fatores ambientais, socioeconômicos e culturais15,17,27,28.
A análise da influência de dois polimorfismos do gene humano NRAMP1, realizada por Buu et al20 na suscetibilidade para hanseníase, foi realizada por meio de estudo de associação em seu formato mais simples, baseado na comparação de frequências alélicas de marcador genético específico entre indivíduos afetados e não afetados.
O objetivo principal deste estudo foi analisar a frequência alélica por meio de dois haplótipos do gene NRAMP1, sendo um com a inserção de quatro pares de bases (CAAA) com 163 pb e outro com deleção de quatro pares com 159 pb entre pacientes com hanseníase e grupo de não doentes.
MATERIAIS E MÉTODOS
Pacientes com hanseníase e um grupo de não doentes, representados pelos contatos consanguíneos (CCOS) e contatos não consanguíneos (CNCOS), foram selecionados em unidades de saúde dos Municípios de Tucuruí, Breu Branco, Curionópolis e Redenção, todos localizados a sudeste do Estado do Pará, após aprovação do projeto pelo Comitê de Ética em Pesquisa do Instituto Evandro Chagas (Protocolo CEP/IEC/CAAE 0013.0072-000-09 de 11/2011), e carta de anuência das Secretarias Municipais de Saúde dos municípios visitados. A classificação utilizada para especificar as formas clínicas da hanseníase foi a Classificação de Madri (1953)29, por meio da qual se dividiu o grupo de pacientes em multibacilares (MB), incluindo as formas dimorfa (HD) e virchoviana (HV); e paucibacilares (PB), incluindo as formas indeterminada (HI) e tuberculoide (HT).
No grupo de pacientes com hanseníase, 71 eram do sexo masculino (71/122 - 58,20%) com média de idade igual a 44,5 e média dos níveis de anticorpos IgM contra PGL-1 do M. leprae igual a 0,416; e 51 eram do sexo feminino (51/122 - 41,80%) com média de idade igual a 41,0 e média dos níveis de anticorpos anti-PGL-1 igual a 0,197. Desse grupo, 61 (61/122 - 41,80%) foram classificados como MB, sendo 5% da forma HV e 95,08% da forma HD. O total de pacientes PB foi 27 (27/122 - 22,13%), sendo dez do sexo masculino (10/27 - 37,00%) com média de idade igual a 37,22 e média de anticorpos anti-PGL-1 igual a 0,151; e 17 do sexo feminino (17/27 - 63,00%) com média de idade igual a 40,56 e média dos anticorpos anti-PGL-1 igual a 0,083. Destes, 15 (15/27 - 55,00%) foram classificados como forma HI e 12 (12/27 - 45,00%) como forma HT.
O grupo de não doentes apresentou 43 indivíduos do sexo masculino (43/110 - 39,0%) com média de idade igual a 30,99 e média de anticorpos anti-PGL-1 igual 0,095 e 67 eram do sexo feminino (67/110 - 61,00%) com média de idade 34,52 e média de anticorpos anti-PGL-1 igual a 0,126. Nesse grupo, 74 (74/110 - 67,00%) foram classificados como CCOS e 36 (36/110 - 33,00%) como CNCOS.
O grupo de não doentes foi subdividido em CCOS (representado por indivíduos que possuíam algum caso de hanseníase na família) e CNCOS (representado por indivíduos que não possuíam nenhum caso de hanseníase na família). O grupo de pacientes e contatos foi selecionado no período de fevereiro de 2008 a março de 2011. Todos os indivíduos selecionados passaram por consulta médica com os profissionais de saúde dos Municípios visitados e foram submetidos a exame clínico dermatoneurológico e teste de sensibilidade cutânea, para aqueles que apresentaram sinais e/ou sintomas compatíveis com hanseníase. Após assinatura do termo de consentimento livre e esclarecido, foi coletado material biológico - sangue total e soro - para realização dos testes imunológicos e de genotipagem para os haplótipos do gene NRAMP1.
Dados demográficos, como idade, sexo, procedência, classificação clínica, estado vacinal com BGC, foram obtidos por meio da aplicação de ficha epidemiológica específica para esse fim.
O DNA humano NRAMP1 foi extraído de amostras de sangue total, por meio do DNAzol BD (Invitrogen) conforme instruções do fabricante. Foi analisado um polimorfismo de inserção/deleção de 4 pb (pares de bases) de CAAA de um elemento Alu detectado na região 3' não traduzida (3'UTR) do NRAMP120.
Foram utilizados os primers NB3'F - 5'CTTTAACACAGTGTCTGGCAC3' e NB3'R2 -5'TCAAGCTCCAGTTTGGAGCCT3'20, amplificação de um fragmento do DNA que sobrepõe o comprimento do polimorfismo, originando um produto de 159 pb quando ocorre a deleção das bases, 163 pb quando ocorre a inserção, 159 pb e 163 pb quando o indivíduo for heterozigoto.
A reação de amplificação com volume final de 25 µL foi composta por 2 µL do DNA genômico, 2,5 µL de tampão da enzima; 0,70 µL de MgCl2, 0,5 µL de desoxinucleotídeo trifosfato (dNTPs), 1 µL de cada primer e 0,2 µL de taq DNA polimerase; e ocorreu com desnaturação inicial a 92° C/10 min, seguida de 40 ciclos de desnaturação a 92° C/1,5 min com anelamento a 61° C/1,5 min e extensão a 72° C/1,5 min. Após os ciclos foi feita uma extensão a 72° C/10 min. Os produtos de PCR foram visualizados por eletroforese em gel de poliacrilamida 8%.
SEQUENCIAMENTO DO GENE NRAMP1
O sequenciamento das amostras foi realizado empregando o método de terminação em cadeia descrito por Sanger et al30. A análise das sequências foi feita utilizando-se o software BioEdit, versão 7.03 para alinhamento e comparação, conforme a seguir: para confirmação do resultado das PCR foi feito o sequenciamento dos fragmentos amplificados de 159 pb e 163 pb. Foram escolhidas algumas amostras de indivíduos homozigotos para deleção e inserção e heterozigotos. A região sequenciada e os primers utilizados encontram-se na figura 1:

Os níveis de anticorpos IgM dirigidos contra PGL-1 do M. leprae foram medidos por meio de ensaio imunoenzimático padrão (ELISA) conforme descrito previamente5,6,7,8,9,10. De forma breve, placas NUNC de 96 poços foram sensibilizadas com antígeno semissintético trisacáride natural ligado a um radical fosfato inerente à albumina bovina (NT-P-BSA), lavadas com PBS Tween a 0,1% e bloqueadas com 100 u.L de solução de PBST a 1% de BSA, incubadas a 37° C por 1 h. Adicionou-se soro de pacientes previamente diluído com solução de PBST a 10% de soro normal de cabrito (NGS) em concentração de 1:300 e novamente incubadas a 37° C por 1 h. As placas foram lavadas quatro vezes com PBST e então se adicionou 50 µL do conjugado anti-IgM humana com a enzima peroxidase em concentração de 1:2000 e incubou-se novamente a 37° C por 1 h. As placas foram novamente lavadas quatro vezes com PBST, foi adicionado o substrato tetrametilbenzidine (TMB, Invitrogen) e as placas foram incubadas em temperatura ambiente e no escuro por aproximadamente 30 min. Dependendo do substrato, este tempo varia bastante. A reação foi parada com a adição de 50 µL da solução de ácido sulfúrico 2,5 N de H2SO4 no soro padrão, que apresentou absorbância de 450 nm. Amostras consideradas positivas apresentaram média de absorbância ≥ 0,2 e as negativas ≤ 0,2.
ANÁLISE ESTATÍSTICA
Inicialmente foi realizada análise de significância para todas as variáveis estudadas, por meio de regressão logística simples, com o software Epi InfoTM 2000. O nível de significância para cada variável individual associada aos desfechos foi realizado por meio de tabelas 2x2, calculada a razão de chances (odds ratio), risco relativo (RR) e o teste qui-quadrado, sendo o nível de significância estabelecido ≤ 0,05. Análises de concordância foram feitas em relação à idade, sexo, classificação clínica, positividade ao anti-PGL-1 e frequência dos haplótipos do gene NRAMP1 tipados.
RESULTADOS
Foi genotipado um total de 122 amostras de pacientes com hanseníase e 110 contatos de pacientes, procedentes de Tucuruí, Breu Branco, Curionópolis e Redenção, ao sudeste do Estado do Pará.
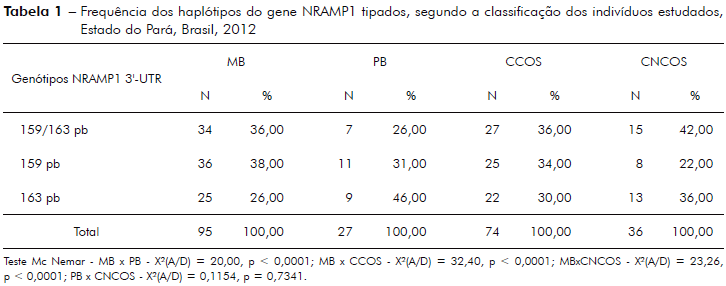
A tabela 1 demonstra a distribuição dos haplótipos do gene NRAMP1 tipados, os quais apresentam caráter heterozigoto (159/163 pb), homozigoto com a deleção de quatro pares de bases (159 pb), e homozigotos com a inserção de quatro pares de bases (163 pb) de acordo com a classificação dos indivíduos estudados.
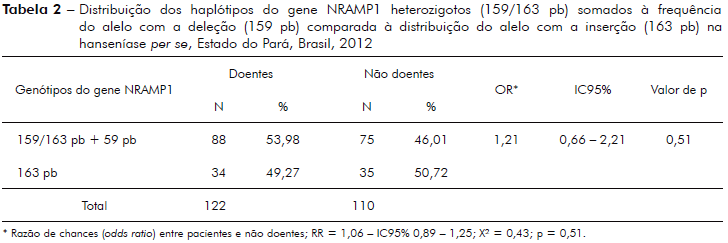
A tabela 2 mostra resultado da análise da frequência dos haplótipos do gene NRAMP1 com caráter heterozigoto (159/163 pb) somados à distribuição do alelo com a deleção de quatro pares de bases (159 pb) comparados à frequência do alelo homozigoto com a inserção de quatro pares de bases (163 pb) na hanseníase per se, comparando-se o grupo de pacientes com hanseníase com o grupo de não doentes, cálculo da odds ratio (OR), risco relativo (RR), teste qui-quadrado e o valor de p.
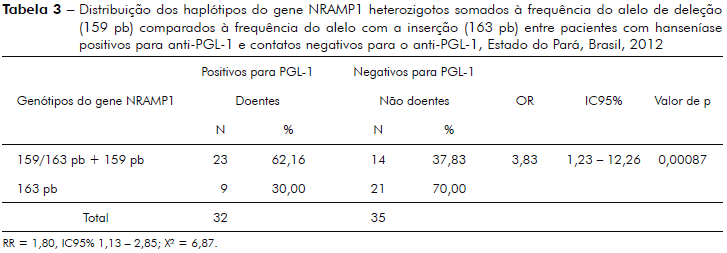
A tabela 3 demonstra a distribuição dos haplótipos do gene NRAMP1 heterozigotos, somados à frequência do alelo de deleção (159 pb), comparados à frequência do alelo com a inserção (163 pb) entre pacientes com hanseníase (MB e PB) positivos para o antígeno PGL-1 do M. leprae e o grupo de não doentes (CCOS + CNCOS) negativos para PGL-1.
A tabela 4 mostra análise da frequência do alelo de deleção (159 pb) comparado à frequência do alelo de inserção (163 pb) entre pacientes MB com PGL-1 positivo e CNCOS com PGL-1 negativo.

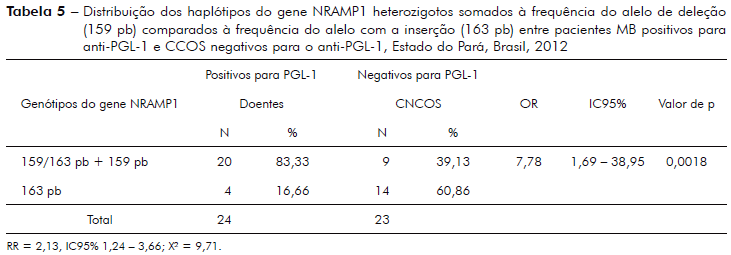
A tabela 5 mostra análise da frequência do haplótipo do gene NRAMP1 que possui o caráter heterozigoto (159/163 pb) somado à frequência do alelo com deleção (159 pb), comparada ao alelo com a inserção (163 pb) entre pacientes MB PGL-1 positivos e CCOS PGL-1 negativos.
DISCUSSÃO
Estudos de mapeamento genético demonstraram o locus do cromossomo 1 do camundongo (Ity, Lsh, Bcg), o qual controla capacidade do macrófago em restringir a replicação de parasitos intracelulares antigenicamente não relacionados e determina resistência natural (BCG-R, dominante) ou suscetibilidade (BCG-S, recessivo) de cepas de camundongo para infecção com diversos patógenos, incluindo várias espécies de micobactérias, como Salmonella Typhimurium e Leishmania donovani. No estudo de Malo et al19 foi feita clonagem posicional no mapeamento genético e físico para isolar o gene candidato para Bcg (Nramp1) o qual codifica uma proteína de transporte macrófago-específica14,16,17,19,32.
Estudo realizado no oeste africano mostrou claramente a associação do polimorfismo do gene NRAMP1(1729+55del4, uma deleção de TGTG na região 3' não traduzida, uma transversão de nucleotídeo único no íntron4 - 469+14G/C e um CA microssatélite na região 5' do gene) com as formas clínicas da hanseníase, no qual se observou número significativo de pacientes heterozigotos entre as formas MB; inclusive, o mesmo polimorfismo já havia demonstrado associação com a tuberculose pulmonar escarro positiva em estudo anterior33.
Outros estudos de associação genética não encontraram significância na suscetibilidade para hanseníase em famílias da Polinésia Francesa e Paquistão34,35, mas esse fato não invalida estudos que encontraram esta diferença e não significa que não há associação com hanseníase, pois a validação na estratificação e tamanho da população de pacientes e o fato das variações testadas não serem polimórficas nessas populações são fatos importantes a serem considerados36.
A distribuição das frequências dos haplótipos do gene NRAMP1 tipados revela diferenças significativas nos percentuais entre os grupos estudados. Análise da frequência dos alelos com 159/163 pb somado à frequência do alelo 159 pb entre pacientes MB, comparado ao grupo PB, apresentou valor do teste McNemar dos pares discordantes, X2(A/D) = 20,00 e valor de p < 0,0001, o mesmo acontecendo para os grupos MBxCCOS - X2(A/D) = 32,40 e valor de p < 0,0001; MBxCNCOS - X2(A/D) = 23,26 e valor de p < 0,0001. Somente para o grupo PBxCNCOS não houve significância estatística, X2(A/D) = 0,1154 e valor de p = 0,7341, fato que sugere que o perfil genético e imunológico dos indivíduos classificados como PB é realmente diferenciado do grupo MB, sendo que, neste último, o polimorfismo do gene humano NRAMP1 estudado pode estar contribuindo para suscetibilidade para hanseníase12,14,27,28,32,37,38.
Roger e colaboradores34 realizaram estudo de ligação para análise de suscetibilidade para hanseníase per se com nove polimorfismos do gene NRAMP1 e três marcadores polimórficos fisicamente ligados, marcadores de microssatélites polimórficos D2S104, D2S173 e D251471. Análises de ligação foram feitas usando pares de irmãos afetados e métodos de pontuação por LOD score, empregando diversos modos de herança, com penetrância completa e reduzida. Esse estudo não encontrou significância na associação do polimorfismo estudado com hanseníase em famílias da Polinésia Francesa.
Shaw e colaboradores35 utilizaram um painel de marcadores na região do cromossomo humano 2q33-Q37, conhecido por ser conservado e conter região do cromossomo murino 1. Conjuntos de marcadores utilizados foram CRYGP1, MAP2, FN1, TNP1, VIL1 e DES e, entre os pares adjacentes de região mais distal (2q35-Q57) definiram COL6A3, D2S55 e D2S3, e também não encontraram significância na associação deste polimorfismo com hanseníase.
Associação do genótipo do NRAMP1 tipado neste estudo, como a maioria dos estudos de associação genética com hanseníase, não encontrou significância estatística para hanseníase per se (p = 0,51, tabela 2)14,28,34,35, porém, quando esta análise foi ampliada com dados de sorologia positiva para anti-PGL-1, observou-se significância estatística para hanseníase per se (p = 0,0087) e risco relativo (RR) considerado de efeito moderado (RR = 1,80, IC95% - 1,13 - 2,85; X2 = 6.87, tabela 3). Achado semelhante foi também encontrado no estudo de Ferreira et al37, os quais utilizaram a reação de Mitsuda para determinar indivíduos com predominância de resposta imunológica do tipo Th1 (celular) e, desta forma, separando com maior precisão casos PB dos MB. Neste estudo foi utilizado o antígeno PGL-1 do M. leprae para discriminar indivíduos potencialmente MB dos PB, posto que o PGL-1 discrimina a imunidade predominantemente humoral.
Teixeira e colaboradores39, ao analisarem três polimorfismos do gene NRAMP1 (274C/T, DS43N e 1729+55del14) na determinação de reação tipo 1 (reversa) e reação tipo II (eritema nodoso hansênico), concluíram que é possível que a composição gênica dos indivíduos estudados mantenha uma relação mais estreita com a reação imunológica do que com a multiplicação bacteriana, representada pela baciloscopia, fato demonstrado neste estudo com o polimorfismo da região 3'UTR ins/Del CAAA, o qual foi claramente associado com o perfil de resposta imunológica predominante observada entre os pacientes com hanseníase por meio da positividade/negatividade ao teste anti-PGL-1.
Outras regiões do gene NRAMP1 foram estudadas e associadas ou não com as formas clínicas da hanseníase como, por exemplo, o estudo de Mochammad e colaboradores40, os quais observaram associação do polimorfismo do gene NRAMP1 (INTA) com hanseníase PB (p = 0,032), mas não com a hanseníase MB (p = 0,173), o que sugere a importância deste gene para os estudos de suscetibilidade para hanseníase.
Alguns estudos de ligação familiar e associação genética com hanseníase mostraram que algumas variações polimórficas do gene NRAMP1 testadas associaram-se às formas MB não somente pelo caráter heterozigoto, mas principalmente, pelo fato do indivíduo ser portador de pelo menos um alelo de deleção12,13'14,28,32,37,38. Neste estudo, observou-se forte associação entre a frequência do alelo com a deleção de CAAA de um elemento Alu da região 3' e hanseníase MB com PGL-1 positivo, comparada aos CNCOS com PGL-1 negativo e portadores do alelo com a inserção (p = 0,025' tabela 4). O risco de um indivíduo MB PGL-1 positivo fazer parte do grupo com a deleção foi 4,57 vezes maior comparado ao grupo de CNCOS, o que é considerado um risco forte do ponto de vista epidemiológico. Tal fato é relevante nas regiões endêmicas, pois isso representa mais um instrumento laboratorial para uso nas ações de vigilância epidemiológica da hanseníase.
Diferenças significantes também foram encontradas neste estudo quando se comparou pacientes MB heterozigotos ou portadores do alelo com a deleção (159 pb) PGL-1 positivo com o grupo de CCOS portadores do alelo com a inserção e PGL-1 negativos (p = 0,0018, tabela 5). E interessante observar que o grupo de CCOS representado por indivíduos que possuem casos de hanseníase na família apresenta semelhanças com o grupo de MB nas frequências dos alelos com a inserção e deleção - heterozigotos (159/163 pb) e com a deleção (159 pb, tabela 1), o que sugere que a transferência de genes com as variações polimórficas pode ser real nesta população e incita a necessidade de estudos mais profundos para apuração destes fatos13,28,32.
CONCLUSÃO
Este estudo de associação genética mostrou que o polimorfismo estudado, embora não tenha sido testado para avaliar suscetibilidade para hanseníase anteriormente, mostrou forte associação com hanseníase MB, comparada com a PB e com o grupo de não doentes. E um achado relevante para áreas endêmicas, pois suscita necessidade da realização de estudos de ligação e associação genética para melhor entendimento dos mecanismos de manutenção de altos índices da doença nessa região e possibilita proposições para vigilância epidemiológica da hanseníase.
AGRADECIMENTOS
Ao Instituto Evandro Chagas por possibilitar e apoiar a realização deste estudo.
REFERÊNCIAS
1 World Health Organization. Global leprosy: update on the 2012 situation. Wkly Epidemiol Rec. 2013 Aug;88(35):365-80. [Link]
2 Ignotti E, Paula RC. Situação epidemiológica da hanseníase no Brasil: análise de indicadores selecionados no período de 2001 a 2010. In: Ministério da Saúde (BR). Secretaria de Vigilância em Saúde. Saúde Brasil 2010: uma análise da situação de saúde e de evidências selecionadas de impacto de ações de vigilância em saúde. Brasília: Ministério da Saúde; 2011. p. 185-202. (Série G. Estatística e Informação em Saúde). [Link]
3 Geluk A, Klein M, Franken LMC, van Meijgaarden K, Wieles B, Pereira KC, et al. Postgenomic approach to identify novel Mycobacterium leprae antigens with potential to improve immunodiagnosis of infection. Infect Immun. 2005 Sep;73(9):5636-44. Doi: 10.1128/IAI.73.9.5636-5644.2005 [Link]
4 Spencer JS, Dockrell HM, Kim HJ, Marques MA, Williams DL, Martins MVSB, et al. Identification of specific proteins and peptides in Mycobacterium leprae suitable for the selective diagnosis of leprosy. J Immunol. 2005 Dec;175(12):7930-8. Doi: 10.4049/jimmunol.175.12.7930 [Link]
5 Bührer-Sékula S. A simple dipstick assay for the detection of antibodies to phenolic Glycolipid 1 of Mycobacterium leprae [thesis]. Departamento de Pesquisa Biomédica. Amsterdam (NL): Royal Tropical Institute; 1998. 123 p.
6 Bührer-Sékula S, Cunha MGS, Ferreira WA, Klatser PR. The use of whole blood in a dipstick assay for detection of antibodies to Mycobcaterium leprae: a field evaluation. FEMS Immunol Med Microbiol. 2006 Jul;21(3):197-201. Doi: 10.1111/j.1574-695X.1998.tb01166.x [Link]
7 Bührer-Sékula S, Sarno EN, Oskam L, Koop S, Wichers I, Nery JAC, et al. Use of ML Dipstick as a tool to classify leprosy patients. Int J Lepr Mycobact Dis. 2000 Dec;68(4):456-63. [Link]
8 Bührer-Sékula S, Cunha MG, Foss NT, Oskan L, Faber WR, Klatser PR. Dipstick assay to identify leprosy patients who have an increased risk of relapses. Trop Med Int Health. 2001 Apr;6(4):317-23. Doi: 10.1046/j.1365-3156.2001.00704.x [Link]
9 Bührer-Sékula S, Smits HL, Gussenhoven GC, van Leeuwen J, Amador S, Fujiwara T, et al. Simple and fast lateral Flow test for the classification of leprosy patients and identification of contacts with high risk of developing leprosy. J Clin Microbiol. 2003 May;41(5):1991-5. Doi: 10.1128/JCM.41.5.1991-1995.2003 [Link]
10 Bührer-Sékula S, van Beers S, Oskam L, Lecco R, Madeira ES, Dutra MAL, et al. A relação entre soroprevalência de anticorpos contra o glicolipídeo fenólico-I entre crianças em idade escolar e endemicidade da hanseníase no Brasil. Rev Soc Bras Med Trop. 2008;41 supl 2:81-8. Doi: 10.1590/S0037-86822008000700017 [Link]
11 Klatser PR, Cho SN, Brennan PJ. The contribution of serological tests to leprosy control. Int J Lepr Other Mycobact Dis. 1996 Dec;64(4 Suppl 1):S63-6.
12 Lang T, Prina E, Sibthorpe D, Blackwell JN. Nramp1 transfection transfer /fy/Lsfi/Bcg-related pleiotropic effects on macrophage activation: influence on antigen processing and presentation. Infect Immun. 1997 Feb;65(2):380-6. [Link]
13 Abel L, Sanchez FO, Oberti J, Thuc NV, Hoa LV, Lap VD, et al. Susceptibility to leprosy is linked to the human NRAMP1 gene. J Infect Dis. 1998 Jan;177(1):133-45. Doi: 10.1086/513830 [Link]
14 Meisner SJ, Mucklow S, Warner G, Sow SO, Lienhardt C, Hill AVS. Association of NRAMP1 polymorphism with leprosy per se in Weast Africans. Am J Trop Med Hyg. 2001 Dec;65(6):733-5. [Link]
15 Prevedelo FC, Mira MT. Hanseníase: uma doença genética? An Bras Dermatol. 2007 Sep-Oct;82(5):451-9. Doi: 10.1590/S0365-05962007000500009 [Link]
16 Vidal SM, Tremblay ML, Govoni G, Gauthier S, Sebastiani G, Malo D, et al. The Ity/Lsh/Bcg locus: natural resistance to infection with intracellular parasites is abrogated by disruption of the Nramp1 gene. J Exp Med. 1995 Sep;182(3):655-66. Doi: 10.1084/jem.182.3.655 [Link]
17 Cellier M, Govoni G, Vidal S, Kwan T, Groulx N, Liu J, et al. Human natural resistance-associated macrophage protein: cDNA cloning, chromosomal mapping, genomic organization, and tissue-specific expression. J Exp Med. 1994 Nov;180(5):1741-52. Doi: 10.1084/jem.180.5.1741 [Link]
18 Skamene E, Schurr E, Gross P. Infecction genomics: NRAMP1 as a major determinant of natural resistance to intracellular infections. Annu Rev Med. 49:275-87. Doi: 10.1146/annurev.med.49.1.275 [Link]
19 Malo D, Vogan K, Vidal S, HU J, Cellier M, Schurr E, et al. Haplotype mapping and sequence analysis of the mouse Nramp gene predict susceptibility to infection with intracellular parasites. Genomics. 1994 Sep;23(1):51-61. Doi: 10.1006/geno.1994.1458 [Link]
20 Buu NT, Cellier M, Gros P, Schurr E. Identification of a highly polymorphic length variant in the 3'UTR of NRAMP1 . Immunogenetics. 1995 Sep;42(5):428-9. Doi: 10.1007/BF00179408 [Link]
21 Bueno R. Estudo do gene NRAMP1 canino em macrófagos infectados com Leishmania (Leishmania) chagasi [tese]. Belo Horizonte (MG): Universidade Federal de Minas Gerais, Escola de Veterinária; 2006. 105 p. [Link]
22 Gruenheide S, Cellier M, Vidal S, Gros P. Identification and characterization of a second mouse Nramp gene. Genome. 1995 Jan;25(2):514-25. Doi: 10.1016/0888-7543(95)80053-O [Link]
23 Canonne-Hergaux F, Gruenheid S, Govoni G, Gros P. The Nramp1 protein and its role in resistance to infection and macrophage function. Proc Assoc Am Physicians. 1999 Jul-Aug;111(4):283-9. Doi: 10.1046/j.1525-1381.1999.99236.x [Link]
24 Jabado BN, Jankowski A, Dougaparsad S, Picard V, Grinstein S, Gros P. Natural resistance-associated macrophage protein 1 (Nramp1) functions as a pH-dependent the phagosomal membrane. J Exp Med. 2000 Nov;192(9):1237-47. Doi: 10.1084/jem.192.9.1237 [Link]
25 Forbes JR, Gros P. Divalentmetal transport by NRAMP proteins at the interface of host-pathogen interactions. Rev Trends Microbiol. 2001 Aug;9(8):397-403. Doi: 10.1016/S0966-842X(01)02098-4 [Link]
26 Wyllie S, Seu P, Goss JA. The natural resistance-associated macrophage protein 1 Slc11a1 (formerly Nramp1) and iron metabolism in macrophages. Microbes Infect. 2002 Mar;4(3):351-9. Doi: 10.1016/S1286-4579(02)01548-4 [Link]
27 Abel L, Dessein A. Genetic epidemiology of infectious diseases in humans: design of population-based studies. Emerg Infect Dis. 1998 Oct-Dec;4(4):593-603. Doi: 10.3201/eid0404.980409 [Link]
28 Bakker M. Epidemiology and prevention of leprosy: a cohort study in Indonesia [thesis]. Amsterdam (NL): University of Amsterdam, Academic Medical Center; 2005.
29 Souza CS. Hanseníase: formas clínicas e diagnóstico diferencial. Medicina. 1997 jul-set;30(3):325-34. Doi: 10.11606/issn.2176-7262.v30i3p325-334 [Link]
30 Sanger F, Nicklen S, Coulson AR. DNA sequencing with chain terminating inhibitors. Proc Nat Acad Sci USA. 1977;74(12):5463-7. [Link]
31 Hall TA. BioEdit: a user-friendly biological sequence aligment editor and analysis program for Windows 95/98/NT. Nucleic Acids Symposium Ser. 1999;(41):95-8. [Link]
32 Blackwell JM. Genetics of host resistance and susceptibility to intramacrophage pathogens: a study of multicase families of tuberculosis, leprosy and leishmaniasis in north-eastern Brazil. Int J Parasitol. 1998 Jan;28(1):21-8. Doi: 10.1016/S0020-7519(97)00175-6 [Link]
33 Bellamy RJ. Susceptibility to infectious diseases: the importance of host genetics. Cambridge: Cambridge University Press; 2003. (Advances in molecular and cellular microbiology; no. 4). Doi: 10.1017/CBO9780511546235 [Link]
34 Roger M, Levee G, Chanteau S, Gicquel B, Schurr E. No evidence for linkage between leprosy susceptibility and human natural resistance-associated macrophage protein 1 (NRAMP1 ) gene in French Polynesia. Int J Lepr Other Mycobact. 1997 Jun;65(2):197-202. [Link]
35 Shaw MA, Atkinson S, Dockrell H, Hussain R, Lins-Lainson Z, Shaw J, et al. An RFLP map for 2q33-q37 from multicase mycobacterial and leishmanial disease families: no evidence for an Lsh/Ity/Bcg gene homologue influencing susceptibility to leprosy. Ann Hum Genet. 1993 Oct;57(4):251-7. Doi: 10.1111/j.1469-1809.1993.tb00899.x [Link]
36 Cellier M, Gros P, editors. The NRAMP1 family. Georgetown: Eurekah; 2004. (Molecular biology intelligence unit).
37 Ferreira FR, Goulart LR, Silva HD, Goulart IM. Susceptibility to leprosy may be conditioned by an interaction between the NRAMP1 promoter polymorphisms and the lepromin response. Int J Lepr Other Mycobact Dis. 2004 Dec;72(4):457-67. [Link]
38 Remus N, Alcais A, Abel L. Human genetics of common mycobacterial infections. Immuno Res. 2003 Oct;28(2):109-29. Doi: 10.1385/IR:28:2:109 [Link]
39 Teixeira MAG, Silva NL, Hatagima A, Magalhães V. Polimorfismos do gene Nramp1 em indivíduos com reações hansênicas, atendidos em dois centros no Recife, Nordeste do Brasil. Rev Soc Bras Med Trop. 2010 mai-jun;43(3):281-6. Doi: 10.1590/S0037-86822010000300014 [Link]
40 Hatta M, Ratnawati, Tanaka M, Ito J, Shirakawa T, Kawabata M. NRAMP1/SLC11A1 Gene polymorphisms and host susceptibility to Mycobacterium tuberculosis and M. leprae in South Sulawesi, Indonesia. Southeast Asian J Trop Med Public Health. 2010 Mar;41(2);386-94. [Link]
 Correspondência / Correspondence / Correspondencia:
Correspondência / Correspondence / Correspondencia:
Maria do Perpétuo Socorro Amador Silvestre
Instituto Evandro Chagas
Rodovia BR 316, km 7, s/n. Bairro: Levilândia
CEP: 67030-000 Ananindeua-Pará-Brasil
Tel.: + 55 (91) 3214-2121
E-mail: socorroamador@iec.pa.gov.br
Recebido em / Received / Recibido en: 20/4/2012
Aceito em / Accepted / Aceito en: 18/9/2012