INTRODUÇÃO
Cepas de Escherichia coli colonizam o trato gastrointestinal de seres humanos e animais poucas horas após o nascimento e constituem o principal membro anaeróbio facultativo da microbiota intestinal desses organismos1. Essa espécie, pertencente à família Enterobacteriaceae, raramente causa doença em seus hospedeiros. Apesar de seu papel como comensal, algumas cepas de E. coli são capazes de causar uma grande variedade de doenças, devido à aquisição de fatores de virulência por transferência horizontal1. Dentre essas doenças, a diarreia é uma das principais, sendo as E. coli diarreiogênicas (ECD) responsáveis por inúmeros surtos de diarreia registrados no mundo2.
Atualmente são conhecidos seis patotipos de ECD: E. coli enteroinvasora (EIEC); E. coli enterotoxigênica (ETEC); E. coli enteropatogênica (EPEC), subdividida em EPEC típica (tEPEC) e EPEC atípica (aEPEC); E. coli produtora de toxina de Shiga (STEC), e seu subgrupo E. coli enterohemorrágica (EHEC); E. coli enteroagregativa (EAEC); e E. coli que adere difusamente (DAEC)1. Dentre esses patotipos, cepas EPEC destacam-se como um importante agente diarreiogênico presente em países em desenvolvimento2.
Recentemente, uma nova classificação foi proposta por Clements et al.3, na qual as E. coli são divididas em oito patotipos: os seis anteriormente descritos, acrescidos de E. coli aderente invasiva (AIEC), associada com a doença de Crohn; e E. coli enteroagregativa produtora da toxina Shiga (STEAEC), responsável por um grande surto diarreico em 2011 na Europa.
As EPEC podem ser definidas pela presença do gene eae, o qual codifica uma proteína denominada intimina, que é requerida para a adesão à mucosa intestinal, produzindo uma lesão do tipo attaching and effacing (A/E). Essa lesão é iniciada pela adesão da bactéria nos enterócitos, levando à perda das microvilosidades intestinais. A intimina é altamente variável entre os diferentes sorotipos de EPEC, e algumas variantes antigênicas distintas têm sido identificadas como α, β, γ, δ, ε, ι, κ, η, ζ, entre outras4. As EPEC podem ser classificadas em típicas e atípicas. Linhagens que possuem o plasmídeo EAF (EPEC adherence factor) são chamadas de tEPEC e as que não possuem são chamadas de aEPEC. O gene bfp contido no plasmídeo EAF codifica um fator de adesão denominado bundle-forming pili (BFP), sendo que o plasmídeo EAF também possui genes necessários para a expressão da intimina2.
Cepas aEPEC pertencem a um grupo diarreiogênico que aumentou muito sua frequência no Brasil nos últimos anos. Em algumas regiões do país, essas bactérias parecem ter substituído as tEPEC como principal agente diarreiogênico. Isso indica que, no Brasil, assim como ocorreu em países desenvolvidos, está havendo mudança no perfil epidemiológico das EPEC de típicas para atípicas3,5.
Cepas tEPEC raramente são isoladas de animais, sendo o homem seu principal reservatório natural6. Em contraste, cepas aEPEC são isoladas de humanos e animais, com ou sem diarreia, em frequências equivalentes, não permitindo a definição de um reservatório para essas bactérias7,8.
Diversos estudos associam animais de estimação, de criação e alguns silvestres como possíveis reservatórios e fonte de infecção de aEPEC para humanos9. Apesar disso, nenhum comparou, de forma ampla, amostras isoladas de humanos e animais por técnicas de biologia molecular, como o sequenciamento, para comprovar tais hipóteses.
Origens clonais comuns entre cepas bacterianas isoladas de animais e humanos podem identificar fontes de infecção e reservatórios animais de diferentes patógenos. Amostras de ECD, para os animais e para o homem, na maioria das vezes, possuem fatores de virulência semelhantes, codificados por genes de origem cromossômica ou plasmidial, cujas similaridades ou diferenças podem explicar aspectos filogenéticos e patogenicidade de certos grupos de E. coli para humanos.
A análise epidemiológica molecular de cepas bacterianas provenientes de diferentes origens é uma avaliação das relações de parentesco existentes entre elas. A principal questão dessas análises é saber se diferentes isolados de um determinado patógeno representam a disseminação de um organismo comum ou clone. Nesse sentido, a epidemiologia tem buscado desenvolver técnicas que proporcionem uma melhor avaliação das relações existentes entre diferentes isolados bacterianos.
O conceito básico das técnicas de tipagem molecular propõe que amostras bacterianas, epidemiologicamente relacionadas, possuem um precursor comum; dessa forma, estudos utilizando essas técnicas permitem uma melhor compreensão sobre os mecanismos evolutivos, sobre o surgimento de cepas virulentas e reservatórios de cepas bacterianas10. Diante do exposto, devido aos possíveis riscos acarretados à saúde pública por linhagens de EPEC, são necessários estudos que avaliem a distribuição e frequência dessas cepas nos animais, possibilitando a comparação com cepas de EPEC isoladas de humanos.
MATERIAIS E MÉTODOS
OBTENÇÃO DAS AMOSTRAS
Foram obtidas amostras fecais ou porções dos intestinos delgado e grosso, linfáticos e mesentéricos e fragmentos de fígado e baço de 630 animais silvestres (roedores, aves e marsupiais) de ambos os sexos, capturados em três municípios do estado do Pará (Marabá, Parauapebas e Canaã dos Carajás), de março de 2008 a dezembro de 2009 (Figura 1). Os espécimes fecais coletados foram acondicionados em meio de transporte Cary Blair e, juntamente com o pool de órgãos mantidos sob refrigeração, encaminhados diretamente ao Laboratório de Enteroinfecções Bacterianas do Instituto Evandro Chagas (IEC), onde foram processadas.
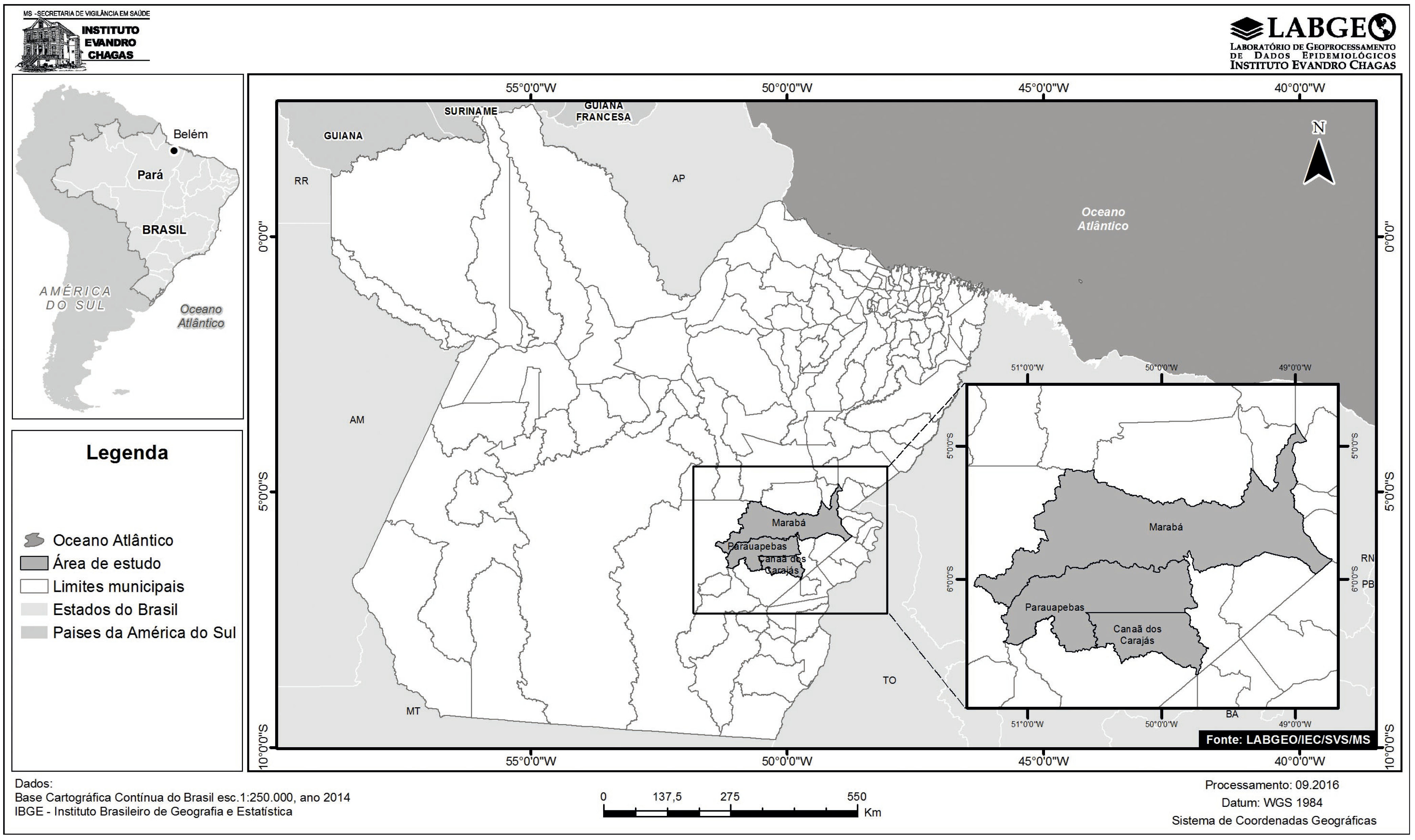
Figura 1 - Localização dos municípios de Parauapebas, Marabá e Canaã dos Carajás, estado do Pará, Brasil
Os experimentos com animais foram aprovados pelo Instituto Brasileiro do Meio Ambiente e dos Recursos Naturais Renováveis, processo nº 02018001038/08-11.
ISOLAMENTO E IDENTIFICAÇÃO BIOQUÍMICA
Para isolamento de E. coli, o material fecal em suspensão ou do pool de órgãos foi semeado em meios Ágar MacConkey - MC (Difco, EUA) e incubado à temperatura de 35-37 ºC, por 18-24 h. Após esse período, as colônias suspeitas de E. coli foram submetidas aos meios de triagem Triple Sugar Iron Agar - TSI (Difco, EUA). A caracterização bioquímica dos isolados bacterianos foi realizada utilizando o kit VITEK® 2 (bioMérieux, Brasil).
IDENTIFICAÇÃO DAS CATEGORIAS DE E. coli DIARREIOGÊNICAS
Três a cinco isolados de E. coli selecionados de cada animal foram mantidos em caldo Luria com glicerol a -70 ºC para pesquisa de EPEC, EIEC, STEC, ETEC e EAEC, utilizando a reação em cadeia da polimerase (PCR) Multiplex11,12. Os controles positivos (EPEC E2348/69, EHEC EDL 931, ETEC H10407, EIEC EC 299/07, EAEC 042) e negativo (K12 DH5α) foram cedidos gentilmente pela Dra. Tânia Vaz, do Instituto Adolfo Lutz, São Paulo.
PESQUISA DE GENES DE VIRULÊNCIA DE E. coli
As cepas foram cultivadas em Ágar Nutriente (Difco, EUA) para serem utilizadas no procedimento de extração de DNA. O material extraído foi armazenado a -20 ºC por um tempo máximo de 20 dias para ser utilizado nas reações da PCR Multiplex. Para essa, foram utilizados iniciadores descritos por Aranda et al.13 e Toma et al.14 (Tabela 1). As cepas identificadas como aEPEC tiveram o seu gene eae sequenciado, utilizando a plataforma ABI 3130 (Applied Biosystems, EUA) com o kit ABI PRISM™ Dye Terminator Cycle Sequencing (Applied Biosystems, EUA). As sequências produzidas foram editadas no programa Sample Manager, acoplado ao ABI 3130 DNA Sequencer. Para análise das sequências, foi utilizado os programas BioEdit v7.1.315, Mega v5.0516 e DnaSP v517.
Tabela 1 - Iniciadores utilizados na PCR Multiplex e seus respectivos produtos de amplificação
| Iniciadores | Sequência (5'-3') | Gene-alvo | Produtos de amplificação (pb) | Referência |
|---|---|---|---|---|
| eae-1 eae-2 | CTGAACGGCGATTACGCGAA CGAGACGATACGATCCAG | eae | 917 | Aranda et al.13 |
| BFP-1 BFP-2 | AATGGTGCTTGCGCTTGCTGC GCCGCTTTATCCAACCTGGTA | bfpA | 326 | Aranda et al.13 |
| aggRks-1 aggRksa-2 | GTATACACAAAAGAAGGAAGC ACAGAATCGTCAGCATCAGC | aggR | 254 | Toma et al.14 |
| LT-f LT-r | GGCGACAGATTATACCGTGC CGGTCTCTATATTCCCTGTT | elt | 450 | Aranda et al.13 |
| ST-f ST-r | ATTTTTMTTTCTGATTTRTCTT CACCCGGTACARGCAGGATT | est | 190 | Aranda et al.13 |
| IpaH-1 IpaH-2 | GTTCCTTGACCGCCTTTCCGATACCGTC GCCGGTCAGCCACCCTCTGAGAGTAC | ipaH | 600 | Aranda et al.13 |
| VTcom-u VTcom-d | GAGCGAAATAATTTATATGTG TGATGATGGCAATTCAGTAT | stx1/stx2 | 518 | Toma et al.14 |
EXTRAÇÃO DO DNA E PCR MULTIPLEX
O DNA dos isolados caracterizados fenotipicamente como E. coli e das cepas de referência dos controles positivos e negativo foi extraído pelo método de fervura e congelamento, seguindo as recomendações de Starnbach et al.18 e Baloda et al.19.
A PCR foi realizada a partir de 2 μL de cada DNA extraído e 23 μL da solução mix, contendo entre 0,5 e 1,5 μL, de acordo com cada iniciador (Invitrogen, Brasil); 10 mM de dNTP mix dATP, dCTP, dGTP, dTTP (Invitrogen, Brasil); 0,5 U de Platinum Taq DNA Polymerase; tampão Taq 1X; 50 mM de MgCl2 (Invitrogen, Brasil) e água estéril ultra pura para um volume final de 25 μL. As preparações da PCR Multiplex foram colocadas no termociclador automático de gradiente modelo VeritiTM 96-Well Thermal Cycler (Applied Biosystems, EUA) e submetidas a ciclos específicos de amplificação que consistiu de um passo de 2 min a 50 ºC (hot-start), um passo de 5 min a 95 ºC (desnaturação inicial), seguido por 40 ciclos de 1 s a 95 ºC, 50 ºC e 72 ºC, e um passo final de extensão de 7 min a 72 ºC.
Os amplicons foram analisados por eletroforese em gel de agarose a 2%, corado com brometo de etídeo (10 mg/mL) em tampão TBE (Tris base 0,89M, ácido bórico 0,45 M, EDTA 1 mM, pH 8,4) e visualizados sob luz UV com auxílio de um transiluminador (Vilber Lourmat, França). Como marcador de tamanho molecular, foi usado o Ladder de 1 Kb plus (Invitrogen, Brasil). Posteriormente, o gel foi fotografado por um sistema de fotodocumentação, Bioimaging Systems (UPV, EUA).
SEQUENCIAMENTO DO GENE eae DA aEPEC
Os fragmentos dos genes eae obtidos de oito isolados foram purificados com o kit comercial Kit BigDye® X Terminator (Applied Biosystems, EUA) e sequenciados para a confirmação, usando o primer forward específico para o gene eae. Para o sequenciamento, foi preparado um mix contendo 200-300 ng do DNA suspeito e o primer específico na concentração de 1,5 pmol/mL, e, posteriormente, alinhadas e comparadas com as sequências para o gene eae aEPEC: isoladas de pacientes diarreicos, (KT591325.1 ζ e KT591333.1 β2), isolada de animal (KT591282.1 ε1) e isolada de humano e animal (KT591233.1 β1).
RESULTADOS
Das 630 culturas analisadas, fezes e pool de órgãos, foram isoladas E. coli em 263 (41,7%) amostras (Tabela 2). Após a PCR, foi observada amplificação para o gene eae em 3,04% (8/263), distribuídos em roedores com 2,66% (7/263) e marsupiais com 0,4% (1/263) (Tabela 3; Figura 2).
Tabela 2 - Número de E. coli isoladas de animais silvestres capturados em três municípios do estado do Pará (Marabá, Parauapebas e Canaã dos Carajás), Brasil
| Classes | E. coli | não DEC* | |
|---|---|---|---|
| Nº de isolados | aEPEC | ||
| Aves | 34 | - | 34 |
| Marsupiais | 22 | 1 | 21 |
| Quelônios | - | - | - |
| Roedores | 195 | 7 | 188 |
| Lagomorfos | - | - | - |
| Répteis | 12 | - | 12 |
| Total | 263 | 8 | 255 |
* E. coli não diarreiogênica; Sinal convencional utilizado: - Dado numérico igual a zero não resultante de arredondamento.
Tabela 3 - Cepas aEPEC isoladas de animais silvestres (roedores e marsupiais) capturados em três municípios do estado do Pará (Marabá, Parauapebas e Canaã dos Carajás), Brasil
| Registro IEC | Animal | Nome científico | Município |
|---|---|---|---|
| 445 | Roedor | Proechimys guyannensis | Parauapebas |
| 447 | Roedor | Proechimys guyannensis | Parauapebas |
| 574 | Marsupial | Monodelphis emiliae | Parauapebas |
| 611 | Roedor | Oligoryzomys sp. | Parauapebas |
| 630 | Roedor | Oryzomys capito | Canaã dos Carajás |
| 812 | Roedor | Oxymycterus sp. | Parauapebas |
| 813 | Roedor | Oxymycterus sp. | Parauapebas |
| 856 | Roedor | Oxymycterus sp. | Parauapebas |
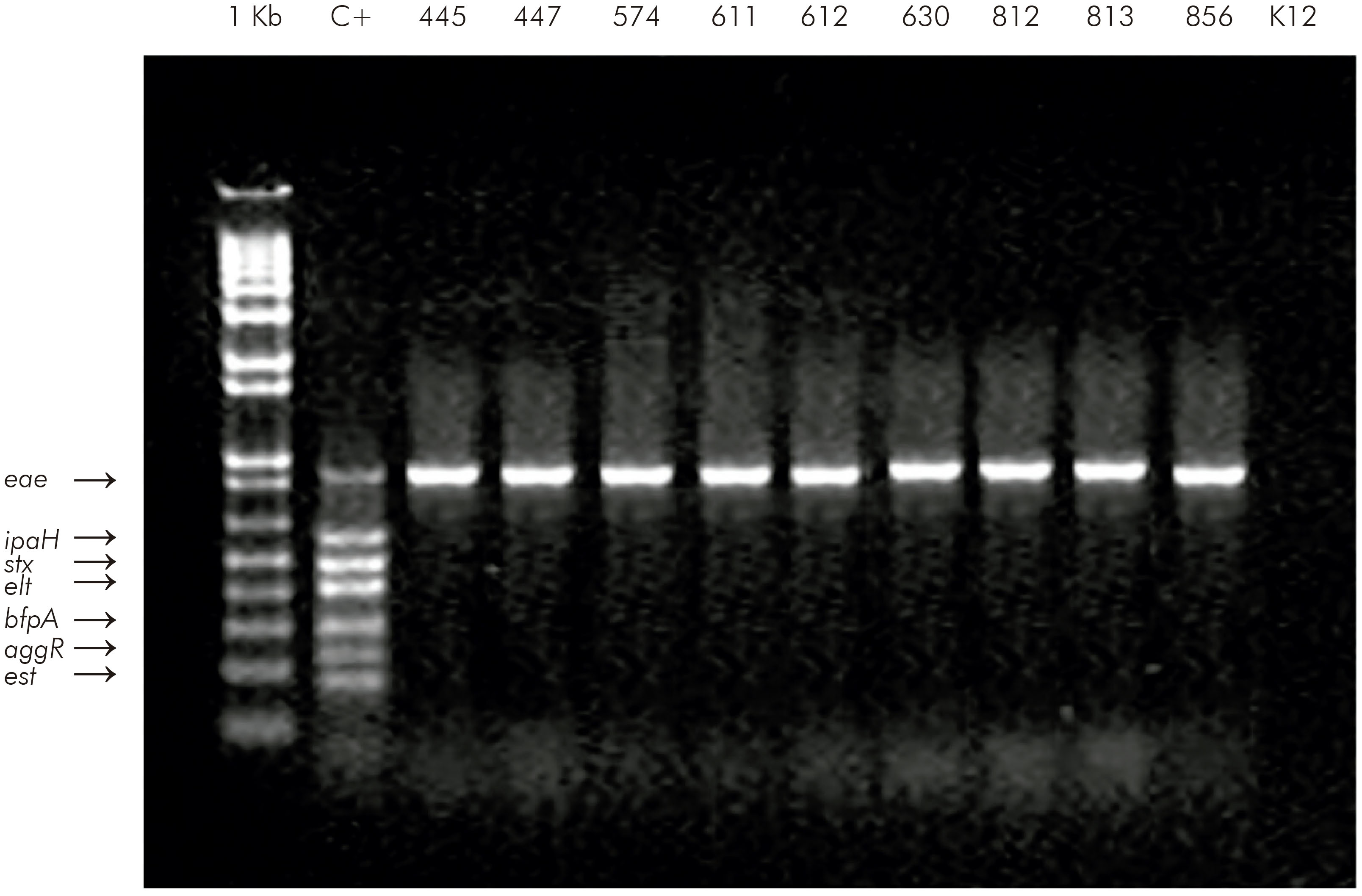
Figura 2 - PCR Multiplex - 1: Ladder 1 Kb; 2: Controle positivo (cinco categorias de DEC); 3, 4, 5, 6, 7, 8, 9, 10, 11: aEPEC; e 12: Controle negativo (E. coli K12 DH5).
Após PCR e sequenciamento do gene eae, as amostras foram alinhadas pelo programa BioEdit v7.1.315 com as mutações descritas na tabela 4. Foram observadas taxas de transição/transversão (R) igual a 7.47, segundo modelo de Kimura20 2-parâmetros. A análise do teste de Tajima's Neutrality demonstrou diversidade nucleotídica (π) de 0.014298 para um número de segregações por sítio igual a 24 nos 917pb do gene eae das oito amostras analisadas. Após análises das sequências do gene eae das aEPEC dos oito isolados de origem animal, foi observada a presença de quatro variantes distintas para a intimina, que foram β1 (amostras 574, 812 e 813), β2 (amostra 630), ζ (amostras 445 e 447) e ε (amostras 611 e 856).
Tabela 4 - Alinhamento das sequências obtidas com a utilização do BioEdit v7.1.3 com seus respectivos pontos de mutações
| Pontos polimórficos | 717 | 741 | 783 | 786 | 810 | 909 | 978 | 987 | 990 | 999 | 1012 | 1014 | 1023 | 1035 | 1040 | 1041 | 1050 | 1053 | 1089 | 1096 | 1113 | 1137 | 1257 | 1293 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| KT591325.1_zeta_intimina | A | A | G | T | T | T | A | C | C | T | C | G | C | T | G | G | T | G | T | C | T | T | T | A |
| EAE_445 | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| EAE_447 | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| KT591333.1_beta2_intimina | . | . | . | . | C | . | . | . | . | . | . | . | . | C | . | . | . | . | . | . | . | . | . | . |
| EAE_630 | . | . | . | . | C | . | . | . | . | . | . | . | . | C | . | . | . | . | . | . | . | . | . | . |
| KT591282.1_epsilon1_intimina | C | G | T | C | . | C | G | T | T | C | T | A | T | C | A | A | C | A | C | T | C | C | C | G |
| EAE_611 | C | G | T | C | . | C | G | T | T | C | T | A | T | C | A | A | C | A | C | T | C | C | C | G |
| EAE_856 | C | G | T | C | . | C | G | T | T | C | T | A | T | C | A | A | C | A | C | T | C | C | C | G |
| KT591233.1_beta1_intimina | C | T | T | . | C | C | G | T | T | C | T | A | T | C | A | A | C | A | C | T | C | C | C | G |
| EAE_574 | C | T | T | . | C | C | G | T | T | C | T | A | T | C | A | A | C | A | C | T | C | C | C | . |
| EAE_812 | C | T | T | . | C | C | G | T | T | C | T | A | T | C | A | A | C | A | C | T | C | C | C | G |
| EAE_813 | C | T | T | . | C | C | G | T | T | C | T | A | T | C | A | A | C | A | C | T | C | C | C | G |
Os nucleotídeos idênticos são indicados por pontos; KT591325.1, KT591333.1, KT591282.1, KT591233.1: Sequências de referências para as classes de intiminas analisadas.
Após análise filogenética pelo método de agrupamento de pares não ponderados com base na média aritmética (unweighted pair group method with arithmetic mean - UPGMA), a árvore consenso apresentou a formação de dois grupos, compostos de dois ramos distintos cada, agrupando, no primeiro grupo, KT591282.1, ε1 intimina (611 e 856) com KT591233.1, β1 intimina (574, 812 e 813); e, no segundo grupo, KT591325.1, ζ intimina (445 e 447) com KT591333.1, β2 intimina (630) (Figura 3).
DISCUSSÃO
Os marcadores de virulência analisados revelaram que as aEPEC isoladas possuem características genéticas semelhantes àquelas encontradas em humanos com as sequências do gene eae conservadas nas amostras 445, 447 e 630, provenientes de isolados de roedores. Estudos anteriores demonstraram a ocorrência de animais como reservatório de cepas consideradas como potenciais agentes de doenças que podem ser transmitidas entre seres humanos e animais21,22,23.
Estudos com aEPEC têm focado principalmente na comparação entre cepas isoladas de casos diarreicos e de pacientes saudáveis24,25,26. Mais recentemente, a identificação da presença de tipos de EPEC potencialmente patogênicos para humanos, isolados de animais, pode indicar que a transmissão desses microrganismos entre animais e seres humanos ocorre e tem um impacto sobre a saúde pública6; porém pouco se tem avaliado acerca da ocorrência desses patógenos em animais silvestres. Dessa forma, o presente estudo buscou a identificação das subclasses de intiminas em animais silvestres e observou a ocorrência das subclasses ζ, β1, β2 e ε, como aquelas que estão neles circulando. Como essas subclasses já foram descritas em seres humanos e animais de criação6,24,25,26, os dados podem apontar para uma possível contaminação da fauna silvestre por cepas provenientes da contaminação ambiental ou que esses espécimes estão servindo de reservatórios naturais para esse patógeno.
Dentre os animais estudados, os roedores apresentaram maior frequência de aEPEC, com amostras apresentando sequências altamente conservadas para o gene eae, quando comparadas a isolados humanos6. Apenas um marsupial foi identificado como portador da aEPEC, porém a sequência isolada apresentou 100% de similaridade com aquelas observadas em roedores. A grande diversidade genômica da E. coli lhe confere uma notável plasticidade ecológica; graças a ela, esses microrganismos adaptam-se rapidamente a diferentes ambientes, podendo, dessa forma, passar de organismo de vida livre a comensal do trato intestinal dos animais de sangue quente e, ainda, a patógenos que infectam humanos e animais6,9,25,27,28,29,30.
Xu et al.6, ao analisarem as subclasses de intiminas em humanos, animais e alimentos, identificaram as intiminas λ, β2, μ, ι2, ξ, ζ, α2 e π em pacientes diarreicos. A subclasse γ1 foi identificada concomitantemente em pacientes diarreicos e em suínos; a intimina κ, identificada tanto em alimento (carne crua) como em pacientes diarreicos; e as classes ε2, η2 e ι1, tanto em pacientes diarreicos como em assintomáticos, demonstrando a grande variabilidade e distribuição das aEPEC entre as amostras analisadas. Anteriormente, Moura et al.31 descreveram o subtipo β1 como o mais frequente em isolados de animais e humanos; nesse mesmo sentido, Aidar-Ugrinovich et al.22 já haviam descrito que, no Brasil, o subtipo β1 se apresentava como o mais frequente em EPEC isoladas de bovinos; semelhante ao observado por Cortés et al.32, que identificaram o subtipo β1 como o mais prevalente nas cepas isoladas de ovinos, seguido do subtipo γ2. No presente estudo, os subtipos mais frequentes foram β1 (amostras 574, 812 e 813), β2 (amostra 630), ζ (amostras 445 e 447) e ε (amostras 611 e 856), presentes em oito aEPEC das 263 E. coli identificadas. Porém, como não foram isoladas tEPEC, e 255 amostras foram caracterizadas como não diarreicas, sugere-se que a variabilidade existente pode ser maior, como observado na literatura, reforçando a necessidade de mais estudos que esclareçam a variabilidade genética existente entre as EPEC isoladas de humanos e animais na Região Amazônica.
CONCLUSÃO
Os dados demonstraram que as aEPEC isoladas dos animais silvestres possuem características genéticas semelhantes e mostraram proximidade com o gene eae de aEPEC encontradas em humanos e em animais de criação, possibilitando inferir que os animais avaliados servem como possíveis reservatórios de cepas de origem humana, bem como potenciais transmissores de cepas animais com caráter zoonótico.










 texto em
texto em