Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Epidemiologia e Serviços de Saúde
versão impressa ISSN 1679-4974versão On-line ISSN 2237-9622
Epidemiol. Serv. Saúde v.22 n.2 Brasília jun. 2013
http://dx.doi.org/10.5123/S1679-49742013000200008
ARTIGO ORIGINAL
Diversidade molecular de rotavírus do grupo A na cidade de Manaus, Estado do Amazonas, Brasil, 2004 a 2006*
Group A rotavirus molecular diversity in the city of Manaus, Amazonas state, Brazil 2004-2006
Giane Zupellari dos Santos MeloI; Cristóvão Alves da CostaII; Ilia Gilmara Carvalho dos SantosIII
IUniversidade do Estado do Amazonas
IIInstituto Nacional de Pesquisas da Amazônia
IIIUniversidade Federal do Amazonas
RESUMO
OBJETIVO: descrever a ocorrência de genótipos de rotavírus A em crianças na idade de 1 a 36 meses no município de Manaus, Estado do Amazonas, Brasil.
MÉTODOS: estudo descritivo, com resultados de análises laboratoriais de fezes diarréicas de crianças de 1 a 36 meses, atendidas em prontos-socorros infantis de Manaus-AM no período de junho/2004 a dezembro/2006; foram empregados os métodos de eletroforese de ácido ribonucleico em gel de poliacrilamida, transcriptase reversa e reação em cadeia da polimerase.
RESULTADOS: constatou-se 25% de positividade para rotavírus A - 30,7% e 44,7% para genótipos G e P, respectivamente -, com predominância dos genótipos G2 (17%) e P[8] (66,7%); das amostras positivas para os genótipos G e P, 38,3% e 11,7%, respectivamente, não apresentaram características para qualquer dos genótipos G e P pesquisados.
CONCLUSÃO: a elevada proporção de ausência de características dos genótipos G e P pesquisados sugere a circulação de genótipos diferentes desses.
Palavras-chave: Gastroenterite; Infecções por Rotavírus; Rotavirus; Epidemiologia Descritiva.
ABSTRACT
OBJECTIVE: verify rotavirus A genotype occurrence in children aged 1 to 36 months in Manaus-AM.
METHODS: descriptive and retrospective study conducted from June/2004 to December/2006, through laboratory analysis of diarrheal stools of children aged 1 to 36 months cared for at a Child Emergency service in Manaus/Amazonas.
RESULTS: 25% rotavirus A positivity was found using Polyacrylamide Gel Electrophoresis of Ribonucleic Acid. Reverse Transcriptase followed by Polymerase Chain Reaction were applied to positive samples and revealed 30.7% G genotypes and 44.7% P genotypes, with predominance of G2 (17%) and P[8] (66.7%) genotypes. 38.3% and 11.7% of the G and P genotype positive samples, respectively, showed no features of any of the G and P genotypes surveyed.
CONCLUSION: 38.3% and 11.7% G and P genotypes, respectively, showed no characteristics of the genotypes studied, suggesting that genotypes different to these are in circulation.
Key words: Gastroenteritis; Rotavirus Infections; Genotyping Techniques; Epidemiology Descriptive.
Introdução
As gastroenterites apresentam como sintoma comum a diarreia, que pode ser causada por uma ampla gama de patógenos, incluindo bactérias, vírus e protozoários.1 Seis categorias de vírus estão associadas à gatroenterite, sendo o rotavírus do grupo A (RVA) responsável por 30% dos casos da doença. Outros vírus causadores da gastroenterite são o adenovírus entérico, o astrovírus, o coronavírus e os calicivírus (Sapovirus; e Norovirus e sua espécie típica, o Norwalk) .2 A diarreia é responsável por uma em cada cinco mortes de crianças, cerca de 1,5 milhões a cada ano, matando mais crianças do que a síndrome da imunodeficiência adquirida - aids -, a malária e o sarampo juntos.1
A diarreia é a segunda principal causa de morte entre crianças menores de 5 anos de idade. A diarreia causada por rotavírus, especialmente, no ano de 2008, respondeu por 453,000 mortes de crianças nessa faixa etária em todo o mundo, por 37% das mortes por diarreia e por 5% de todas as mortes em menores de 5 anos.3
No Brasil, um estudo que avaliou a epidemiologia do RVA e sua associação com a morbidade e a mortalidade de crianças menores de 5 anos estimou que a diarreia por RVA foi a causa de mais de 92.400 hospitalizações e 850 mortes no ano de 2003.4
Outro estudo, realizado em 2003, com o objetivo de avaliar os aspectos epidemiológicos e clínicos da doença diarréica por RVA na cidade de Manaus, capital do Estado do Amazonas, Brasil, observou 46% de positividade para RVA em 380 amostras fecais de crianças com até 3 anos de idade.5
O rotavírus do grupo A é transmitido de pessoa a pessoa por via fecal-oral e pela via respiratória, ou ainda, mais raramente, pela água e alimentos e pelo contato com objetos contaminados; seu período de incubação varia de um a três dias.6,7 As principais manifestações clínicas da doença são vômitos, diarreia aquosa não sanguinolenta e febre. Os vômitos, geralmente mais intensos e duradouros que nas diarreias agudas provocadas por outros patógenos, juntamente com a diarreia aquosa, podem levar à desidratação e, conseqüentemente, ao aumento da mortalidade.
Os rotavírus apresentam partículas arredondadas de 100nm de diâmetro. A partícula viral completa é composta por triplo capsídeo protéico contendo o genoma de RNA (ácido ribonucléico) de dupla fita segmentado, que codifica proteínas estruturais e não estruturais.8 Esses segmentos de dupla fita de RNA codificam seis proteínas virais - VP1, VP2, VP3, VP4, VP6 e VP7 - e seis proteínas não estruturais - NSP1, NSP2, NSP3 NSP4 e NSP5 e NSP6. O capsídeo intermediário é constituído pela proteína VP6 e seu antígeno é usado para caracterizar oito diferentes grupos de rotavírus, classificados de A a H. O capsídeo interno ou 'core' consiste das proteínas VP1, VP2 e VP3; e o externo das proteínas, VP7 e VP4, que forma a base do sistema de classificação para os tipos G (VP7) e P (VP4).9
Até o momento, 23 tipos de rotavírus G e 30 tipos de rotavírus P foram identificados. Esses genótipos podem ser compartilhados entre humanos e animais. Entretanto, estudos têm demonstrado que as combinações G1P[8], G2P[4], G3P[8], G4P[8] e G9p[8] são mais comumente encontradas em humanos.10 Embora os sorotipos e genótipos citados já tenham sido encontrados em infecção em humanos, cinco combinações de genótipos P e G são mais prevalentes, em nível mundial. Essas combinações são geralmente encontradas entre o genótipo P[8], associado com os tipos G1, G3, G4 e G9, e o genótipo G2, associado ao genótipo P[4];11 porém, casos de combinações incomuns vêm sendo encontrados em vários países.12
No ano de 2006, o Programa Nacional de Imunizações implantou no Brasil a vacina contra rotavírus para crianças menores de seis meses. Essa vacina, denominada de Vacina Oral de Rotavírus Humano (VORH), licenciada no mercado internacional com o nome comercial de Rotarix®, é elaborada com vírus isolados de humanos atenuados para manter a capacidade imunogênica, não a patogênica.13 É uma vacina monovalente, ou seja, a cepa utilizada em sua composição possui apenas um genótipo: G1[P8] da cepa RIX4414.13
A partir da introdução da vacina contra RVA no Calendário Nacional de Imunização, a tendência esperada é de que o número de casos da doença por RVA e o número de óbitos decorrentes de suas consequências apresente um declínio. Entretanto, é de vital importância que se obtenha conhecimento da epidemiologia molecular do agente da doença, no sentido de se acompanhar possíveis alterações no cenário da doença desde o período pós-vacinação.
O presente estudo teve como principal objetivo caracterizar a diversidade das amostras de RVA humanos circulantes entre crianças na idade de 1 a 36 meses, na cidade de Manaus-AM, Brasil.
Métodos
Trata-se de um estudo descritivo. Foram incluídos os resultados de análises laboratoriais de amostras de fezes coletadas de 607 crianças na idade entre 1 e 36 meses com diarreia aguda, de ambos os sexos, atendidas em prontos-socorros públicos da cidade de Manaus, Estado do Amazonas, entre junho de 2004 e dezembro de 2006.
Manaus é a capital do Estado do Amazonas, localizado na Região Norte do Brasil. Segundo dados da Fundação Instituto Brasileiro de Geografia e Estatística (IBGE), em 2010, o município contava com uma população de 1.802,014 habitantes, dos quais 129.262 eram crianças menores de 3 anos de vida.14
Os pontos de coleta das amostras de fezes estavam distribuídos em diversos locais da cidade, com uma representatividade de 80% das instituições públicas de atendimento a crianças em Manaus-AM: Hospital e Pronto-Socorro da Criança (HPSC) da Zona Sul, HPSC da Zona Leste e HPSC da Zona Oeste da cidade, e Instituto de Saúde da Criança do Amazonas (ICAM). Apenas uma instituição de atendimento a crianças - que hoje representa 20% do total geral das instituições com essas características - não foi incluída no estudo porque sua inauguração aconteceu após o início da pesquisa. As amostras foram coletadas diariamente, incluindo os finais de semana e feriados.
Foram incluídas na pesquisa todas as crianças que apresentavam diarreia aguda cujos pais ou responsáveis legais, após serem devidamente esclarecidos sobre os objetivos do estudo, assinaram o Termo de Consentimento Livre e Esclarecido. Foram excluídas da pesquisa as crianças de origem indígena, por não se haver solicitado a devida autorização das organizações indígenas locais, conforme prevêem normas regulamentadoras para pesquisa envolvendo povos indígenas.15 A escolha unicamente de crianças moradoras do município de Manaus-AM não foi um critério de inclusão no estudo mas, entre todas as amostras coletadas, não havia uma única sequer pertencente a crianças moradoras de outro município.
Mediante análises laboratoriais das amostras fecais coletadas, determinaram-se as variáveis de positividade para RVA e o perfil da epidemiologia molecular dos genótipos G e P de RVA circulantes. As amostras de fezes foram coletadas em recipiente plástico de 10mL e armazenadas a -20oC, até o envio para análise no laboratório de virologia do Instituto Nacional de Pesquisas da Amazônia (INPA). O preparo das suspensões fecais foi realizado a 20%, em tampão Tris/Cálcio (Tris/HCl 0.1M; CaCl2 1.5mM; pH7.3).16 Foi realizada a triagem de todas as amostras (607) por eletroforese de RNA em gel de poliacrilamida (EGPA), para avaliação da positividade para rotavírus A.16
As amostras positivas pelo EGPA foram submetidas à genotipagem para identificação de VP7 (genótipo G) e VP4 (genótipo P) por transcrição reversa (RT-PCR), seguida de semi-nested multiplex na reação em cadeia de polimerase.17 Para tal, o genoma viral foi extraído a partir de 300µL de suspensão fecal a 20% (peso/volume), com o reagente Trizol (Invitrogen do Brasil). As suspensões de RNA foram estocadas a -20oC até seu uso na reação. "Primers" consenso Beg9 e End916 foram usados na primeira reação de PCR (30 ciclos) para amplificar o gene da VP7. O produto da primeira amplificação foi utilizado na segunda reação de amplificação pela PCR para a genotipagem de VP7 (genótipos G) (25 ciclos). Foram utilizados os seguintes "primers": aBT1 (G1), aCT2 (G2), aET3 (G3), aDT4 (G4) e aFT9 (G9).17 Para a genotipagem de P, foram usados os "primers" consenso 4Con2 e 4Con317 na primeira reação de PCR (30 ciclos). O produto dessa amplificação foi utilizado na segunda reação de amplificação para genotipagem dos genótipos P (25 ciclos), usando-se o conjunto de "primers": 1T-1 (P[8]), 2T-1 (P[4]), 3T-1 (P[6]), 4T-1 (P[9]) e 5T-1 (P[10]).17 Os produtos da reação de PCR foram analisados por eletroforese em gel de 1,5% de agarose, que continha 0,5µg de brometo de etídio por mL, e foi visualizado por iluminação de luz ultravioleta.
Por se tratar de estudo com seres humanos, o projeto foi submetido e aprovado pelo Comitê de Ética do Instituto Nacional de Pesquisas da Amazônia - INPA: Processo no 005/2004.
Resultados
De um total de 607 amostras de fezes de crianças com diarreia aguda, 152 (25,0%) apresentaram positividade para RVA pela análise genómica em eletroforese em gel de poliacrilamida. A análise das 152 amostras por RT-PCR demonstrou que em 68 amostras (44,7%), os genótipos P puderam ser identificados, em 47 (31,0%), foram identificados genótipos G, e 37 (24,3%) não amplificaram com os "primers" utilizados.
Das 47 amostras positivas para os genótipos VP7, os G1 e G2 foram identificados em sete amostras. Foram identificados os genótipos G9, G3 e G4 em cinco, três e uma amostra respectivamente. Detectou-se G1+G9 em seis amostras e G1+G3 em uma. Os "primers" utilizados para identificação dos genótipos G não amplificaram em cerca de um terço das amostras. (Tabela 1).

Entre as 68 amostras positivas para os genótipos de VP4, o genótipo P[8] foi identificado em 2/3 delas. O genótipo P[4] foi encontrado em nove amostras e os genótipos P[6], P[9] e P[10] foram encontrados cada um em uma amostra. Os genótipos P[4]+P[10] foram identificados em duas amostras; e os genótipos P[4]+P[6], em uma das amostras. Sete dessas 68 amostras não amplificaram com os "primers" utilizados para identificação dos genótipos P (Tabela 2).

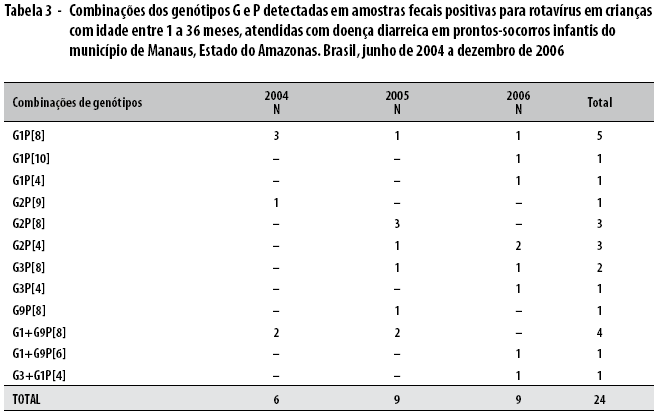
Pela segunda reação de amplificação pela PCR para a genotipagem dos genótipos G e P, foram identificadas 24 amostras que apresentaram combinações definidas para os dois genótipos. Essas combinações apareceram nas seguintes proporções: G1P[8] foi encontrado em cinco amostras; G1P[10], G1P[4], G2P[9], G3P[4] e G9P[8], em uma amostra; G2P[8] e G2P[4], em três amostras; o G3P[8] foi encontrado em duas amostras (8,33%); as combinações G1+G9P[8] foram encontradas em quatro amostras; e as combinações G1+G9P[6] e G1+G3P[4], ambas em uma amostra. Ressalta-se que não houve retestagem dessas amostras. O genótipo G1P[8] foi predominante durante os anos de estudo, seguido de uma combinação de G1+G9P [8] e dos genótipos G2P[8] e G2P[4] (Tabela 3).
Discussão
Este estudo encontrou uma positividade de 25% de rotavírus em amostras de fezes de crianças atendidas com gastrenterites na cidade de Manaus, Amazonas. Após a genotipagem das amostras positivas, a maioria revelou ser do genótipo P, seguida de amostras positivas para o genótipo G e de amostras que não amplificaram com os "primers" utilizados.
Estudos realizados no Brasil, nas últimas décadas, com crianças menores de 36 meses de idade, demonstram que a positividade para RVA no país apresentou uma variação de 11,9 a 48%.12,18 Na Região Amazônica, os RVA foram encontrados nas proporções de 18,4% em Belém, Estado do Pará,19 e de 46% em Manaus-AM. No presente estudo, desenvolvido ao longo de 30 meses, encontrou-se 25% de positividade para os rotavírus.
A vacina contra rotavírus introduzida no Brasil, incluída no Calendário do Programa Nacional de Imunizações desde 2006, tem mostrado eficácia na redução de casos graves da doença. Não obstante, estudo posterior a esse período encontrou genótipos diferentes dos conferidos pela vacina (G1P[8]), sugerindo que os genótipos encontrados podem estar a adquirir vantagem seletiva sobre os genótipos P[8],20 o que poderia levar ao aumento na ocorrência de casos graves da doença.
Estudo realizado em Manaus nos anos de 2007 e 2008, com crianças menores de 12 meses de idade, encontrou uma positividade de 9,5% das amostras para RVA,21 enquanto no presente estudo, a proporção dessa positividade foi de 25%. A partir da comparação desses dados, não se pode afirmar se houve uma redução dos casos de diarreia por RVA na cidade de Manaus-AM, uma vez que as populações dos dois estudos diferiram no fator idade das crianças analisadas. Mesmo assim, os resultados de ambas pesquisas indicam que o agente viral permaneceu sendo um problema de Saúde Pública infantil, a despeito da introdução da vacina no Calendário Nacional.
Neste estudo especialmente, constatou-se que cerca de um terço das amostras identificadas pelo método de RT-PCR, seguida de Semi-Nested Multiplex16 como genótipo G, não amplificaram com os "primers" utilizados para a caracterização do genótipo G. Tal fato sugere que genótipos diferentes dos pesquisados aqui, G1, G2, G3, G4 e G9, poderiam estar circulando na região de Manaus-AM. Um desses genótipos, o G5, foi encontrado também no Pará;12 o G12 também foi encontrado em estudo realizado em cinco Estados da Região Norte do Brasil.22
Os genótipos G1, G4 e G9 foram encontrados com maior prevalência em estudos realizados noutras partes do mundo, 10 embora outros genótipos G, como G5, G6, G8 e G10, venham apresentando significativa importância epidemiológica.23
O genótipo G2 tem sido encontrado com certa frequência no Brasil. Na década de 1990, em um estudo que monitorou as cepas de RVA na cidade do Rio de Janeiro-RJ, encontrou-se o genótipo G2 em 22% das amostras positivas para o genótipo G.23 Este mesmo genótipo foi encontrado ainda em Belém-PA, em 20% das amostras positivas para o genótipo G.13 O genótipo G9, identificado em uma em cada dez das amostras do presente estudo, também foi encontrado em estudo realizado no município de Goiânia-GO, em 34% das amostras positivas para o genótipo G.24
Os genótipos P[8], P[4], P[6] e P[9] são, reconhecidamente, os genótipos P mais encontrados mundialmente.24 No estudo ora apresentado, os genótipos P[8] e P[4] foram responsáveis, respectivamente, por 66,2% e 13,2% das amostras positivas para o genótipo P. Este achado demonstra que o perfil circulante no município de Manaus-AM foi semelhante ao definido por outros estudos realizados no Brasil, como o de Santos e colaboradores (2003),23 que encontrou o genótipo P[8] em 45,9% e o genótipo P[4] em 18.9% das amostras estudas no município do Rio de Janeiro-RJ.
No mundo, cinco combinações de genótipos G e P são consideradas de maior importância epidemiológica: G1P[8], G2P[4], G3P[8] e G4P[8] e G9P[8].22 Dos quatro tipos mais comuns de combinações, três foram encontradas neste estudo: o G1P[8], responsável por 12,5% das combinações; o G2P[4], presente em cinco das combinações; e o G3P[8] em duas combinações.
O presente estudo identificou que a maioria das amostras foi genotipada como G1P[8], concordando com pesquisas que demonstraram ser esse genótipo de alta prevalência.22
Neste estudo, a combinação de genótipos G1P[8] e sua variação de G1+G9 P[8] apresentaram maior prevalência nos anos de 2004 e 2005, período anterior à introdução da vacina. Somente um caso foi constatado em 2006, indicando a eficácia da vacina logo após sua introdução no Calendário Nacional de Imunização. A partir de 2005, o genótipo G2P[4] já foi constatado em uma amostra (4,7%), e em duas amostras no ano de 2006 (8,33%). Este achado sugere que, após a introdução da vacina no Calendário Nacional, outras combinações de genótipos foram detectadas na epidemiologia do RVA. Situação semelhante foi constatada em estudo realizado no Rio de Janeiro-RJ entre 2005 e 2007, quando a combinação G2P[4] foi identificada em 1,4% das amostras em 2005 e em 41% das amostras em 2006.20
Foi constatado que 40% das combinações encontradas neste estudo foram caracterizadas como combinações de genótipos P[4] ou P[8] e combinações de P[4]P[10] ou P[4]P[6] com genótipos caracterizados como G que, entretanto, não foram identificadas na segunda amplificação pelo "primers" utilizados para caracterização desse genótipo. O mesmo ocorreu com o genótipo G9, encontrado em combinações do genótipo P que tampouco foram identificadas pelos "primers" utilizados na segunda amplificação (5%). Quatro combinações apresentaram características negativas, tanto para os genótipos G quanto para os P pesquisados (uma em cada dez). O número elevado desse tipo de combinação pode ser um indicativo da circulação de genótipos P na região estudada; e principalmente de genótipos G, que passaram a ser encontrados em estudos mais recentes.22 Um desses genótipos é o G5, encontrado no Pará,12 e também no Rio de janeiro em 2005.23 Outros genótipos G que podem ser citados neste estudo são o G8 e o G10, que embora considerados atípicos, já foram relatados em outro estudo.26 Outros genótipos G e P que podem ser citados são o G11, o G12 e o P[25], identificados em estudo realizado no Nepal, onde, na caracterização molecular do RVA em amostras de fezes de crianças e adultos, foram encontrados os genótipos G11 e G12 e o genótipo P[25], este último na forma da combinação P[25]G11.27
Os dados encontrados neste estudo demonstram que genótipos anteriormente tidos como restritos a animais, que ocorrem em humanos como resultado de eventos de transmissão zoonótica - a exemplo do G5 encontrado em suínos28 ou do G6, do G8 e do G10 isolados inicialmente em bovinos29 -, podem estar sendo transmitidos de animais para humanos, seja como vírus inteiros ou como reorganização de segmentos de genes.
Os "primers" utilizados neste estudo possibilitaram a identificação de genótipos P e G, comumente encontrados em estudos dessa natureza: os genótipos G1, G2, G3, G4 e G9, e ainda, P[8], P[4], P[6], P[9] e P[10]. Porém, a utilização desses "primers" foi insuficiente para definir mais de um terço das amostras positivas para o genótipo G, e mais de uma em cada dez amostras positivas para o genótipo P, sugerindo que outros genótipos G e P possam estar em circulação na região estudada.
O fato de um número elevado de amostras positivas para RVA humano apresentar combinações com os genótipos P ou G não genotipados demonstra que devem ser desenvolvidos estudos utilizando "primers", para identificação de genótipos G e P que tanto garantam o reconhecimento de possíveis alterações genéticas desses genótipos - fato já descrito em outro estudo30 - como sejam compatíveis com genótipos que vêm sendo encontrados em outras regiões do Brasil e do mundo.
O rotavírus ainda é um dos principais agentes causadores de doença diarreica, levando a óbito milhares de crianças em todo o mundo. Este cenário exige a mobilização de autoridades sanitárias nacionais e internacionais, na tentativa de diminuir os casos da doença. O conhecimento do perfil molecular do rotavírus é um norteador para o desenvolvimento de ações que possam minimizar o número desses casos da doença e seus efeitos na população infantil.
Agradecimentos
À Secretaria de Estado de Saúde do Amazonas, por permitir a coleta de amostras nos Prontos-Socorros da Criança da Zona Leste, Zona Sul e Zona Oeste, e no Instituto da Criança do Amazonas.
Contribuição dos autores
Melo GZS participou da coleta das amostras, análise de dados e redação do manuscrito.
Costa CA participou da revisão da análise de dados, discussão, escolha da metodologia e redação do manuscrito.
Santos IGC participou das análises laboratoriais.
Todos os autores aprovaram a versão final do manuscrito.
Referências
1. World Health Organization. Diarrhoea: why children are still dying and what can be done. Geneva: UNICEF/WHO; 2009.
2. Sabrá A, Sabrá S, Rodrigues G. Diarreias Infecciosas. In: Tavares W, Marinho LAC, organizadores. Rotinas de diagnóstico e tratamento das doenças infecciosas e parasitárias. 2. ed. São Paulo: Atheneu; 2010. p. 323-9.
3. Tate JE, Burton AH, Boschi-Pinto C, Steele AD, Duque J, Parashar UD. 2008 estimate of worldwide rotavirus-associated mortality in children younger than 5 years before the introduction of universal rotavirus vaccination programmes: a systematic review and meta-analysis. Lancet Infect Dis [Internet]. 2012 Feb [cited 2013 Fev 25];12(2):136-41. Disponível em:http://www.rehydrate.org/rotavirus/pdf/rotavirus-associated-mortality-in-children.pdf
4. Sartori AM, Valentim J, Soárez PC, Novaes HMD. Rotavirus morbidity and mortality in children in Brazil. Rev Panam Salud Publica. 2008;23(2):92-100.
5. Rocha LA. Aspecto epidemiológico e clínico do rotavírus em crianças hospitalizadas com diarreia aguda em Manaus, Brasil [dissertação]. Manaus (AM): Universidade Federal do Amazonas; 2003.
6. Sonnen G, Henry N. Rotavírus. In: Wilson W, Sande M, organizadores. Doenças Infecciosas: diagnóstico e tratamento. Porto Alegre: Artmed; 2004.
7. Heymann DL. Control of communicable diseases manual. 19th ed. Washigton D.C: American Public Health Association; 2008.
8. Souza CPR, Araújo DV. Análise de custo da hospitalização por rotavírus e o impacto do tratamento com Nitazoxanida. Rev Panam Infectol. 2009;11(1):33-7.
9. Matthijnssens J, Otto PH, Ciarlet M, Desselberger U, Van Ranst M, Johne R. VP6-sequence-based cutoff values as a criterion for rotavirus species demarcation. Arch Virol. 2012 Jun;157(6):1177-82.
10. Estes MK, Kapikian AZ. Rotaviruses. In: Knipe DM, Howley PM, editors. Fields virology. 5th ed. Philadelphia: Lippincott Williams and Wilkins; 2007. p. 1917-74.
11. Castello AA, Arguelles MH, Rota RP, Olthoff A, Jiang B, Glass RI, et al. Molecular epidemiology of group A rotavirus diarrhea among children in Buenos Aires, Argentina, from 1999 to 2003 and emergence of the infrequent genotype G12. J Clin Microbiol. 2006;44(6):2046-50.
12. Mascarenhas JDP, Linhares AC, Gabbay YB, Leite JPG. Detection and Characterization of Rotavirus G and P Types from Children Participating in a Rotavirus Vaccine Trial in Belém, Brazil. Mem Inst Oswaldo Cruz. 2002 jan;97(1):113-7.
13. Ministério da Saúde (BR). Secretaria de Vigilância em Saúde. Departamento de Vigilância Epidemiológica. Vigilância Epidemiológica e prevenção pela Vacina Oral de Rotavírus Humano - VORH. Brasília: Ministério da Saúde; 2006.
14. IBGE-cidades@ [base de dados na internet]. Brasília: IBGE; [s.d.] [citado 26 maio 2004]. Disponível em: http://www.ibge.gov.br/cidadesat/topwindow.htm?1
15. Brasil. Ministério da Saúde. Conselho Nacional de Saúde. Resolução MS/CNS no 304 de 09 de agosto de 2000. Aprova as normas para pesquisas envolvendo seres humanos. Área de Povos Indígenas. [Brasília]: Conselho Nacional de Saúde; 2000.
16. Pereira HG, Azeredo RS, Leite JPG, Candeias JAN, Rácz ML, Linhares AC, et al. Electrophoretic study of the genome of human rotaviruses from Rio de Janeiro, Sao Paulo and Pará, Brazil. J Hyg. 1983 Feb;90(1):117-25.
17. Gouvea V, Glass RI, Woods P, Taniguchi K, Clark HF, Forrester B, et al. Polymerase chain reaction amplification and typing of rotavirus nucleic acid from stool specimens. J Clin Microbiol. 1990 Feb;28(2):276-82.
18. Serravalle K, Santos N, Sardi SI, Silva SPS, Junior HCR, Matos AP, et al. Molecular characterization of group A rotavirus isolates from hospitalized children in Salvador, Bahia, Brazil. Braz J Infect Dis. 2007;11(1):35-9.
19. Linhares AC, Gabbay YB, Mascarenhas JDP, Freitas RB, Oliveira CS, Bellesi N, et al. Estudo prospectivo das infecções por rotavírus em Belém, Pará, Brasil: uma abordagem clínico-epidemiológica. J Pediatr. 1994;70(4):220-5.
20. Carvalho-Costa FA, Araújo IT, Assis RM, Fialho AM, Bóia MN, Alves DPD, et al. Rotavirus Genotype Distribution after Vaccine Introduction, Rio de Janeiro, Brazil. Emerg Infect Dis. 2009 Jan;15(1):95-7.
21. Pinto CC. Prevalência de rotavírus em crianças de até 01 ano de idade em Manaus-AM. [dissertação]. Manaus (AM): Universidade Federal do Amazonas; 2009.
22. Soares LS, Lobo PS, Mascarenhas JDP, Neri DL, Guerra SFS, Oliveira ASL, et al. Identification of lineage III of G12 rotavirus strains in diarrheic children in the Northern Region of Brazil between 2008 and 2010. Arch Virol. 2012 Jan;157(1):135-9.
23. Santos N, Soares CC, Volotão EM, Albuquerque MCM, Hoshino Y. Surveillance of Rotavirus Strains in Rio de Janeiro, Brazil, from 1997 to 1999. J Clin Microbiol. 2003 Jul;41(7):3399-3402.
24. Costa PSS, Cardoso DDP, Grisi SJFE, Silva PA, Fiaccadori F, Souza MBLD, et al. Infecções e reinfecções por Rotavírus A: genotipagem e implicações vacinais. J Pediatr. 2004 abr; 80(2):119-22.
25. Soares LS, Mascarenhas JDP, Gabbay YB, Gusmão RHP, Linhares AC. Molecular characterization of G1 human rotaviruses detected in children from Belém, Pará, Brazil. Rev Pan-Amaz Saude. 2010 mar;1(1):125-30.
26. Carvalho-Costa FA, Volotão EM, Assis RMS, Fialho AM, Andrade JSR, Rocha LN, et al. Laboratory-based Rotavirus Surveillance During the Introduction of a Vaccination Program, Brazil, 2005-2009. Pediatr Infect Dis J. 2011 Jan;30(1 Suppl):S35-41.
27. Uchida R, Pandey BD, Sherchand JB, Ahmed K, Yokoo M, Nakagomi T, et al. Molecular Epidemiology of Rotavirus Diarrhea among Children and Adults in Nepal: Detection of G12 Strains with P[6] or P[8] and a G11P[25] Strain. J Clin Microbiol. 2006 Oct;44(10):3499-505.
28. Silva MF, Tort LF, Goméz MM, Volotão EM, Mendonça MC, Bello G, et al. VP7 Gene of human rotavirus A genotype G5: Phylogenetic analysis reveals the existence of three different lineages worldwide. J Med Virol. 2011 Feb;83(2):357-66.
29. Laird AR, Ibarra V, Ruiz-Palacios G, Guerrero ML, Glass RI, Gentsch JR. Unexpected Detection of Animal VP7 Genes among Common Rotavirus Strains Isolated from Children in Mexico. J Clin Microbiol. 2003 Sep;41(9):4400-3.
30. Aladin F, Nawaz S, Iturriza-Gómara M, Gray J. Identification of G8 rotavirus strains determined as G12 by rotavirus genotyping PCR: Updating the current genotyping methods. J Clin Virol. 2010 Apr;47(4):340-4.
 Endereço para correspondência:
Endereço para correspondência:
Giane Zupellari dos Santos Melo -
Av. Via Láctea, no 607,
Condomínio Islamorada,
Conjunto Morada do Sol,
Torre Norte, Apto. 203,
Bairro Aleixo, Manaus-AM, Brasil.
CEP: 69060-085
E-mail: gzsantos3@hotmail.com
Recebido em 22/12/2012
Aprovado em 18/04/2013
*Estudo apresentado como defesa de dissertação para o Programa de Mestrado em Patologia Tropical da Universidade Federal do Amazonas - UFAM - em 2008. A pesquisa, coordenada pelo Dr. Cristóvão Alves da Costa, recebeu auxílio financeiro da Fundação de Amparo a Pesquisa do Estado do Amazonas (FAPEAM no 236/2003).