Contribuições do estudo
Principais resultados
O custo para realização da autocoleta de swab nasofaríngeo combinada com o teste molecular (RT-PCR) para detecção do SARS-CoV-2 utilizando a técnica pool testing foi cerca de dez vezes menor do que nos métodos habituais envolvendo testagem individual.
Implicações para os serviços
A autocoleta de swab é uma estratégia que exige infraestrutura mínima e teve boa adesão dos participantes. Combinada com a técnica de pool testing, mostrou-se economicamente viável e válida para a pesquisa de SARS-CoV-2 em inquéritos epidemiológicos.
Perspectivas
A autocoleta de swabs nasofaríngeos permite a obtenção de amostras de boa qualidade e, combinada com a técnica pool testing, poderá viabilizar a expansão da testagem para SARS-CoV-2 e incrementar o controle de surtos em escolas e ambientes de trabalho.
Introdução
A COVID-19 (Coronavirus Disease-19), doença causada pelo SARS-CoV-2 (Severe Acute Respiratory Syndrome Coronavirus 2), já acometeu mais de 200 milhões de indivíduos e causou mais de 4 milhões de mortes em todo o mundo, 13% delas no Brasil.1 Apesar de a vacinação no país ter-se iniciado em janeiro de 2021, cumpre manter um sistema de vigilância epidemiológica capaz de detectar precocemente eventuais surtos e promover ações de contenção da transmissão da doença.2
Há preocupação com grupos sob elevado risco de contágio, a exemplo da comunidade escolar no retorno às atividades presenciais.3 Nos Estados Unidos, a estratégia de testagem em pool de indivíduos assintomáticos foi adotada na Duke University e nas escolas do estado de Massachussets, no retorno às atividades presenciais, visando rastrear e deter a propagação do vírus.3,4
O pool testing consiste na realização do teste molecular RT-PCR (Reverse Transcriptase-Protein Chain Reaction) simultaneamente, em grupos de amostras.4 Essa técnica tem sido utilizada em vários países, por ser econômica e eficaz para testagem populacional.3,5,6 Em indivíduos assintomáticos, a realização do pool testing auxilia na detecção precoce, permitindo interromper a cadeia de transmissão, especialmente em grupos com maior exposição ao SARS-CoV-2.3,6-9
A autocoleta também representa uma opção econômica para testagem em massa, pois não necessita de profissionais treinados e equipamentos de proteção individual (EPIs). Estudos comparativo de amostras de swab nasofaríngeo coletadas por profissionais de saúde e por autocoleta mostraram resultados semelhantes.8,9
Dada a urgência de aumentar a cobertura de testes para COVID-19, o objetivo do estudo foi demonstrar a viabilidade da utilização combinada da autocoleta de swab nasofaríngeo com pool testing para detecção do SARS-CoV-2 em inquéritos epidemiológicos.
Métodos
A presente experiência, de delineamento transversal, realizada em fevereiro de 2021, é um subprojeto de pesquisa longitudinal prospectiva conduzida em Belo Horizonte, MG, com o objetivo de avaliar a expansão da realização de RT-PCR para SARS-CoV-2 por pool testing em pessoas com síndrome gripal.
Todos os estudantes do 9° ao 12° períodos do Curso de Medicina da Universidade Federal de Minas Gerais, no cumprimento dos estágios hospitalares obrigatórios, foram convidados a participar do estudo. Caso aceitassem, responderiam a um questionário para caracterização da amostra com informações sobre idade (em anos), sexo (masculino; feminino) e serviço onde realizava o estágio hospitalar. O questionário foi respondido utilizando-se o Google Forms. A constituição da amostra de participantes foi de conveniência, sem cálculo amostral prévio.
Os participantes forneceram amostras de swab nasofaríngeo, obtidas por autocoleta realizada em cabines individuais, sem supervisão direta de profissionais, e contou com a exposição de cartazes sobre a técnica. Os estudantes também receberam vídeo instrucional sobre a autocoleta, antes do procedimento. O swab foi descartado em recipiente próprio, dentro das próprias cabines. O tubo contendo a amostra e solução de inativação e transporte viral10 foi identificado, lacrado e entregue aos pesquisadores, que aguardavam do lado de fora das cabines.
Para preparação dos pools, foram adicionados 47 microlitros de cada amostra individual em microtubos de 1,5 mL. As amostras foram processadas para extração de RNA, segundo o protocolo da empresa QIAGEN Inc. (Alemanha). As reações RT-PCR foram realizadas com as sondas para o gene endógeno da RNaseP humana e o gene (E) que codifica o envelope viral, utilizando-se o termociclador QuantStudio 5 (Applied Biosystems), de acordo com o protocolo Charité.11 Na estratégia de testagem em pool, se o resultado de um pool é detectável, faz-se necessário o processamento individual de todas as amostras presentes nesse agrupamento.5 As amostras ficaram estocadas a 4°C.
Conforme descrito na literatura, quando a prevalência de COVID-19 na comunidade é de 1%, o tamanho ótimo do pool é de 11 amostras (Quadro 1).12,13 Considerando-se que nenhum dos participantes estava sintomático no momento da coleta, optou-se pela testagem em pools de dez amostras, o que seria adequado para detectar RNA viral mesmo com carga viral muito baixa.12-14 Foram preparados 15 pools contendo dez amostras e um pool com quatro amostras.
Quadro 1 Estimativa de tamanhos ótimos de pools para realização de RT-PCR para SARS-CoV-2,a segundo a prevalência de COVID-19 na comunidade
| Prevalência de COVID-19 na comunidade (%) | Tamanho ótimo do pool (número de amostras por pool) |
|---|---|
| 1 | 11 |
| 2 | 8 |
| 3 | 6 |
| 4 | 6 |
| 5 | 5 |
| 6 | 5 |
| 7 | 4 |
| 8 | 4 |
| 9 | 4 |
| 10 | 4 |
| 20 | 3 |
Fonte: Costa et al.12 e Cherif et al.13. a) RT-PCR (Reverse Transcriptase-Protein Chain Reaction) para SARS-CoV-2 (Severe Acute Respiratory Syndrome Coronavirus 2).
Após o processamento das amostras em pool, quatro pools de dez amostras foram processados individualmente, para avaliar possíveis falhas no processo de autocoleta. Nesta etapa, procurou-se verificar se as amostras continham RNA endógeno do indivíduo. O cálculo do número de amostras/pools necessários para essa verificação foi realizado com o uso do software MatLab, estimando-se 90% de probabilidade de detectar pelo menos uma amostra inadequada caso tivessem ocorrido erros em 5% das amostras.
Os resultados foram apresentados a partir de frequências absolutas e relativas. Os custos, em reais, para extração e reação do RT-PCR individual e em pool, considerando-se apenas os materiais plásticos e reagentes, foram calculados com base nos valores de mercado praticados em agosto de 2021 e disponíveis em sítios eletrônicos dos fornecedores de produtos biomédicos. Foram incluídos no cálculo os EPIs necessários para realização da coleta das amostras de swab nasofaríngeo, se esta fosse realizada por um profissional de saúde e não por autocoleta. Foram realizadas, também, simulações de custos, caso alguns dos pools apresentassem resultado detectável, utilizando-se o programa Excel. A simulação considerou o custo dos procedimentos realizados em amostras individuais ou em pools, a partir do custo dos materiais e insumos calculados na etapa anterior.
O projeto do estudo foi aprovado pelo Comitê de Ética em Pesquisa da Universidade Federal de Minas Gerais (CEP/UFMG): Certificado de Apresentação para Apreciação Ética (CAAE) nº 35074720.3.0000.5149. Todos os estudantes incluídos na amostra assinaram o Termo de Consentimento Livre e Esclarecido em formato eletrônico.
Resultados
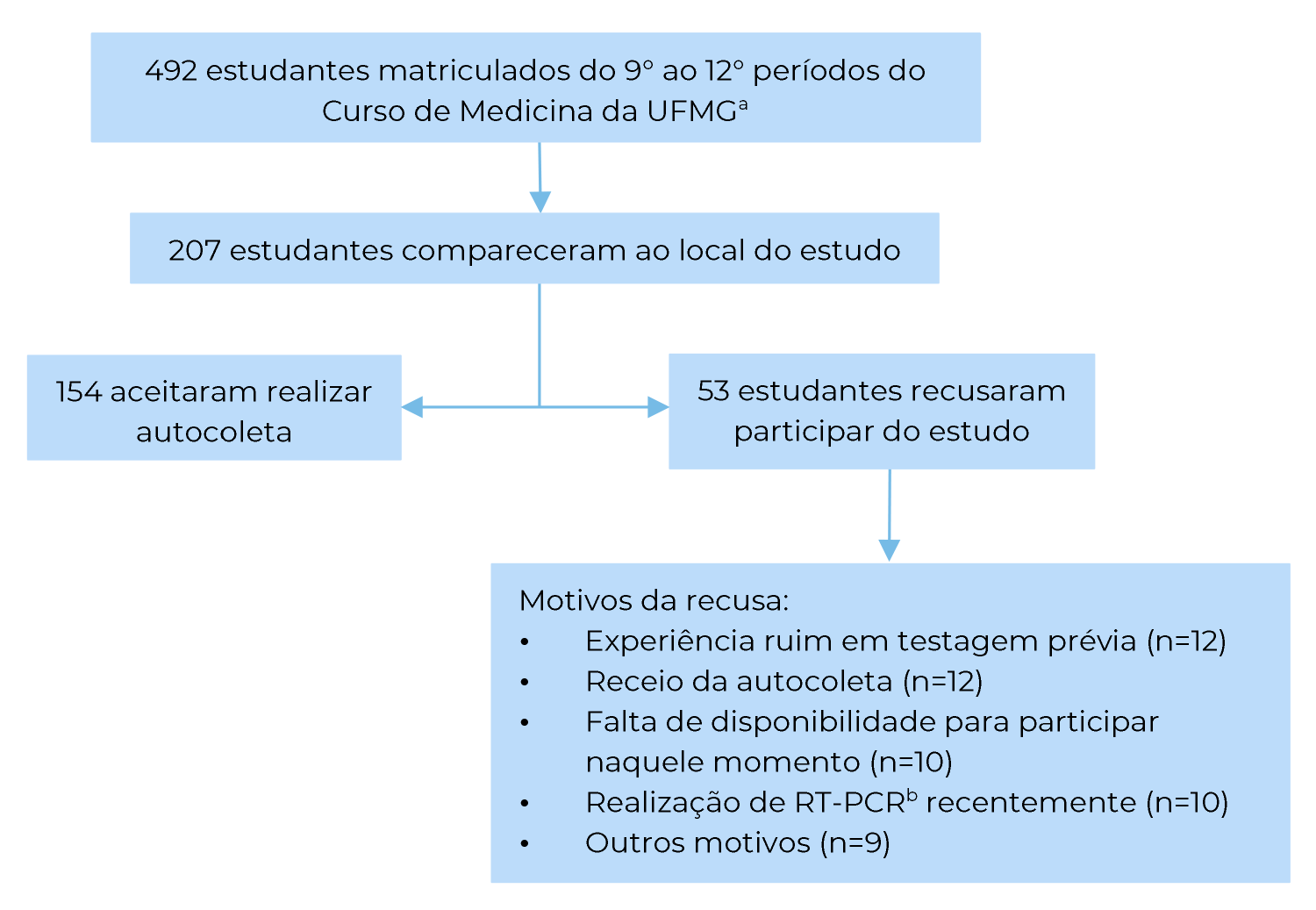
Dos 492 estudantes matriculados do 9° ao 12° períodos, 207 (42,1%) compareceram nos dias previamente agendados pelo Colegiado de curso para realização de procedimentos acadêmicos, sendo que 154 aceitaram participar da pesquisa (74,4% dos presentes nos dias agendados). Mais da metade dos participantes era do sexo masculino (54,7%) e tinha entre 20 e 24 anos de idade (56,0%). Todos os participantes realizavam estágio curricular obrigatório em hospitais da rede pública de saúde do município.
Dos 207 estudantes presentes na Faculdade de Medicina nos dias de realização do estudo, 53 recusaram-se a realizar a autocoleta do swab (25,6%). As principais justificativas para a recusa foram: experiência ruim em testagem prévia (n=12), receio da autocoleta (n=12), falta de disponibilidade para participar naquele momento (n=10), realização de RT-PCR recentemente (n=10) e outros motivos (n=9). A Figura 1 apresenta as etapas de composição da amostra.
a) UFMG: Universidade Federal de Minhas Gerais; b) RT-PCR: Reverse Transcriptase-Protein Chain Reaction para SARS-CoV-2 (Severe Acute Respiratory Syndrome Coronavirus 2).
Figura 1 Fluxograma para composição da amostra do estudo, composta de estudantes do 9° ao 12° períodos do Curso de Medicina da Universidade Federal de Minas Gerais, cursando estágios hospitalares obrigatórios, Belo Horizonte, 2021
A obtenção de amostras durou cerca de 5 minutos por pessoa. Todos os participantes receberam o resultado da testagem após dois dias úteis. Em nenhum dos pools foi detectada a presença de RNA viral. O RNA endógeno foi detectado adequadamente nas 40 amostras examinadas individualmente para analisar a qualidade do material obtido por autocoleta.
A Tabela 1 apresenta os custos do procedimento adotado no presente estudo, comparados aos custos do procedimento habitual (coleta de swab por um profissional de saúde e processamento individual das amostras) e a outros cenários hipotéticos. O valor final por amostra, no presente estudo, foi cerca de dez vezes menor do que o valor por amostra no procedimento habitual. O custo por amostra aumenta progressivamente, à medida que mais pools apresentam resultado detectável. Mesmo se metade dos pools precisassem ser processados individualmente, realizando-se autocoleta do swab e testagem inicial em pools de dez amostras, o valor por amostra corresponderia à metade do valor estimado no procedimento habitual.
Tabela 1 Custos, em reais, dos materiais e reagentes para realização do procedimento adotado no presente estudo,a comparado com o procedimento habitualb e outros cenários hipotéticos, para a testagem de estudantes do 9° ao 12° períodos do Curso de Medicina da Universidade Federal de Minas Gerais, cursando estágios hospitalares obrigatórios, Belo Horizonte, 2021
| Materiais e reagentes, por procedimentos | Preço unitárioe (R$) | Número de amostras | Valor dos materiais e reagentes (R$) | Total (pool + testes individuais) (R$) | Valor final por amostra (R$) |
|---|---|---|---|---|---|
| Procedimento adotado na pesquisaa | |||||
| RT-PCR processado em pools | 36,96 | 16 | 591,36 | 591,36 | 3,84 |
| Procedimento habitualc | |||||
| EPI para coleta de swabd | 3,00 | 154 | 462,00 | 6.153,84 | 39,96 |
| RT-PCR processado individualmente | 36,96 | 154 | 5.691,84 | ||
| Outros cenários hipotéticos com autocoleta do swab + processamento de 150 amostras, inicialmente em pools de dez amostras | |||||
| Cenário 1: 14 pools não detectáveis + 1 pool detectável | 36,96 | 15 | 554,40 | 924,00 | 6,16 |
| 10 amostras processadas individualmente | 36,96 | 10 | 369,60 | ||
| Cenário 2: 13 pools não detectáveis + 2 pools detectáveis | 36,96 | 15 | 554,40 | 1.293,60 | 8,62 |
| 20 amostras processadas individualmente | 36,96 | 20 | 739,20 | ||
| Cenário 3: 12 pools não detectáveis + 3 pools detectáveis | 36,96 | 15 | 554,40 | 1.663,20 | 11,09 |
| 30 amostras processadas individualmente | 36,96 | 30 | 1.108,80 | ||
| Cenário 4: 11 pools não detectáveis + 4 pools detectáveis | 36,96 | 15 | 554,40 | 2.032,80 | 13,55 |
| 40 amostras processadas individualmente | 36,96 | 40 | 1.478,40 | ||
| Cenário 5: 10 pools não detectáveis + 5 pools detectáveis | 36,96 | 15 | 554,40 | 2.402,40 | 16,02 |
| 50 amostras processadas individualmente | 36,96 | 50 | 1.848,00 | ||
| Cenário 6: 9 pools não detectáveis + 6 pools detectáveis | 36,96 | 15 | 554,40 | 2.772,00 | 18,48 |
| 60 amostras processadas individualmente | 36,96 | 60 | 2.217,60 | ||
| Cenário 5: 8 pools não detectáveis + 7 pools detectáveis | 36,96 | 15 | 554,40 | 3.141,60 | 20,94 |
a) Autocoleta do swab nasofaríngeo + RT-PCR (Reverse Transcriptase-Protein Chain Reaction para SARS-CoV-2) realizado em pool testing; b) Coleta do swab nasofaríngeo por profissional de saúde + processamento individual do RT-PCR; c) 15 pools com 10 amostras e 1 pool com 4 amostras (total = 154 participantes), sendo todos os pools com resultado não detectável; d) Considerando-se todos os equipamentos de proteção individual (EPIs) necessários para um profissional trabalhar 8 horas/dia e coletar 154 swabs nasofaríngeos (máscara PFF2, faceshield, luvas, capotes, toucas e aventais), excluindo os custos com profissional para coleta; e) Valores praticados em agosto de 2021.
Discussão
Os resultados mostraram que, em relação à técnica de autocoleta, (i) houve adesão da maioria dos convidados, (ii) necessitou-se de infraestrutura mínima e (iii) a autocoleta pôde ser realizada rapidamente, resultando em amostras com qualidade aceitável. A detecção do RNA endógeno, nas 40 amostras avaliadas individualmente, sugere que não ocorreram erros na autocoleta do swab nasofaríngeo.
Com relação à estratégia de autocoleta, em estudo conduzido nos Estados Unidos, Guest et al. demonstraram que a maioria das amostras de esfregaço orofaríngeo coletadas pelos próprios participantes foram adequadas para o teste de RNA de SARS-CoV-2.15 A ausência de falhas significativas no processo de autocoleta favorece a realização de inquéritos populacionais ou com grupos específicos, dispensando a presença de profissional treinado, aumentando a capacidade de testagem, reduzindo a exposição do profissional de saúde e os custos com EPIs, indispensáveis nesse procedimento.8 Portanto, os resultados da presente pesquisa indicam que a autocoleta é um recurso útil para vigilância da infecção por COVID-19 em indivíduos assintomáticos.
Observou-se resistência de parte dos universitários em realizar o procedimento, se não por simples temor, pela lembrança de experiências anteriores desagradáveis. Esses elementos sugerem a necessidade de maior sensibilização da população-alvo, no sentido de promover a adesão à autocoleta.9 Entretanto, a maioria dos convidados aderiu à autocoleta e foi capaz de realizar o procedimento com as instruções recebidas.
O custo médio dos procedimentos depende da prevalência da COVID-19 na comunidade: quanto maior a prevalência, maior a probabilidade de as amostras terem de ser processadas individualmente.12-14 A análise das amostras obtidas por autocoleta utilizando pool testing mostrou-se mais econômica do que se as amostras fossem analisadas individualmente, inclusive em cenários hipotéticos, nos quais alguns pools mostrassem resultados detectáveis.
Para coletar, processar e apresentar os resultados, foram necessários dois dias úteis. A rápida liberação dos resultados permite a adoção de ações de vigilância epidemiológica cabíveis, o que é particularmente relevante para o controle de surtos em comunidades fechadas, como as de estudantes e trabalhadores.3,4,7-9 A qualidade das amostras foi verificada, validando o resultado encontrado. A técnica de pool testing viabilizou a testagem rápida de número relativamente grande de pessoas, com importante redução dos custos.
Como limitação do estudo, aponta-se que o tamanho da amostra foi menor do que o esperado. A pesquisa foi realizada em dois dias, quando todos os estudantes de medicina cursando os estágios obrigatórios deveriam comparecer à universidade para procedimentos acadêmicos. No entanto, muitos estudantes optaram por realizar tais procedimentos remotamente, reduzindo o número de elegíveis para o estudo. Apesar disso, o número de participantes foi suficiente para o delineamento proposto. Estudos de viabilidade são relevantes para a saúde pública, podendo contribuir com o planejamento e realização de estudos maiores. Seria interessante manter o monitoramento desses estudantes, adotando-se as estratégias descritas, desde que continuem a atuar em cenários com maior risco de contaminação pelo SARS-CoV-2.
Conclui-se que as estratégias de autocoleta de swab nasofaríngeo e pool testing, utilizadas em conjunto, implicam economia de tempo, recursos materiais e humanos, mostrando-se economicamente viáveis, seja para a realização de inquéritos populacionais, seja na agilização de medidas para contenção de surtos de COVID-19.










 texto en
texto en