Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista Pan-Amazônica de Saúde
versão impressa ISSN 2176-6215versão On-line ISSN 2176-6223
Rev Pan-Amaz Saude v.1 n.1 Ananindeua mar. 2010
http://dx.doi.org/10.5123/S2176-62232010000100015
ARTIGO ORIGINAL | ORIGINAL ARTICLE | ARTÍCULO ORIGINAL
Caracterização fenotípica e genotípica de Serratia marcescens provenientes de Unidade Neonatal de Referência em Belém, Pará, Brasil
Phenotypic and genotypic characterization of Serratia marcescens from a Neonatal Unit in Belém, Pará State, Brazil
La caracterización fenotípica y genotípica de Serratia marcescens proveniente de la Unidad de Neonatología de Referencia de Belém (Pará, Brasil)
Raimundo Gladson Corrêa CarvalhoI; Irna Carla do Rosário Souza CarneiroII; Marcelo Sena PinheiroIII; Surama da Costa PinheiroIV; Paulo Sérgio Roffe AzevedoIV; Schirley Dias dos SantosV; Ana Roberta Fusco da CostaVI; Francisco Lúzio de Paula RamosVI; Karla Valéria Batista LimaVI
ICurso de Pós-graduação em Análises Clínicas, Centro Universitário do Pará, Belém, Pará, Brasil. Laboratório de Patologia Clínica e Laboratorial Dr. Paulo Azevedo, Belém, Pará, Brasil
IIDepartamento de Medicina, Universidade Federal do Pará, Belém, Pará, Brasil
IIICurso de Pós-graduação em Análises Clínicas, Centro Universitário do Pará, Belém, Pará, Brasil
IVLaboratório de Patologia Clínica e Laboratorial Dr. Paulo Azevedo, Belém, Pará, Brasil
VCurso de Pós-graduação em Análises Clínicas, Centro Universitário do Pará, Belém, Pará, Brasil. Seção de Bacteriologia e Micologia, Instituto Evandro Chagas/SVS/MS, Ananindeua, Pará, Brasil
VISeção de Bacteriologia e Micologia, Instituto Evandro Chagas/SVS/MS, Ananindeua, Pará, Brasil
Endereço para correspondência
Correspondence
Dirección para correspondencia
RESUMO
A Serratia marcescens tem sido relatada como importante agente de infecções relacionadas à saúde, destacando-se por apresentar elevado nível de resistência intrínseca aos antimicrobianos usados em neonatologia, além de persistir por longos períodos no ambiente hospitalar. Neste trabalho foram avaliadas, por métodos fenotípicos e moleculares, S. marcescens recuperadas a partir de colonização do trato gastrointestinal ou sepse tardia em neonatos internados em Unidade Neonatal em Belém. A identificação das S. marcescens e o teste de sensibilidade foram realizados por meio de sistema automatizado Vitek (BioMérieux); a suscetibilidade ao ertapenem foi avaliada com auxílio de disco contendo 10 µg da droga (Oxoide). A genotipagem foi feita por ERIC-PCR usando os primers ERIC1 (5'-TGAATCCCCAGGAGCTTACAT-3') e ERIC2 (5'-AAGTAAGTGACTGGGGTGAGCG-3'). Foram obtidas 22 cepas de S. marcescens, sendo 15 recuperadas de hemoculturas, e 7 de vigilância (swab retal); todas apresentaram resistência a: ampicilina, ampicilina-sulbactam, gentamicina e cefalotina. Não foi observada resistência a: ciprofloxacina, imipenem, meropenem e ertapenem. Quanto aos demais antibióticos avaliados, o perfil de suscetibilidade foi variável. Foram obtidos 11 padrões de amplificação por ERIC-PCR, dois foram compartilhados por 14 isolados. Foi possível observar um padrão polimórfico característico para as cepas provenientes de colonização gastrointestinal, exceto em dois casos, que apresentaram padrões genotípicos relacionadas a casos de sepse. Os dados obtidos neste trabalho confirmam o elevado índice de resistência da S. marcescens aos antimicrobianos; no entanto, todos os isolados apresentaram sensibilidade à ciprofloxacina e aos carbapenêmicos. A tipagem por meio de antibiograma e ERIC-PCR sugere dispersão de clones associados à colonização ou sepse entre alas na Unidade Neonatal do hospital estudado.
Palavras-chave: Serratia marcescens; Técnica de Tipagem Bacteriana; Reação em Cadeia da Polimerase; Resistência Microbiana a Medicamentos.
ABSTRACT
Serratia marcescens has been reported as an important agent of health care-related infections and has been highlighted for presenting a high level of intrinsic resistance to antimicrobials used in neonatology, besides persisting in hospital environments for long periods. In this work, S. marcescens was recovered from colonies in the gastrointestinal tract or late sepsis in newborn infants hospitalized in a Neonatal Unit in Belém. The identification of S. marcescens and the sensitivity test was carried out using a Vitek (BioMérieux) automated system; susceptibility to ertapenem was assessed using e-test strips (Oxoid). Genotyping was executed by ERIC-PCR using the primers ERIC1 (5'-TGAATCCCCAGGAGCTTACAT-3') and ERIC2 (5'-AAGTAAGTGACTGGGGTGAGCG-3'). Twenty-two strains of S. marcescens were recovered: 15 from hemocultures and 7 from surveillance (rectal swab culture). All presented resistance to ampicillin, ampicillin-sulbactam, gentamicin and cephalothin. There were no indications of resistance to ciprofloxacin, imipenem, meropenem or ertapenem. The susceptibility profiles varied for other antibiotics. Eleven amplification patterns by ERIC-PCR were obtained, and two were shared by 14 isolates. It was possible to observe a characteristic polymorphic pattern in the strains from gastrointestinal colonization, except for two cases, which presented genotypic patterns related to cases of sepsis. The data obtained in this work confirm the high level of resistance of S. marcescens against antimicrobials; however, all isolates displayed sensitivity to ciprofloxacin and carbapenemics. Antibiogram and ERIC-PCR typing suggest a dispersion of clones associated with colonization or sepsis among the wards of the Neonatal Unit in the surveyed hospital.
Keywords: Serratia marcescens; Bacterial Typing Techniques; Polymerase Chain Reaction; Bacterial Drug Resistance.
RESUMEN
La Serratia marcescens ha sido considerada como un agente importante de las infecciones relacionadas con la salud (IRAS, por sus siglas en portugués), y se ha destacado su alto nivel de resistencia intrínseca a los antimicrobianos utilizados en neonatología, además de persistir durante largos períodos de tiempo en el ambiente hospitalario. En este trabajo fueron evaluados a través de métodos fenotípicos y moleculares S. marcescens recuperados a partir de la colonización del tracto gastrointestinal o sepsis de aparición tardía en recién nacidos hospitalizados en la Unidad de Neonatología (UN) de Belém. La identificación de S. marcescens y las pruebas de sensibilidad se realizaron mediante el sistema automatizado Vitek (Biomerieux); la susceptibilidad al ertapenem se evaluó mediante la prueba de epsilometría (Oxoid). El genotipado se hizo mediante ERIC-PCR, utilizando partidores ERIC1 (5'-TGAATCCCCAGGAGCTTACAT-3') y ERIC2 (5'- AAGTAAGTGACTGGGGTGAGCG-3'). Se obtuvieron 22 cepas de S. marcescens, 15 recuperadas de hemocultivos y 7 de seguimiento (hisopado rectal); todas presentan resistencia a la ampicilina, ampicilina-sulbactam, gentamicina y cefazolina. No se presentó resistencia a la ciprofloxacina, imipenem, meropenem y ertapenem. En cuanto a los demás antibióticos evaluados, el perfil de susceptibilidad fue variable. Se obtubieron 11 patrones de amplificación por ERICPCR, dos de ellos compartidos por 14 aislamientos. Fue posible observar un patrón característico de las cepas polimórficas de la colonización gastrointestinal, excepto en dos casos que presentaron patrones de genotipos relacionados con los casos de sepsis. Los datos de este estudio confirman el alto nivel de resistencia de S. marcescens a los antibióticos, aunque todas las cepas fueron sensibles a ciprofloxacina y carbapenémicos. El tipaje a través de antibiograma y ERIC-PCR sugieren la dispersión de clones asociados con la colonización o la sepsis entre las salas de la Unidad de Neonatología del hospital estudiado.
Palabras clave: Serratia marcescens; Técnica de Tipificación Bacteriana; Reacción en Cadena de la Polimerasa; Farmacorresistencia Bacteriana.
INTRODUÇÃO
A Serratia marcescens, pertencente à família Enterobacteriaceae, tem sido relatada como importante agente de infecções relacionadas à saúde (IRAS), destacando-se pelo potencial de disseminação e com o agravante de apresentar elevado nível de resistência intrínseca às drogas usadas em neonatologia, e aos agentes antisséticos. Por colonizar a pele e o trato gastrointestinal de indivíduos adultos e neonatos, este patógeno persiste por longos períodos no ambiente hospitalar11,2,6. Portanto, após o relato de infecções, é necessário investigar a origem do patógeno e manter vigilância, por meio de tipagem, para o efetivo controle e/ou erradicação dos casos.
Vários métodos têm sido usados para tipagem de cepas epidêmicas de S. marcescens. Eles envolvem tanto caracterização fenotípica quanto genotípica, e são baseados no pressuposto de que organismos relacionados possuem características únicas, que os distinguem dos não-relacionados. As características fenotípicas (bioquímica, resistência aos antimicrobianos, sorotipagem, fagotipagem, etc) podem não ser tão discriminatórias, então o uso de métodos moleculares é sempre necessário para confirmação de clonalidade. As técnicas que utilizam eletroforese de gel em campo pulsado (PFGE) e ribotipagem apresentam boa discriminação, mas têm a desvantagem de serem trabalhosas, e necessitarem longo período de tempo para execução, além de equipamentos e reagentes caros9.
Técnicas moleculares baseadas na amplificação de ácidos nucleicos, como o Enterobacterial Repetitive Intergenic Consensus (ERIC), têm sido utilizadas pela facilidade de execução, reprodutibilidade, além da concordância com os resultados obtidos por ribotipagem5,2,12.
Neste trabalho são avaliadas cepas de S. marcescens, no tocante ao perfil de resistência aos antimicrobianos e à caracterização genética, provenientes de colonização ou sepse tardias ocorridas em Unidade Neonatal (UN) de referência em Belém.
MATERIAIS E MÉTODOS
CARACTERIZAÇÃO DA INSTITUIÇÃO E DAS AMOSTRAS
O estudo foi desenvolvido em UN com capacidade para 103 leitos, de hospital de alta complexidade localizado na Cidade de Belém, Pará. A unidade é constituída por Berçário Interno (para nascidos no próprio hospital); Unidade de Tratamento Intensivo (UTI) neonatal; e Berçário Externo (BE). O Berçário Interno, por sua vez, é dividido em cinco alas: Cuidados Especiais (CE), Cuidados Intermediários (CI), Semi-Intensiva (SI) e Sala de Transição (ST). A principal demanda de recém-nascidos, atendida pela unidade, é de prematuros de baixo peso.
Nos meses do estudo, foram realizadas 675 hemoculturas e 75 culturas de vigilância, para avaliar colonização do trato intestinal por cepas de S. marcescens.
COLHEITA E SELEÇÃO DAS AMOSTRAS
As hemoculturas foram realizadas a partir de 3 mL de sangue venoso de cada recém-nascido utilizando o sistema automatizado BACTEC 9120 (Becton Dickinson). A vigilância quanto à colonização do agente pelos neonatos foi realizada por cultivo de swab retal, obtido a partir do sétimo dia de internação dos neonatos, e repetido semanalmente até a alta hospitalar. As culturas primárias positivas foram repicadas em Ágar sangue (Difco), Ágar Cled (Difco) e Ágar Mac-Conckey (Difco) e incubadas em estufa bacteriológica (Fanen) a 37o C por 24 h. As colônias decorrentes de bacilos Gram-negativos não produtores de oxidase foram suspensas em solução salina 0,45% padronizada em colorímetro (BioMerieux) até a concentração equivalente ao tubo 0,5 da escala de Mac-Farland (1,5 106 UFC/ mL) e encubadas em cartões GN para identificação em sistema automatizado VITEK (BioMérieux). Foram selecionadas as espécies S. marcescens para este estudo.
A cepa de S. marcescens ATCC 8100 foi usada como controle na genotipagem, juntamente com uma amostra não relacionada ao surto.
TESTE DE SENSIBILIDADE
O perfil de suscetibilidade foi avaliado em sistema automatizado VITEC (BioMérieux) seguindo recomendação do fabricante. Foi avaliada a suscetibilidade da S. marcescens a: ampicilina (AM), ampicilina-sulbactam (SAM), amicacina (AN), aztreonan (ATM), cefepima (FEP), cefotaxima (CTX), cefoxitina (FOX), ceftazidima (CAZ), cefalotina (KF), ciprofloxacina (CIP), gentamicina (GN), piperacilina-tazobactam (PTZ), sulfatrimetoprima (STX), imipenem (IPM), meropenem (MEM). A sensibilidade ao ertapenem foi avaliada com auxílio de disco contendo 10 µg da droga (Oxoide). A leitura dos halos de inibição foi feita com auxílio de régua milimétrica.
GENOTIPAGEM DAS CEPAS POR ERIC-PCR
O DNA bacteriano foi extraído utilizando o método de fervura e congelamento (15 min cada) e submetido à amplificação pela técnica de reação em cadeia da polimerase (PCR), empregando os primers ERIC1 (5'- TGAATCCCCAGGAGCTTACAT-3') e ERIC2 (5'- AAGTAAGTGACTGGGGTGAGCG-3'), segundo o protocolo inicialmente descrito por Liu et al5. A análise do polimorfismo dos fragmentos de restrição foi feita após eletroforese em gel de agarose 2%, seguida da visualização em transiluminador de UV.
O desenvolvimento deste trabalho foi aprovado pelo Comitê de Ética em Pesquisa do Instituto Evandro Chagas, protocolo CEP/IEC no 003/07, em 14 de junho de 2007.
RESULTADOS
Foram obtidas 22 culturas de S. marcescens, recuperadas a partir de episódios de sepse e colonização neonatais tardias, sendo 15 provenientes de hemoculturas de neonatos e sete de cultura de vigilância. Todas as amostras apresentaram resistência a: ampicilina, ampicilina-sulbactam, gentamicina e cefalotina. Não foi observada resistência a: ciprofloxacina, imipenem, ertapenem, meropenem. Quanto aos demais antibióticos avaliados, o perfil de suscetibilidade foi variável (Quadro 1).
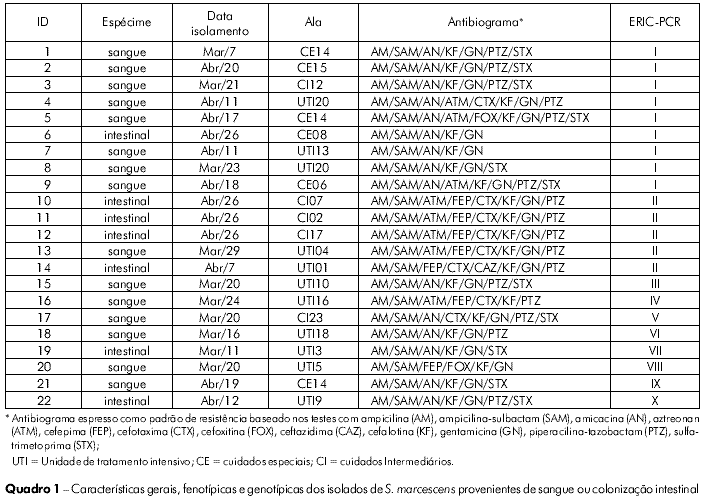
Foram obtidos 11 padrões de amplificação por ERIC-PCR, oito apresentaram padrões com cinco ou mais amplificações na faixa de 100 a 3.000 pb (Figura 1), enquanto quatro amostras apresentaram padrões com menos de cinco amplificações (padrões 17, 19, 21). Dois padrões polimórficos foram compartilhados por 14 isolados (Quadro 1, Figura 1).
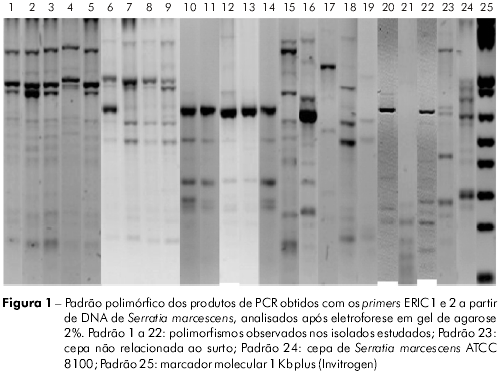
No grupo I, oito cepas foram provenientes de hemoculturas de neonatos internados em diferentes alas da UN, enquanto a cepa de S. marcescens designada pelo número 6 foi recuperada de colonização intestinal. No grupo II, as cepas 10, 11, 12 e 14 foram provenientes de swab retal (vigilância), enquanto a cepa 13 foi de hemocultura.
DISCUSSÃO
A Serratia marcescens pode estar associada a infecções esporádicas ou epidêmicas com o agravante de apresentar elevado nível de resistência aos antimicrobianos recomendados na terapêutica em neonatologia11,6 e aos agentes antisséticos utilizados, o que garante a persistência deste patógeno por longos períodos no ambiente hospitalar. Uma importante característica deste agente é a habilidade em produzir β-lactamases, que conferem resistência aos antimicrobianos β-lactâmicos de espectro estendido, complicando a terapia.
O esquema empírico de tratamento das IRAS em UN depende do tempo de aparecimento da infecção (precoce: aquela evidenciada nas primeiras 48 h de vida do recém-nascido; ou tardia: evidência clínica e laboratorial de infecção após as 48 h de vida do neonato); da realização prévia de procedimentos invasivos; do conhecimento da microbiota local; e dos perfis de resistência bacteriana em cada hospital1. Os protocolos podem ser definidos como formas estruturadas de suporte do manejo técnico, as quais incluem definição de objetivos terapêuticos, sequência temporal de cuidados, e estratégias diagnósticas e terapêuticas.
No presente estudo, todas as amostras de S. marcescens se apresentaram resistentes à ampicilina e à associação ampicilina/sulbactam. O índice de resistência aos aminoglicosídeos gentamicina e amicacina também foi elevado (Quadro 1). Martinez et al7, ao avaliarem 90 amostras de S. marcescens isoladas em 1994, no Hospital das Clínicas da Faculdade de Medicina de Ribeirão Preto, observaram 99% de resistência à ampicilina, 41% à gentamicina e 21% à amicacina.
Neste estudo, mais de 70% (16) das cepas de S. marcescens avaliadas apresentaram resistência ao β-lactâmico piperacilina associado ao inibidor de β-lactamase tazobactam. A associação tazobactam-piperacilina tem sido uma alternativa terapêutica para as IRAS causadas por bacilos Gram-negativos e anaeróbios produtores de β-lactamases em UTI, e mais recentemente em UTIN quando esses microrganismos são isolados em infecções tardias. Na UN avaliada, a associação tazobactam-piperacilina não deverá ser considerada para tratamento de infecções por S. marcescens.
Os agrupamentos observados após análise do polimorfismo obtido por ERIC-PCR para as cepas provenientes de pacientes internados em diferentes alas configuram disseminação delas entre as salas de CE, CI e UTI. De maneira geral, foi possível observar um padrão polimórfico característico para as cepas provenientes de colonização gastrointestinal; no entanto, em dois casos, cepas provenientes de colonização também estavam relacionadas a casos de sepse (Quadro 1). Não foram avaliados fatores de risco associados à septicemia.
Levando em consideração que não houve evolução do quadro infeccioso nos neonatos portadores das cepas 10, 11, 12 e 14, confirma-se a necessidade de uma associação de fatores de risco para o desenvolvimento de sepse, já que 80% deles não apresentaram quadro de septicemia.
A genotipagem baseada na amplificação de elementos repetidos, semelhante ao ERIC-PCR, tem sido usada com sucesso para caracterização de vários organismos, como o Mycobacterium tuberculosis10, Campylobacter8, Vibrio cholerae3, entre outros.
Foram avaliadas 22 amostras de S. marcescens, sendo sete recuperadas a partir de escarro de pacientes submetidos a ventilação mecânica durante surto de pneumonia, e 15 não relacionadas ao surto5. Os dados de tipagem por ERIC-PCR evidenciaram dois agrupamentos entre as cepas relacionadas ao surto e distintos perfis entre as cepas não-relacionadas. Tais dados foram confirmados por ribotipagem e o perfil de resistência aos antibióticos.
Neste estudo, foram observadas diferenças entre os dados de tipagem por ERIC-PCR e padrão de resistência aos antimicrobianos. Houve concordância entre os métodos de tipagem para as cepas 1, 2 e 3 (Grupo I); 6 e 7 (Grupo I); 10, 11, 12 e 13 (Grupo II), ou seja, em 64,29% das amostras avaliadas. Vários fatores podem justificar tal resultado. Primeiro: o padrão de suscetibilidade não foi determinado seguindo o método de discodifusão, padronizado e recomendado atualmente pelo Clinical and Laboratory Standards Institute (CLSI)4; tal técnica é importante por ser a mais completamente descrita, para a qual foram desenvolvidos padrões de interpretação apoiados por muitos dados laboratoriais e clínicos, cujas dificuldades e melhorias são constantemente discutidas. Segundo: o perfil de suscetibilidade bacteriana pode apresentar alterações em espaço de tempo inferior às mudanças nos padrões de ERIC-PCR. Os padrões polimórficos apresentados neste estudo mantiveram excelente reprodutibilidade, evidenciando boa discriminação entre cepas relacionadas ao surto e as não relacionadas.
Em todos os agrupamentos gerados por ERIC-PCR, o perfil de sensibilidade permitiu subdivisão das cepas, com maior variação de suscetibilidade aos antibióticos aztreonam, cefotaxima e cefoxitima; no entanto, tal achado fica restrito às cepas com alto índice de resistência.
As cepas de S. marcescens 8, 19 e 21 apresentaram o mesmo padrão de resistência, diferenciando-se quanto ao polimorfismo obtido por ERIC-PCR. A baixa resolução fenotípica pode ser decorrente do baixo índice de resistência encontrado para estas amostras, pois 100% das cepas apresentaram resistência aos antimicrobianos AM, SAM, NA, KF, GN. As cepas 8, 19 e 21 apresentaram também resistência à STX, o que foi observado em 50% da amostragem avaliada. Ou seja, a tipagem por caracterização do perfil de resistência pode ser um indicativo de transmissão; no entanto, outros métodos devem ser avaliados para sua confirmação.
Observa-se a necessidade de aumentar a amostragem de estudo para melhor avaliação da tipagem por análise de antibiograma e ERIC-PCR, além de desenvolver genotipagem secundária das amostras por PFGE, atualmente considerado "padrão ouro".
No caso do agrupamento das cepas provenientes de vigilância, a S. marcescens atua apenas como um indicador de transmissão. Assim como a S. marcescens, é possível que outros patógenos sejam transmitidos horizontal e concomitantemente, os quais podem ter passado despercebidos por não constituírem o foco da investigação.
Os dados obtidos neste trabalho confirmam o elevado índice de resistência das cepas de S. marcescens; no entanto, todas as cepas avaliadas apresentaram sensibilidade aos carbapenêmicos e à ciprofloxacina. A tipagem por meio de ERIC-PCR permitiu o agrupamento de cepas, sugerindo disseminação de clones associados à colonização ou sepse entre alas na UN do hospital estudado.
REFERÊNCIAS
1 Agência Nacional de Vigilância Sanitária (BR). Prevenção e controle de infecção hospitalar. Brasília: Ministério da Saúde; 2005.
2 Assadian O, Berger A, Aspöck C, Mustafa S, Kohlhauser C, Hirschl AM. Nosocomial outbreak of Serratia marcescens in a neonatal intensive care unit. Infect Control Hosp Epidemiol. 2002 Aug;23(8):457-6. DOI: 10.1086/502085 [ Links ]
3 Chokesajjawatee N, Zo YG, Colwell RR. Determination of clonality and relatedness of Vibrio cholerae isolates by genomic fingerprinting, using long-range repetitive element sequence-based PCR. Appl Environ Microbiol. 2008 Sep;74(17):5392-401. doi:10.1128/AEM.00151-08 [ Links ]
4 Clinical and Laboratory Standards Institute (USA). Performance standards for antimicrobial disk susceptibility tests; approved standard. 8th ed. Wayne, Pennsylvania; 2003. [ Links ]
5 Liu PYF, Lau YJ, Hu BS, Shir JM, Cheung MH, Shi ZY, et al. Use of PCR to study epidemiology of Serratia marcescens isolates in nosocomial infection. J Clin Microbiol. 1994;32(8):1935-8. [ Links ]
6 Maragakis LL, Winkler A, Tucker MG, Cosgrove SE, Ross T, Lawson E, et al. Outbreak of multidrug-resistant Serratia marcescens infection in a neonatal intensive care unit. Infect Control Hosp Epidemiol. 2008;29(5):418-23. DOI: 10.1086/587969 [ Links ]
7 Martinez R, Gironi RHAR, Santos VR. Sensibilidade bacteriana a antimicrobianos, usados na prática médica - Ribeirão Preto-SP - 1994. Medicina (Ribeirão Preto). 1996 abr-set;29(2-3):278-84. [ Links ]
8 Mouwen DJM, Weijtens MJBM, Capita R, Alonso-Calleja C, Prieto M. Discrimination of enterobacterial repetitive intergenic consensus PCR types of Campylobacter coli and Campylobacter jejuni by Fourier transform infrared spectroscopy. Appl Environ Microbiol. 2005 Aug;71(8):4318-24. DOI:10.1128/AEM.71.8.4318-4324.2005 [ Links ]
9 Parvaz P, Tille D, Meugnier H, Perraud M, Chevallier P, Ritter J, et al. A rapid and easy PCR-RFLP method for genotyping Serratia marcescens strains isolated in different hospital outbreaks and patient environments in the Lyon area, France. J Hosp Infect. 2002 Jun;51(2):96-105. [ Links ]
10 Sechi LA, Zanetti S, Dupré I, Delogu G, Fadda G. Enterobacterial repetitive intergenic consensus sequences as molecular targets for typing of Mycobacterium tuberculosis strains. J Clin Microbiol. 1998 Jan;36(1):128-32. [ Links ]
11 Villari P, Crispino M, Salvadori A, Scarcella A. Molecular epidemiology of an outbreak of Serratia marcescens in a neonatal intensive care unit. Infect Control Hosp Epidemiol. 2001 Oct;22(10):630-4. DOI: 10.1086/501834 [ Links ]
12 Vries JJ, Baas WH, Van der Ploeg K, Heesink A, Degener JE, Arends JP. Outbreak of Serratia marcescens colonization and infection traced to a healthcare worker with long-term carriage on the hands. Infect Control Hosp Epidemiol. 2006 Nov;27(11):1153-8. DOI: 10.1086/508818 [ Links ]
 Correspondência / Correspondence / Correspondencia:
Correspondência / Correspondence / Correspondencia:
Karla Valéria Batista Lima
Instituto Evandro Chagas,
Seção de Bacteriologia e Micologia
Rodovida BR 316, km 7, s/no, Levilândia
CEP: 67030-000
Ananindeua-Pará-Brasil
Recebido em / Received / Recibido en: 30/07/2009
Aceito em / Accepted / Aceito en: 01/10/2009










 texto em
texto em