Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Pan-Amazônica de Saúde
versión impresa ISSN 2176-6215versión On-line ISSN 2176-6223
Rev Pan-Amaz Saude v.1 n.1 Ananindeua mar. 2010
http://dx.doi.org/10.5123/S2176-62232010000100018
Caracterización molecular de rotavirus humano tipo G1 detectado en niños de Belém (Estado de Pará, Brasil)
Luana da Silva SoaresI; Joana D'Arc Pereira MascarenhasI; Yvone Benchimol GabbayI; Rosa Helena Porto GusmãoII; Alexandre da Costa LinharesI
ISeção de Virologia,
Instituto Evandro Chagas/SVS/MS, Ananindeua, Pará, Brazil
IICentro de Ciências Biológicas
e da Saúde, Universidade do Estado do Pará, Belém, Pará,
Brazil
Endereço para correspondência
Correspondence
Dirección para correspondencia
Título original: Molecular characterization of G1 human rotaviruses detected in children from Belém, Pará, Brazil. Traducido por: Rocio Tamara (resumen) y Lota Moncada (artículo)
RESUMEN
Los rotavirus son responsables por 40% de los casos de gastroenteritis infantil en el mundo, con más de 611 mil muertes al año, y el rotavirus tipo G1 representa su genotipo mas común en circulación. El objetivo de este trabajo fue realizar la caracterización molecular de las muestras de rotavirus de tipo G1 obtenido en niños que participaron en estudios realizados anteriormente en Belém, ciudad situada en el norte de Brasil, durante un período de 21 años (1982 a 2003). El rotavirus de tipo G1 fue detectado por electroforesis en gel de poliacrilamida, e inmunoensayo enzimático (IEE), y por la reacción en cadena de la polimerasa precedida por transcriptasa inversa para los genes VP7 y VP4. De las 798 especies positivas para rotavirus, 330 (41%) presentaron especificidad G1 por IEE, usando anticuerpos monoclonales. Se analizó mediante la reacción en cadena de la polimerasa precedida de trascripción inversa un total de 148 cepas de tipo G1. La caracterización de genotipos P y electroferotipos de rotavirus de tipo G1 se produjo en las frecuencias de 78% y 88%, respectivamente. Se identificaron tres variedades de electroferotipos largos, siendo el L1 predominante (79%). La combinación G1P [8] fue el más frecuente, representando el 64% de los casos. Las infecciones mixtas G1P[6]+P[8], G1P[4]+P[8], G1P[4]+P[6] y G1P[4]+P[6]+P[8] fueron encontradas en 11 (7%), 11 (7%), 3 (2%) y 1 (0,6%) de las muestras, respectivamente. Se identificó una muestra que presentaba una infección mixta G1+G4. Cabe señalar que este es el primer estudio que analiza la caracterización molecular del rotavirus tipo G1 en Brasil. Nuestros hallazgos harán posible una mejor comprensión de la diversidad molecular asociada a la infección por rotavirus de tipo G1 en nuestra región.
Palabras clave: Gastroenteritis; Infecciones por Rotavirus; Variación Genética.
INTRODUCCIÓN
El rotavirus del grupo A (RV-A) es el agente etiológico más común de la diarrea severa en el mundo, siendo responsable por 40% de las infecciones por gastroenteritis y resultando en 611 mil muertes de recién nacidos y niños, anualmente, particularmente en los países más pobres. El alcance global de esta enfermedad es bien reconocido - en verdad, todas los niños presentan un caso de gastroenteritis causada por rotavirus antes de los 5 años de edad7,24,25.
Este virus pertenece a la familia Reoviridae Reoviridae y se clasifica en siete grupos (A-G) y cuatro subgrupos (I, II, I + II, y no-I/II) de acuerdo a las especificidades de los epítopos de la proteína VP6 localizados en la cápside interna. El genoma del rotavirus contiene 11 segmentos de RNA de cadena doble (dsRNA) localizados en el core de la cápside de triple camada. La cápside externa está compuesta de dos proteínas estructurales, VP4 e VP7, que definen los genotipos P (proteína clivada por proteasas) y G (glicoproteína), respectivamente. Con base en la movilidad de los 11 segmentos en geles de poliacrilamida, los rotavirus pueden ser identificados como electroferotipos largos, cortos o súper cortos11.
Hasta hoy, al menos 23 rotavirus del tipo G y 30 del tipo P fueron identificados tomándose por base el análisis de la secuencia nucleotídica de los genes VP7 y VP4, respectivamente. Sin embargo, se sabe que pocos genotipos causan infección en humanos5,6,11,31,32. A pesar de un gran número de combinaciones G/P ser teóricamente posible, estudios epidemiológicos en todo el mundo han registrado la circulación de cinco tipos más comunes: G1P[8], G2P[4], G3P[8], G4P[8] y G9P[8]6,7,11,19,29.
El genotipo G1 es el más prevalente y ha sido detectado en frecuencias que varían entre 36% y 74% en diferentes regiones del mundo, siendo que las muestras G1P[8] representan aproximadamente 65% de todos los rotavirus identificados29. Castello et al6 identificaron especímenes G1P[8] en 40% de las infecciones por rotavirus en América Latina. Recientemente, Leite et al19 detectaron el tipo G1 en 43% de los casos durante el período pre-vacunación en Brasil; en contraste, luego de la introducción de la vacunación, la ocurrencia de este genotipo se redujo a 3% de las infecciones por rotavirus, debido a la gran predominancia de muestras G2P[4].
El análisis de la secuencia del gen VP7 de rotavirus G1 en Italia reveló la existencia de al menos, tres linajes genéticos. Estas variantes antigénicas pueden ser responsables por la circulación continua de rotavirus del tipo G12. Phan et al28 analizaron el gen VP7 de rotavirus del tipo G1 colectados en varias partes del mundo y sugirieron una nueva taxonomía, que incluye 11 linajes y 17 sublinajes.
El objetivo del presente estudio fue de realizar la caracterización molecular de las muestras de rotavirus del tipo G1 obtenidas de niños que participaron en varios estudios anteriormente llevados a cabo en Belém, norte de Brasil, por un período de 21 años (de 1982 a 2003).
MATERIALES Y MÉTODOS
PACIENTES Y ESPECÍMENES CLÍNICOS
Los especímenes obtenidos en cinco estudios sobre gastroenteritis viral realizados en Belém, Brasil, entre diciembre de 1982 y octubre de 2003, se analizaron con vistas a la detección de rotavirus1,15,20,21. De los 798 especímenes positivos para rotavirus por ensayo inmunoenzimático (EIA), 330 (41%) presentaban especificidad G1 cuando se utilizaron anticuerpos monoclonales. Para confirmar ese resultado, fueron analizadas 148 muestras de rotavirus del tipo G1 por medio de la reacción en cadena mediada por la polimerasa precedida de transcripción reversa (RT-PCR). Este análisis involucró todas las muestras de rotavirus G1 detectadas en los Estudios A, B, C y D, y 20% de los casos de Estudio E. Este estudio fue aprobado por el Comité de Ética del Instituto Evandro Chagas, Secretaría Nacional de Vigilancia en Salud.
EXTRACCIÓN DEL RNA
Rotavirus dsRNA was extracted from 10% fecal suspensión by using guanidinium isothiocyanate-silica nucleic acid extraction as described previously by Boom et al3.
ELECTROFORESIS EN GEL DE POLIACRILAMIDA (PAGE)
El perfil del RNA fue analizado por PAGE con tinción con nitrato de plata, conforme descrito anteriormente16,27.
CARACTERIZACIÓN DE LOS GENOTIPOS G YPDE ROTAVIRUS POR MEDIO DE REACCIÓN EN CADENA MEDIADA POR LA POLIMERASA PRECEDIDA POR TRANSCRIPCIÓN REVERSA (RT-PCR)
El genotipo G (VP7) fue determinado por transcripción reversa seguida por PCR-multiplex, como ya descrito en la literatura9,14,18. Dos diferentes grupos de iniciadores específicos se utilizaron para el genotipado: mezcla A, conteniendo los iniciadores específicos para G1 (9T1-1), G2 (9T1-2), G3 (9T-3P), G4 (9T-4) y G9 (9T-B); y mezcla B, conteniendo iniciadores específicos para los genotipos G5 (FT5), G6 (DT6), G8 (HT8) y G10 (ET10).
La determinación del genotipo P (VP4) fue realizada por medio de RT-PCR seguida de nested-PCR, conforme descrito por Gentsch et al12. En resumen, el gen completo de la proteína VP4 fue sometido a transcripción reversa, y un fragmento de 876 pares de base fue amplificado. Se utilizaron iniciadores específicos para los genotipos P[8] (1T-1), P[4] (2T-1), P[6] (3T-1) y P[9] (4T-1).
RESULTADOS
EPIDEMIOLOGÍA MOLECULAR DE LA INFECCIÓN POR ROTAVIRUS DEL TIPO G1
La mayor frecuencia de rotavirus del tipo G1 (68%, 232 de 343) se encontró durante una vigilancia realizada en 2003, en hospitales pediátricos, mientras que la menor (11%, 31 de 281) se verificó durante otra investigación involucrando niños hospitalizados, realizada entre 1998 y 2000 (Tabla 1). La edad promedio de los 148 niños infectados por G1 fue de 13 meses de edad (variando entre 2 meses y 3 años), de las los 70 (47%) tenían <1 año, 68 (46%) estaban en la franja entre 1-2 años y 10 (7%) tenían >2 años de edad.
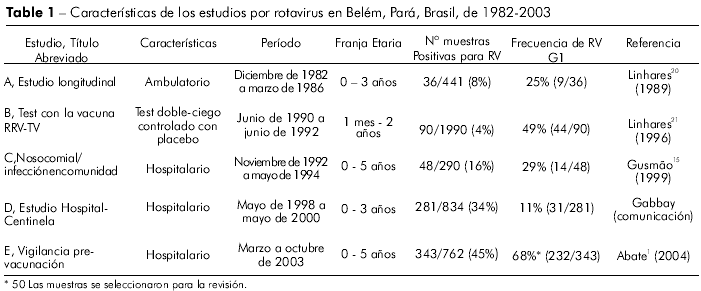
PAGE DE LAS MUESTRAS G1
Los perfiles de RNA fueron visualizados en 116 (78%) de las 148 muestras testeadas. Tres variedades de electroferotipos largos (L1, L2 y L3) fueron identificadas con base en las diferencias de migración de los segmentos genómicos 2, 5 y 10. Cuando se realiza la comparación del electroferotipo L1 con el L2, el segmento 2 de la variedad L2 migró más lentamente que su correlato L1. En relación a L3, se observaron diferencias de migración entre los segmentos 5 y 10, en comparación a L1. Un segmento extra fue encontrado en una muestra presentando el perfil L1 (Figura 1). El electroferotipo más frecuente fue L1 (79%, 92 de 116). El Estudio E presentó el mayor número de muestras caracterizadas por PAGE (96%, 48 de 50).
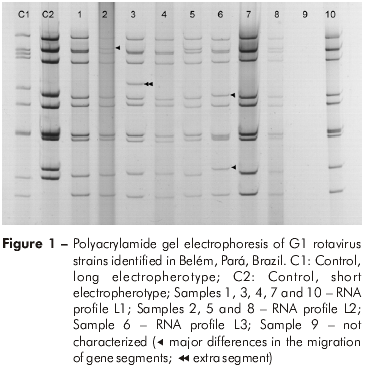
GENOTIPADO DE LOS TIPOS G Y P DE ROTAVIRUS
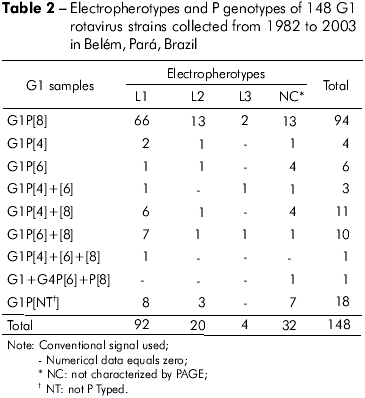
Fueron detectadas 147 muestras conteniendo especificidad para el tipo G1 y una muestra presentando una infección mixta G1+G4. El genotipo P puede ser determinado en 130 (88%) muestras. El tipo P[8] fue lo más frecuente (64% de los casos). Además de eso, las infecciones mixtas P[6] + P[8], P[4] + P[8], P[4] + P[6] e P[4] + P[6] + P[8] fueron encontradas en 11 (7%), 11 (7%), 3 (2%) e 1 (0.6%) muestras, respectivamente. La Tabla 2 presenta la caracterización de 148 muestras por PAGE y RT-PCR (genes VP4 y VP7). La combinación G1P[8], que presenta el estándar L1, fue responsable por 45% de las infecciones.
DISCUSIÓN
El rotavirus del tipo G1 todavía aparenta ser el genotipo más prevalente, representando cerca de mitad de las muestras circulantes en todo el mundo29. En países desarrollados, el genotipo G1 presenta las frecuencias más altas (70%-73%), mientras que en países en desarrollo, esas tasas parecen ser menores (36%-57%). Esa diferencia puede ser atribuida a la circulación más frecuente de muestras incomunes, así como a la emergencia de nuevos genotipos en los países más pobres13,29. En este estudio, el tipo G1 fue detectado en 41% de los casos de rotavirosis durante los 21 años de investigación usando diferentes abordajes en las poblaciones estudiadas. Los resultados aquí presentados son semejantes a los de estudios realizados en Rio de Janeiro, en donde 50% de las muestras obtenidas de niños con diarrea aguda eran del tipo G133. Parra et al26 identificaron el tipo G1 en 17% de los casos en una investigación realizada en el Paraguay, entre 1998 y 2000. Esta diferencia puede estar relacionada a la alta prevalencia del G4 y a la detección del G9 en aquel estudio.
Las tasas de rotavirus G1 variaron entre 11% y 68% en las investigaciones analizadas. Las menores tasas se observaron en el período entre 1998 y 2000. Estos resultados se relacionan, probablemente, a la alta frecuencia de especímenes no tipados, bien como a la emergencia del G9 durante este período. Santos et al30 registraron que 79% de todas las muestras analizadas en la ciudad de Salvador eran del tipo G9. Resultados semejantes también fueron observados en Goiás, en donde Costa et al8, detectaron 34% de infecciones por rotavirus del tipo G9. Estos descubrimientos sugieren que la emergencia del tipo G9 en Brasil fue seguida por una amplia circulación de muestras presentando este genotipo.
Se destaca que, en este estudio, todas las muestras G1 presentaron electroferotipos largos. Estos perfiles de RNA presentaron tres diferentes estándares, lo que demuestra la existencia de una gran diversidad de electroferotipos en la región. Todas las variedades fueron detectadas en los especímenes de los Estudios B y D, que, por su vez, presentaron una alta frecuencia de muestras incomunes y no tipadas (datos no presentados). Luz et al22 detectaron dos variedades de electroferotipos largos, y 19 % de los especímenes del tipo G1 presentaron perfil corto, con tres variedades circulando entre niños con diarrea en el Estado de Maranhão.
El G1P[8] ya fue considerado en diversos países como el genotipo circulante predominante29. En el presente estudio, este genotipo fue detectado en 64% de las muestras. Datos semejantes fueron obtenidos por Gentsch et al13 y Castello et al6 en investigaciones realizadas en América Latina. Esos estudios registraron la ocurrencia de esta combinación en 52% y 40% de las infecciones por rotavirus, respectivamente. En Brasil, Carmona et al4 detectaron el G1P[8] en 67% de los especímenes colectados en un período de ocho años en São Paulo.
En nuestro análisis encontramos una infección mixta involucrando a los genotipos G1+G4. Mascarenhas et al23 analizaron muestras fecales de un niño recién nacido hospitalizado con diarrea leve/moderada adquirida en comunidad y observaron la ocurrencia de mezcla G1+G4. Kebaabetswe et al17, en una investigación realizada en Botsuana abarcando niños con gastroenteritis, detectaron las infecciones mixtas G1+G2, G1+G8 y G1+G3+G9 en 6%, 22% y 6% de los casos, respectivamente.
Es importante resaltar la frecuencia de infecciones mixtas involucrando a los rotavirus del tipo G1 (17% de los casos). Das et al10 documentaron 30% de los casos con infecciones mixtas en una investigación con niños hospitalizados en India, a pesar de otros tipos G haber sido descritos. Las mayores tasas de infecciones mixtas fueron detectadas principalmente en países en desarrollo, probablemente por la ocurrencia de rearreglos genéticos de rotavirus, lo que puede llevar a la emergencia de nuevos tipos G y P de rotavirus circulantes, bien como al escape de especímenes mutantes.
Por lo que sabemos, este es el primer estudio a abordar la caracterización molecular de rotavirus del tipo G1 en Brasil. Nuestros hallazgos suministran datos que permitirán una mejor comprensión a respecto de la diversidad molecular asociada a las infecciones por este tipo de rotavirus antes del advenimiento de la vacuna. Abordajes más específicos abarcando la secuenciación de nucleótidos pueden ser aplicados para evaluar la circulación de variedades genéticas del tipo G1 y su impacto sobre estrategias de vacunación.
REFERENCES
1 Abate H, Linhares AC, Venegas G, Vergara R, Lopez P, Jimenez E, et al. Results of a hospital-based study on rotavirus gastroenteritis in Latin American children [abstract]. In: 24th International Congress of Pediatrics (ICP); 2004 Aug 15-20; Cancun, Mexico; 2004. p. 656.
2 Arista S, Giammanco GM, De Grazia S, Ramirez S, Lo Biundo C, Colomba C, et al. Heterogeneity and temporal dynamics of evolution of G1 human rotaviruses in a settled population. J Virol. 2006 Nov;80(21):10724-33. DOI:10.1128/JVI.00340-06 [ Links ]
3 Boom R, Sol CJ, Salimans MM, Jansen CL, Wertheimvan Dillen PM, Noordaa J. Rapid and simple method for purification of nucleic acids. J Clin Microbiol. 1990 Mar;28(3):495-503. [ Links ]
4 Carmona RC, Timenetsky MC, Morillo SG, Richtzenhain LJ. Human rotavirus serotype G9, São Paulo, Brazil, 1996-2003. Emerg Infect Dis. 2006 Jun;12(6):963-8. [ Links ]
5 Castello AA, Argüelles MH, Rota RP, Olthoff A, Jiang B, Glass RI, et al. Molecular epidemiology of group A rotavirus diarrhea among children in Buenos Aires, Argentina, from 1999 to 2003 and emergence of the infrequent genotype G12. J Clin Microbiol. 2006 Jun;44(6):2046-50. DOI: 10.1128/JCM.02436-05 [ Links ]
6 Castello AA, Arvay ML, Glass RI, Gentsch J. Rotavirus strain surveillance in Latin America: a review of the last nine years. Pediatr Infect Dis J. 2004 Oct;23(10 Suppl):S168-72. [ Links ]
7 Centers for Disease Control and Prevention. Rotavirus surveillance—worldwide, 2001-2008. MMWR Morb Mortal Wkly Rep. 2008 Nov;57(46):1255-7. [ Links ]
8 Costa PS, Cardoso DD, Grisi SJ, Silva PA, Fiaccadori F, Souza MB, et al. Rotavirus A infections and reinfections: genotyping and vaccine implications. J Pediatr (Rio J). 2004 Mar-Apr;80(2):119-22. [ Links ]
9 Das BK, Gentsch JR, Cicirello HG, Woods PA, Gupta A, Ramachandran M, et al. Characterization of rotavirus strains from newborns in New Delhi, India. J Clin Microbiol. 1994 Jul;32(7):1820-2. [ Links ]
10 Das S, Varghese V, Chaudhuri S, Barman P, Kojima K, Dutta P, et al. Genetic variability of human rotavirus strains isolated from Eastern and Northern India. J Med Virol. 2004 Jan;72(1):156-61. [ Links ]
11 Estes MK, Kapikian AZ. Rotaviruses. In: Knipe DM, Howley PM, editors. Fields virology. 5th ed. Philadelphia: Lippincott Williams and Wilkins; 2007. p. 1917-74.
12 Gentsch JR, Glass RI, Woods P, Gouvea V, Gorziglia M, Flores J, et al. Identification of group A rotavirus gene 4 types by polymerase chain reaction. J Clin Microbiol. 1992 Jun;30(6):1365-73. [ Links ]
13 Gentsch JR, Laird AR, Bielfelt B, Griffin DD, Banyai K, Ramachandran M, et al. Serotype diversity and reassortment between human and animal rotavirus strains: implications for rotavirus vaccine programs. J Infect Dis. 2005 Sep;192 Suppl 1:S146-59. [ Links ]
14 Gouvea V, Santos N, Timenetsky MC. Identification of bovine and porcine rotavirus G types by PCR. J Clin Microbiol. 1994 May;32(5):1338-40. [ Links ]
15 Gusmão RH, Mascarenhas JD, Gabbay YB, Lins-Lainson Z, Ramos FL, Monteiro TA, et al. Rotavirus subgroups, G serotypes, and electrophoretypes in cases of nosocomial infantile diarrhoea in Belém, Brazil. J Trop Pediatr. 1999 Apr;45(2):81-6. DOI:10.1093/tropej/45.2.81 [ Links ]
16 Herring AJ, Inglis NF, Ojeh CK, Snodgrass DR, Menzies JD. Rapid diagnosis of rotavirus infection by direct detection of viral nucleic acid in silver-stained polyacrylamide gels. J Clin Microbiol. 1982 Sep;16(3):473-7. [ Links ]
17 Kebaabetswe LP, Sebunya TK, Matsheka MI, Ndung’u T. Detection and molecular characterisation of group a rotavirus from children in northern Botswana. East Afr Med J. 2005 Apr;82(4):203-8. [ Links ]
18 Leite JP, Alfieri AA, Woods PA, Glass RI, Gentsch JR. Rotavirus G and P types circulating in Brazil: characterization by RT-PCR, probe hybridization, and sequence analysis. Arch Virol. 1996;141(12):2365-74. [ Links ]
19 Leite JP, Carvalho-Costa FA, Linhares AC. Group A rotavirus genotypes and the ongoing Brazilian experience: A review. Mem Inst Oswaldo Cruz. 2008 Dec;103(8):745-53. DOI:10.1590/S0074-02762008000800001 [ Links ]
20 Linhares AC, Gabbay YB, Freitas RB, Travassos da Rosa ES, Mascarenhas JD, Loureiro ECB. Longitudinal study of rotavirus infections among children from Belém, Brazil. Epidemiol Infect. 1989 Feb;102(1):129-45. [ Links ]
21 Linhares AC, Gabbay YB, Mascarenhas JD, Freitas RB, Oliveira CS, Bellesi N, et al. Immunogenicity, safety and efficacy of tetravalent rhesus-human, reassortant rotavirus vaccine in Belém, Brazil. Bull World Health Organ. 1996;74(5):491-50. [ Links ]
22 Luz CR, Mascarenhas JD, Gabbay YB, Motta AR, Lima TV, Soares LS, et al. Rotavirus serotypes and electropherotypes identified among hospitalized children in São Luís, Maranhão, Brazil. Rev Inst Med Trop Sao Paulo. 2005 Sep-Oct;47(5):287-93. DOI:10.1590/S0036-46652005000500009 [ Links ]
23 Mascarenhas JD, Leite JP, Lima JC, Heinemann MB, Oliveira DS, Araújo IT, et al. Detection of a neonatal human rotavirus strain with VP4 and NSP4 genes of porcine origin. J Med Microbiol. 2007 Apr;56(Pt 4):524-32. DOI:10.1099/jmm.0.46635-0 [ Links ]
24 Parashar UD, Gibson CJ, Bresse JS, Glass RI. Rotavirus and severe childhood diarrhea. Emerg Infect Dis. 2006 Feb;12(2):304-6. [ Links ]
25 Parashar UD, Hummelman EG, Bresee JS, Miller MA, Glass RI. Global illness and deaths caused by rotavirus disease in children. Emerg Infect Dis. 2003 May;9(5):565-72. [ Links ]
26 Parra GI, Bok K, Martínez V, Russomando G, Gómez J. Molecular characterization and genetic variation of the VP7 gene of human rotaviruses isolated in Paraguay. J Med Virol. 2005 Dec;77(4):579-86. DOI:10.1002/jmv.20495 [ Links ]
27 Pereira HG, Azeredo RS, Leite JP, Barth OM, Sutmoller F, Farias V, et al. Comparison of polyacrylamide gel electrophoresis (PAGE), immuno-electron microscopy (IEM) and enzyme immunoassay (EIA) for the rapid diagnosis of rotavirus infection in children. Mem Inst Oswaldo Cruz. 1983 Oct-Dec;78(4):483-90. DOI:10.1590/S0074-02761983000400012 [ Links ]
28 Phan TG, Okitsu S, Maneekarn N, Ushijima H. Evidence of intragenic recombination in G1 rotavirus VP7 genes. J Virol. 2007 Sep;81(18):10188-94. DOI:10.1128/JVI.00337-07 [ Links ]
29 Santos N, Hoshino Y. Global distribution of rotavirus serotypes/genotypes and its implication for the development and implementation of an effective rotavirus vaccine. Rev Med Virol. 2005 Jan-Feb;15(1):29-56. [ Links ]
30 Santos N, Volotão EM, Soares CC, Campos GS, Sardi SI, Hoshino Y. Predominance of rotavirus genotype G9 during the 1999, 2000, and 2002 seasons among hospitalized children in the city of Salvador, Bahia, Brazil: implications for future vaccine strategies. J Clin Microbiol. 2005 Aug;43(8):4064-9. DOI:10.1128/JCM.43.8.4064-4069.2005 [ Links ]
31 Trojnar E, Otto P, Johne R. The first complete genome sequence of a chicken group A rotavirus indicates independent evolution of mammalian and avian strains. Virology. 2009 Apr;386(2):325-33. DOI:10.1016/j.virol.2009.01.034 [ Links ]
32 Ursu K, Kisfali P, Rigó D, Ivanics E, Erdélyi K, Dán A, et al. Molecular analysis of the VP7 gene of pheasant rotaviruses identifies a new genotype, designated G23. Arch Virol. 2009;154(8):1365-9. DOI:10.1007/s00705-009-0439-0 [ Links ]
33 Volotão EM, Soares CC, Maranhão AG, Rocha LN, Hoshino Y, Santos N. Rotavirus surveillance in the city of Rio de Janeiro-Brazil during 2000-2004: detection of unusual strains with G8P[4] or G10P[9] specificities. J Med Virol. 2006 Feb;78(2):263-72. DOI:10.1002/jmv.20535 [ Links ]
 Correspondência
/ Correspondence / Correspondencia:
Correspondência
/ Correspondence / Correspondencia:
Luana da Silva Soares
Instituto Evandro Chagas
Rodovia BR316, km 7, s/no, Levilândia
CEP: 67030-000
Ananindeua-Pará-Brasil
Fax + 00 55 91 32142006
E-mail:luanasoares@iec.pa.gov.br
Recebido em / Received / Recibido en: 31/7/2009
Aceito em / Accepted / Aceito en: 25/9/2009










 texto en
texto en 
 Curriculum ScienTI
Curriculum ScienTI