Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Pan-Amazônica de Saúde
versión impresa ISSN 2176-6215versión On-line ISSN 2176-6223
Rev Pan-Amaz Saude v.1 n.2 Ananindeua jun. 2010
http://dx.doi.org/10.5123/S2176-62232010000200005
ARTÍCULO ORIGINAL
Comparación entre dos métodos de obtención de DNA a ser usados como protocolos alternativos para la detección de parásitos humanos causadores de malaria por nested PCR
Giselle Maria Rachid Viana; Danielle Regina Lima Barbosa; Ediclei Lima do Carmo; José Mário Veloso Peres; José Maria Souza Nascimento; Marinete Marins Póvoa
Laboratório de Pesquisas Básicas em Malária, Seção de Parasitologia, Instituto Evandro Chagas/SVS/MS, Ananindeua, Pará, Brasil
Endereço para correspondência
Correspondence
Dirección para correspondencia
Título original: Comparison of two DNA obtainment methods as alternative protocols for the detection of human malária parasites by nested PCR. Traducido por: Lota Moncada
RESUMEN
Un diagnóstico de laboratorio correcto y preciso de malaria humana todavia se considera un desafio, pues el método de referencia, el de la gota espesa con colocación en Giemsa, presenta limitaciones que dificultan el control de la malaria. Debido a esos problemas, varias investigaciones han tenido como objetivo desarrollar métodos alternativos para el diagnóstico de la malaria. Gran parte de esos estudios aborda métodos de diagnóstico molecular, que han resultado en el desarrollo de algunas alternativas al método de tenido por Giemsa. Sin embargo, esos métodos, por su vez, presentan limitaciones, que incluyen su alto costo, la complejidad del protocolo y la variación de la calidad de las fuentes de DNA y de reactivos. En este aspecto, la técnica de nested PCR ha demostrado ser un buen método y puede ser mejorado usando una fuente de DNA de alta calidad. En este estudio se evaluaron dos métodos para la obtención de DNA de muestras de sangre seca recogidas en papel de filtro: 1) lavado y 2) saponina/Chelex-100. El segundo método presentó sensibilidad y especificidad más altas en relación al primero, pues detectó más infecciones, tanto simples como mixtas, bien como infecciones por Plasmodium malariae. Con base en estos resultados, presentamos el segundo método como el protocolo de elección para la obtención de DNA. La técnica de nested PCR usando saponina/Chelex-100 para extracción de DNA puede ser un método alternativo o complementario de diagnóstico de parásitos de la malaria humana, pero no se considera adecuado para uso de rutina.
Palabras clave: Malaria; ADN; Reacción en Cadena de la Polimerasa.
INTRODUCCIÓN
La malaria es uno de los principales problemas de salud pública en Brasil y alcanza especialmente a las poblaciones pobres en la Región Amazónica16,18. La identificación precisa de las especies de Plasmodium que infectan a humanos es de extrema importancia para que su tratamiento sea viable de forma rápida y adecuada. El método de referencia para el diagnóstico de la malaria es el frotis de gota espesa con tinción de Giemsa (TS); presenta bajo costo, buena sensibilidad, permite la identificación de las formas de las especies de Plasmodium en todas las fases de desarrollo y la cuantificación de los estadios del parásito8,21. Generalmente, su sensibilidad para la detección por el microscopista varía entre 10-30 parásitos µL de sangre; sin embargo, esta sensibilidad puede ser limitada cuando hay una gran cantidad de muestras a ser examinadas en un intervalo de tiempo muy breve. Es posible obtener resultados falsos-negativos debido a los bajos niveles de parasitemia, a la existencia de infecciones mixtas o inexperiencia por parte del microscopista. Este hecho puede comprometer una de las principales estrategias de control de la malaria: un diagnóstico rápido y preciso, que haga viable la realización del tratamiento adecuado2,3,7,20.
Para suplantar algunas limitaciones del método TS, algunos métodos basados en la reacción en cadena de la polimerasa (PCR), como el protocolo nested PCR desarrollado por Kimura et al11, han sido utilizados para detectar e identificar parásitos de la malaria. Esos métodos son más sensibles y específicos que el TS y pueden detectar incluso un único parásito por microtítulo de sangre5,10,21,23. Sin embargo' el éxito de una técnica de PCR depende de varios factores' tales como la calidad de los reactivos y del ADN obtenido de diferentes fuentes. Además' ya se ha demostrado que la amplificación del ADN de muestras de sangre seca en papel filtro es una fuente confiable de ADN cuando las condiciones de transporte' de manipulación y almacenamiento se consideran adecuadas para evitar la contaminación o la degradación del ADN1,4,6,12. Por este motivo' es importante probar distintos métodos de obtener ADN' como protocolos alternativos de detección de parásitos da malaria humana por nested PCR. Este estudio tuvo como objetivo comparar las dos técnicas siguientes: 1) Extracción de ADN por procedimiento de lavados26, y 2) Extracción de ADN por medio de tratamiento de muestras de sangre seca con saponina/Chelex-100 sobre papel filtro27.
MATERIALES Y MÉTODOS
Utilizamos en este estudio' 75 muestras de sangre seca positivas (parasitemia variando de 0'001% a 2%) y 78 negativas sobre discos de papel filtro Whatman®' de 2'5 cm. de diámetro (Titertek' ICN Biomedicals - England). Las muestras de sangre se colectaron de habitantes de los municipios de Novo Repartimento (04o 14' 55'' N; 50o 07' 25'' W), Parauapebas (06o 04' 03" N; 49o 54' 08" W), Tucuruí (03o 45' 03" N; 49o 40' 03" W) y Belém (01o 27' 20" N; 48o 30' 15" W)' en el Estado de Pará' Brasil. Los controles para cada parásito de malaria humana incluyeron muestras positivas bien caracterizadas y agua ultra pura y ADN humano para los controles negativos. Todas las muestras fueron probadas y diagnosticadas según el método de frotis de gota espesa con tinción de Giemsa (TS). La cuantificación de los parásitos se realizó en las láminas con muestras positivas14.
ADN OBTENIDO POR EL MÉTODO DE LAVADO
Las muestras de papel filtro fueron preparadas poniendo cinco gotas de 20 µL de cada muestra sobre discos de papel filtro Whatman® (2'5 cm de diámetro) (Titertek' ICN Biomedicals - England) y secándolas a temperatura ambiente (TA, 25-27o C). Las muestras fueron almacenadas en embalajes plásticos previamente identificados (BHL Limited' Poole -England)' que habían sido mantenidas a una temperatura de -20o C hasta su utilización. Fueron lavadas con agua destilada y solución salina 0,9%, utilizando un sistema de filtrado Millipore® para remover todos los inhibidores de PCR26.
EXTRACCIÓN DE ADN POR EL MÉTODO DE SAPONINA/CHELEX-100
Un cuarto de cada gota seca sobre papel filtro se cortó en pequeños trozos e incubado en hielo con una solución de saponina 0,5% en PBS por 1 h. Posteriormente, la solución fue mezclada usando un "vórtex" y el sobrenadante fue descartado. Se hizo entonces, un lavado con PBS, adicionado 20% de una solución de chelex-100 en agua y la solución fue incubada en baño seco por 1 h. Después de la incubación, el sobrenadante se transfirió a tubos Eppendorf® estériles y almacenado a -20o C27.
REACCIÓN DE NESTED PCR
La reacción de amplificación de la subunidad del gen 18S del ARN ribosómico (rRNA) del Plasmodium (ssu-rRNA) se realizó de acuerdo a lo descrito en el protocolo de Kimura et al11. Los primers P1 y P2 fueron utilizados como primers universales en el primer ciclo, y los primers F2, M1 y V1 se utilizaron como primers específicos para P. falciparum, P. malariae y P. vivax, respectivamente, en el segundo ciclo. Los productos obtenidos por nested PCR se fraccionaron por medio de electroforesis en gel de agarosa 2% (Ultra pure agarose, BRL 155517-014) a 100 V por 1 h y teñida con bromuro de etidio (5 µL/mL). Los tamaños esperados de los productos de la PCR eran de 130 bp para el primer estadio y 100-110 bp para el segundo estadio. Los técnicos que realizaron la nested PCR no tuvieron ciencia de los resultados obtenidos por el análisis por microscopia.
ÉTICA
El protocolo de esta investigación fue aprobado por el Comité de Ética en Investigación del Instituto Evandro Chagas (004/2003-CEP/IEC/SVS/MS). Los objetivos y procedimientos del estudio fueron debidamente explicados a todos los participantes; todos los pacientes firmaron un término de consentimiento libre y esclarecido y también fueron colectados datos sociodemográficos y clínicos.
RESULTADOS
De las 153 muestras' 49'01% (75/153) fueron positivas por TS (con parasitemia variando de 0'001% a 2'0%) y 50'98% (78/153) fueron negativas. Por nested PCR' 43'79% (67/153) de las muestras fueron positivas y 56'20% (86/153) fueron negativas con el método de extracción del ADN por el procedimiento de lavado' mientras que 51'63% (79/153) fueron positivas y 48'36% (74/153) fueron negativas con el método por saponina/chelex-100 (Tabla 1).

El desempeño de nested PCR comparada con el TS fue el siguiente: 1) método de lavado - sensibilidad = 89,33%, especificidad = 100%, precisión = 94,77%; 2) saponina/chelex-100 - sensibilidad = 100%, especificidad = 94,87%, precisión = 97,39%. El acuerdo entre esos métodos y el análisis al microscopio fue casi perfecto [Kappa = 0,8952 y 0,9477, respectivamente; IC 95%: variación de 0,80 a 1,00]. El método de saponina/chelex-100 ofrece muestras de ADN de mejor calidad que el método de lavado.
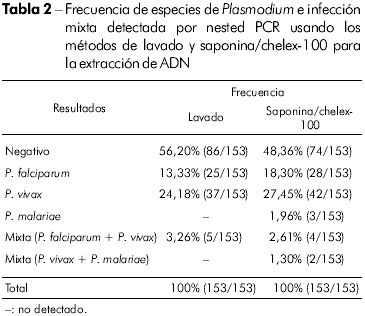
La frecuencia de especies de Plasmodium y de infecciones mixtas detectadas por Nested-PCR usando el ADN extraído por los métodos de lavado y saponina/chelex-100 está demostrada en la tabla 2. El método de saponina/chelex-100 suministró mejores muestras de ADN, lo que queda demostrado por el hecho de que, con este método, nested PCR pudo detectar tanto infecciones simples como mixtas por P. malariae, y detectó un mayor número de otras infecciones que el método de lavado.
La figura 1 presenta los resultados del análisis por nested PCR usando ADN obtenido por el método de saponina/chelex-100. Las franjas 2, 3 y 4 contienen muestras de un único paciente; cada pozo corresponde a reacciones para P. falciparum, P. malariae y P. vivax, respectivamente, demostrando que el paciente presenta una infección mixta por P. malariae y P. vivax.

DISCUSIÓN
A pesar de que el TS aún es el método de referencia para el diagnóstico de malaria, las técnicas moleculares son más sensibles y específicas para detectar parásitos y pueden ser utilizadas para evaluar el desempeño de los análisis por microscopia20,28.
Las muestras de sangre seca sobre papel filtro constituyen un método simple y accesible para colecta y almacenamiento. Han sido ampliamente utilizados en selección para diagnóstico, análisis genético y estudios sobre epidemiología molecular en áreas remotas con clima tropical, donde las condiciones de transporte y almacenamiento no son ideales. La pérdida de sensibilidad de métodos basados en PCR debido a las condiciones de campo ya ha sido informada y es, probablemente, causada por la baja pureza, estabilidad e integridad del ADN obtenido de muestras de sangre sobre papel filtro. Un problema observado con la colecta sobre papel filtro es la limitación den el volumen de las muestras que pueden ser utilizadas para la extracción de ADN. Por lo tanto, un buen aislamiento del ADN es fundamental para asegurar la calidad y la eficacia de métodos moleculares7,20.
Varios estudios tuvieron el objetivo de reducir los costos de protocolos basados en PCR, bien como volverlos rápidos y precisos. Un factor esencial del protocolo para la adquisición de buenos resultados es obtener un muestra de ADN de buena calidad1,5,6,7.
En este estudio, realizamos un análisis comparativo de dos protocolos para la obtención de ADN, llevando en consideración el tiempo necesario, sus costos y la calidad del ADN extraído7. Los resultados demostraron que la técnica de nested PCR con procedimiento de lavado no detectó ocho muestras positivas (seis con P. vivax y dos con P. falciparum) identificadas por TS, mientras que el método de nested PCR con saponina/chelex-100 detectó todas las muestras positivas identificadas por TS, bien como otras cinco muestras (tres infecciones simples por P. malariae y dos infecciones mixtas por P. vivax y P. malariae). Un motivo posible para la falla en la detección de algunas muestras positivas se debe al hecho que el método de lavado no eliminó inhibidores de nested PCR adecuadamente, como la hemoglobina. Los resultados demostraron que el método de nested PCR con saponina/chelex-100 se mostró más sensible y específico que el de lavado, especialmente para la detección de P. malariae.
Análisis de la extracción de ADN por lavado con saponina/chelex-100, como descrito por Warhurst et al26 y Wooden et al27, respectivamente, de 153 muestras de sangre seca sobre papel filtro demostraron que ambos métodos son sensibles y presentan alta especificidad, además de ser de fácil y rápida realización. Además, por el hecho de que esos métodos constituyen protocolos in house, sus costos fueron más bajos que los de los kits comerciales disponible. Sin embargo, el método con saponina/chelex-100 fue considerado más sensible y, consecuentemente, debería ser el protocolo de elección para la detección de parásitos de malaria basado en la extracción de ADN por nested PCR.
El protocolo de nested PCR desarrollado por Kimura et al11 para especies de Plasmodium que infectan a humanos demostró ser útil, ya que es bastante sensible, específico, pasible de ser reproducido y no presenta evidencias de contaminación cruzada en el estudio. Utilizando este protocolo, detectamos infecciones mixtas que habían sido identificadas por TS como infecciones simples, lo que corrobora los datos presentados por Ebrahimzadeh et al5, Scopel et al21, Singh et al22, Snounou et al24 y Toma et al25.
En casos de infección mixta, predomina una especie de Plasmodium, que normalmente es la detectada por ambas técnicas, TS y PCR, mientras que la especie no dominante es identificada apenas por métodos basados en análisis de PCR. Además, es frecuente un bajo nivel de parasitemia de P. malariae principalmente en infecciones mixtas, probablemente por causa de las características de esta especie y del mecanismo de regulado dependiente de la densidad. Por eso, esta especie podría pasar fácilmente sin ser notada en análisis de microscopio cuando P. falciparum o P. vivax son las especies dominantes. Este hecho también ayuda a explicar el mayor número de infecciones mixtas detectadas a través de métodos de biología molecular, cuando comparado a las herramientas de microscopia tradicional27.
Los métodos de diagnóstico de malaria basados en PCR son bastante específicos y sensibles; sin embargo, no pueden sustituir los análisis convencionales por microscopia para uso de rutina9, pues no permiten la cuantificación de las formas de los parásitos. Además, son técnicas complejas, con varias etapas y alto costo. Por lo tanto, pueden ser útiles como herramientas alternativas y complementarias para el diagnóstico de malaria humana en países en desarrollo4,13,19,22.
CONCLUSIÓN
Los resultados sugieren la ocurrencia de infecciones mixtas (P. falciparum + P. vivax; P. vivax + P. malariae) en el área investigada, las que no han sido identificadas por el método de microscopia convencional (TS). Además, los dos métodos para obtención de ADN analizados, el de lavado26 y el de extracción con saponina/chelex-10027, presentaron sensibilidad y alta especificidad, bien como un menor costo en comparación con los kits comerciales; y también fueron considerados de ejecución fácil y rápida. El segundo método, sin embargo, fue considerado el más sensible: suministra un protocolo simple para la extracción de ADN de muestras de sangre seco y podría ser el protocolo de elección para la obtención del ADN.
AGRADECIMIENTOS
A Ivana Pimentel y a los técnicos del Laboratorio de Investigación en Malaria del Instituto Evandro Chagas por su apoyo técnico.
REFERENCIAS
1 Alger J, Acosta MC, Lozano C, Velasquez C, Labrada LA. Stained smears as a source of DNA. Mem Inst Oswaldo Cruz. 1996;91(5):589-91.
2 Alves FP, Durlacher RR, Menezes MJ, Krieger H, Silva LH, Camargo EP. High prevalence of asymptomatic Plasmodium vivax and Plasmodium falciparum infections in native Amazonian populations. Am J Trop Med Hyg. 2002;66(6):641-8. [ Links ]
3 Ciceron L, Jaureguiberry G, Gay F, Danis M. Development of a Plasmodium for monitoring efficacy of antimalarial treatment. J Clin Microbiol. 1999;37(1):35-8. [ Links ]
4 Di Santi SM, Kirchgatter K, Brunialti KC, Oliveira AM, Ferreira SR, Boulos M. PCR - based diagnosis to evaluate the performance of malaria reference centers. Rev Inst Med Trop Sao Paulo. 2004 Jul-Aug;46(4):183-7. Doi:10.1590/S0036-46652004000400002 [ Links ]
5 Ebrahimzadeh A, Fouladi B, Fazaeli A. High rate of detection of mixed infections of Plasmodium vivax and Plasmodium falciparum in South-East of Iran, using nested PCR. Parasitol Int. 2007;56(1):61-4. Doi:10.1016/J.Parint.2006.12.001 [ Links ]
6 Edoh D, Steiger S, Genton B, Beck HP. PCR amplifications of DNA from malaria parasites on fixed and stained thick and thin blood films. Trans R Soc Trop Med Hyg. 1997 May-Jun;91:361-3. [ Links ]
7 Farnert A, Arez AP, Correia AT, Bjorkman A, Snounou G, Rosário V. Sampling and storage of blood and detection of malaria parasites by polymerase chain reaction. Trans R Soc Trop Med Hyg. 1999 Jan-Fev;93:50-3. [ Links ]
8 Gilles HM. The malaria parasite. In: Gilles HM, Warrell DA, editors. Essential Malariology. London: Edward Arnold. 1993. p. 12-34.
9 Haghdoost AA, Mazhari S, Bahadini K. Comparing the results of light microscopy with the results of PCR method in the diagnosis of Plasmodium vivax. J Vec Dis. 2006;43:53-7. [ Links ]
10 Imirzalioglu C, Soydan N, Schaller M, Bretzel RG, Chakraborty T, Domann E. Diagnosis of mixed Plasmodium malariae and P. vivax infection in a development aid volunteer by examination of bone-marrow specimens by real-time PCR. J Clin Microbiol. 2006 Jun;44(6):2307-10. Doi: 10.1128/JCM.02687-05 [ Links ]
11 Kimura M, Kaneko O, Liu Q, Zhou M, Kawamoto F, Wataya Y, et al. Identification of the four species of human malaria parasites by nested PCR that targets variant sequences in the small subunit rRNA gene. Parasitology Int. 1997;46:91-5.
12 Maeno Y, Nakazawa S, Dao le D, Yamamoto N, Giang ND, Van Hanh T, et al. A dried blood sample on filter paper is suitable for detecting Plasmodium falciparum gametocytes by reverse transcription polymerase chain reaction. Acta Trop. 2008;107(2):121-7. Doi:10.1016/j.actatropica.2008.05.001 [ Links ]
13 McNamara DT, Thomson JM, Kasehagen LJ, Zimmerman PA. Development of a multiplex PCR-ligase detection reaction assay for diagnosis of infection by the four parasite species causing malaria in humans. J Clin Microbiol. 2004 Jun;42(6):2403-10. Doi: 10.1128/JCM.42.6.2403-2410.2004 [ Links ]
14 Ministério da Saúde (BR). Manual de Diagnóstico Laboratorial da Malária. 2. ed. Brasília; 2009. 112 p.
15 Ministério da Saúde (BR), Secretaria de Vigilância em Saúde. Boletim epidemiológico da malária no 02. 2005.
16 Ministério da Saúde (BR). Situação Epidemiológica da Malária no Brasil, ano de 2008. Brasília, 2008. Disponível em: http://portal.saude.gov.br/portal/arquivos/pdf/folder_malaria_2009_web.pdf.
17 Organização Mundial de Saúde. revised July, 2003. Fact sheet no 94. Disponível em: http://www.who.int.dsa.
18 Pan American Health Organization. Regional Strategic Plan for Malaria in the Americas 2006-2010. Washington, D.C: PAHO; 2006. 85 p.
19 Rougemont M, Van Saanen M, Sahli R, Hinrikson HP, Bille J, Jaton K. Detection of Four Plasmodium Species in Blood from Humans by 18S rRNA Gene Subunit-Based and Species-Specific Real-Time PCR Assays. J Clin Microbiol. 2004 Dec;42(12):5636-43. [ Links ]
20 Schindler HC, Montenegro L, Montenegro R, Carvalho AB, Abath FG, Jaureguiberry G. Development and optimization of polymerase chain reaction-based malaria diagnostic methods and their comparison with quantitative buffy coat assay. Am J Trop Med Hyg. 2001 Oct;65(4):355-61. [ Links ]
21 Scopel KK, Fontes CJ, Nunes AC, Horta MF, Braga EM. High prevalence of Plasmodium malariae infections in a Brazilian Amazon endemic area (Apiacás - Mato Grosso State) as detected by polymerase chain reaction. Acta Trop. 2004 Mar;90(1):61-4. Doi:10.1016/J.Actatropica.2003.11.002 [ Links ]
22 Singh B, Bobogare A, Cox-Singh J, Snounou G, Abdullah MS, Rahman HA. A Genus - and species-specific nested polymerase chain reaction malaria detection assay for epidemoiologic studies. Am J Trop Med Hyg. 1999 Apr;60(4):687-92. [ Links ]
23 Snounou G, Viriyakosol S, Jarra W, Thaithong S, Brown KN. Identification of the four human malaria parasite species in field samples by the polymerase chain reaction and detection of a high prevalence of mixed infections. Mol Biochem Parasitol. 1993 Apr;58(2):283-92. [ Links ]
24 Snounou G, Viriyakosol S, Zhu XP, Jarra W, Pinheiro L, Rosario VE, et al. High sensitivity of detection of human malaria parasites by the use of nested polymerase chain reaction. Mol Biochem Parasitol. 1993 Oct;61(2):315-20. [ Links ]
25 Toma H, Kobayashi J, Bouakham V, Arakawa T, Sato Y, Nambanya S, et al. A field study on malaria prevalence in southeastern Laos by polymerase chain reaction assay. Am J Trop Med Hyg. 2001;64(5,6):257-61. [ Links ]
26 Warhurst DC, Awad el Kariem FM, Miles MA. Simplified preparation of malarial blood samples for polymerase chain reaction. Lancet. 1991 Feb;337(8736):303-4. [ Links ]
27 Wooden J, Kyes S, Silbley CH. PCR and strain identification in Plasmodium falciparum. Parasitol Today. 1993 Aug;9(8):303-5.
28 World Health Organization. Malaria diagnosis - new perspectives: report of a joint WHO/USAID informal consultation, 25-27 October 1999. Geneva: WHO; 2000. 1091 p.
 Correspondência / Correspondence /
Correspondencia:
Correspondência / Correspondence /
Correspondencia:
Giselle Maria Rachid Viana
Laboratório de Pesquisas Básicas
em Malária,
Seção
de Parasitologia Instituto Evandro Chagas
BR
316, km 7, s/n. 67030-000,
Levilândia. Ananindeua-Pará-Brasil
Phone: 55 91 3214-2148
E-mail:giselleviana@iec.pa.gov.br
Recebido em / Received / Recibido en: 23/7/2009
Aceito
em / Accepted / Aceito en: 21/9/2010










 texto en
texto en 
 Curriculum ScienTI
Curriculum ScienTI