Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista Pan-Amazônica de Saúde
versão impressa ISSN 2176-6215versão On-line ISSN 2176-6223
Rev Pan-Amaz Saude v.1 n.2 Ananindeua jun. 2010
http://dx.doi.org/10.5123/S2176-62232010000200005
Comparação entre dois métodos de obtenção de DNA a serem usados como protocolos alternativos para a detecção de parasitas humanos causadores de malária por nested PCR
Giselle Maria Rachid Viana; Danielle Regina Lima Barbosa; Ediclei Lima do Carmo; José Mário Veloso Peres; José Maria Souza Nascimento; Marinete Marins Póvoa
Laboratório de Pesquisas Básicas em Malária, Seção de Parasitologia, Instituto Evandro Chagas/SVS/MS, Ananindeua, Pará, Brasil
Endereço para correspondência
Correspondence
Dirección para correspondencia
Título original: Comparison of two DNA obtainment methods as alternative protocols for the detection of human malária parasites by nested PCR. Traduzido por: André Monteiro Diniz.
RESUMO
Um diagnóstico laboratorial correto e preciso de malária humana ainda é considerado um desafio, pois o método de referência, o da gota espessa com colocação pelo Giemsa, apresenta limitações que dificultam o controle da malária. Devido a esses problemas, várias pesquisas têm objetivado desenvolver métodos alternativos para o diagnóstico da malária. Grande parte desses estudos aborda métodos de diagnóstico molecular, que têm acarretado o desenvolvimento de algumas alternativas ao método de coloração pelo Giemsa. No entanto, esses métodos, por sua vez, apresentam suas limitações, que incluem seu alto custo, a complexidade do protocolo e a variação da qualidade das fontes de DNA e de reagentes. Neste aspecto, a técnica de nested PCR tem demonstrado ser um bom método e pode ser melhorado usando uma fonte de DNA de alta qualidade. Neste estudo foram avaliados dois métodos para a obtenção de DNA de amostras de sangue seco colhidas em papel de filtro: 1) lavagem e 2) saponina/Chelex-100. O segundo método apresentou sensibilidade e especificidade mais altas em relação ao primeiro, pois detectou mais infecções, tanto simples como mistas, bem como infecções por Plasmodium malariae. Com base nesses resultados, apresentamos o segundo como o protocolo de escolha para a obtenção de DNA. A técnica de nested PCR usando saponina/Chelex-100 para extração de DNA pode ser um método alternativo ou complementar de diagnóstico de parasitas da malária humana, mas não é considerado adequado para o uso de rotina.
Palavras-chave: Malária; DNA; Reação em Cadeia da Polimerase.
INTRODUÇÃO
A malária é um dos principais problemas de saúde pública no Brasil e atinge especialmente populações pobres na Região Amazônica16,18. A identificação precisa das espécies de Plasmodium que infectam humanos é de extrema importância para que seja viabilizado o seu tratamento de forma rápida e adequada. O método de referência para o diagnóstico de malária é o esfregaço de gota espessa com coloração de Giemsa (TS); apresenta baixo custo, boa sensibilidade, permite a identificação das formas das espécies de Plasmodium em todas as fases de desenvolvimento e a quantificação dos estágios do parasita8,21. Geralmente, sua sensibilidade para a detecção pelo microscopista varia entre 10-30 paras¡tas/µL de sangue; no entanto, esta sensibilidade pode ser limitada quando há uma grande quantidade de amostras a serem examinadas em um intervalo de tempo muito curto. É possível obter resultados falsos-negativos devido aos baixos níveis de parasitemia, à existência de infecções mistas ou inexperiência por parte do microscopista. Este fato pode comprometer uma das principais estratégias de controle da malária: um diagnóstico rápido e preciso, que viabilize a realização do tratamento adequado2,3,7,20.
Para suplantar algumas limitações do método TS, alguns métodos baseados na reação em cadeia da polimerase (PCR), como o protocolo Nested-PCR desenvolvido por Kimura et al11, têm sido utilizados para detectar e identificar parasitas da malária. Esses métodos são mais sensíveis e específicos que o TS e podem detectar até mesmo um único parasita por microtítulo de sangue5,10,21,23. Contudo, o sucesso de uma técnica de PCR depende de vários fatores, tais como a qualidade dos reagentes e do DNA obtido de diferentes fontes. Além disso, já foi demonstrado que a amplificação de DNA de amostras de sangue seco em papel-filtro é uma fonte confiável de DNA quando as condições de transporte, de manipulação e armazenamento são consideradas adequadas para evitar a contaminação ou degradação do DNA1,4,6,12. Por esta razão, é importante testar diferentes métodos de obtenção de DNA como protocolos alternativos de detecção de parasitas da malária humana por Nested-PCR. Este estudo objetivou comparar as duas seguintes técnicas: 1) Extração de DNA por procedimento de lavagens26, e 2) Extração de DNA por meio de tratamento d e amostras de sangue seco com saponina/Chelex-100 em papel-filtro27.
MATERIAIS E MÉTODOS
Utilizamos neste estudo 75 amostras de sangue seco positivas (parasitemia variando de 0,001% a 2%) e 78 negativas em discos de papel-filtro Whatma®, de 2,5 cm de diâmetro (Titertek, ICN Biomedicals - England). As amostras de sangue foram coletadas de habitantes dos Municípios de Novo Repartimento (04o14' 55'' N; 50o 07' 25'' W), Parauapebas (06o 04' 03" N; 49o 54' 08" W), Tucuruí (03o 45' 03" N; 49o 40' 03" W) e Belém (01o 27' 20" N; 48o 30' 15" W), no Estado do Pará. Os controles para cada parasita da malária humana incluíram amostras positivas bem caracterizadas e água ultrapura e DNA humano para os controles negativos. Todas as amostras foram testadas e diagnosticadas segundo o método de esfregaço de gota espessa com coloração de Giemsa (TS). A quantificação dos parasitas foi realizada nas lâminas com amostras positivas14.
DNA OBTIDO PELO MÉTODO DE LAVAGEM
As amostras de papel-filtro foram preparadas colocando-se cinco gotas de 20 µL de cada amostra em discos de papel-filtro Whatman® (2,5 cm de diâmetro) (Titertek, ICN Biomedicals - England) e secando-as em temperatura ambiente (TA, 25-27o C). As amostras foram então armazenadas em embalagens plásticas previamente identificadas (BHL Limited, Poole - England), que haviam sido mantidas a uma temperatura de -20o C até a sua utilização. Foram lavadas com água destilada e solução salina 0,9%, utilizando um sistema de filtragem Millipore® para remover todos os inibidores de PCR26.
EXTRAÇÃO DE DNA PELO MÉTODO DE SAPONINA/CHELEX-100
Um quarto de cada gota seca em papel-filtro foi cortado em pequenos pedaços e incubado em gelo com solução de saponina 0,5% em PBS por 1 h. Posteriormente, a solução foi misturada usando-se um "vórtex" e o sobrenadante foi descartado. Foi feita então uma lavagem com PBS, foi adicionada uma solução de Chelex-100 20% em água e a solução foi incubada em banho seco por 1 h. Após a incubação, o sobrenadante foi transferido para tubos Eppendorf® estéreis e armazenado a -20o C27.
REAÇÃO DE NESTED-PCR
A reação de amplificação da subunidade do gene 18S do RNA ribossô mico (rRNA) do Plasmodium (ssu-rRNA) foi conduzida de acordo com o descrito no protocolo de Kimura et al11. Os primers P1 e P2 foram utilizados como primers universais no primeiro ciclo, e os primers F2, M1 e V1 foram utilizados como primers específicos para P. falciparum, P. malariae e P. vivax, respectivamente, no segundo ciclo. Os produtos obtidos pela Nested-PCR foram fracionados por meio de eletroforese em gel de agarose 2% (Ultra pure agarose, BRL 155517-014) a 100 V por 1 h e corada com brometo de etídio (5 µL/mL). Os tamanhos esperados dos produtos da PCR eram de 130 bp para o primeiro estágio e 100-110 bp para o segundo estágio. Os técnicos que realizaram a Nested-PCR não tiveram ciência dos resultados obtidos pela análise por microscopia.
ÉTICA
O protocolo desta pesquisa foi aprovado pelo Comitê de Ética em Pesquisa do Instituto Evandro Chagas (004/2003-CEP/IEC/SVS/MS). Os objetivos e procedimentos do estudo foram devidamente explicados para todos os participantes; todos os pacientes assinaram termo de consentimento livre e esclarecido e dados sociodemográficos e clínicos foram coletados.
RESULTADOS
Das 153 amostras, 49,01% (75/153) foram positivas pelo TS (com parasitemia variando de 0,001% a 2,0%) e 50,98% (78/153) foram negativas. Pela Nesfed-PCR, 43,79% (67/153) das amostras foram positivas e 56,20% (86/153) foram negativas com o método de extração do DNA por procedimento de lavagem, enquanto 51,63% (79/153) foram positivas e 48,36% (74/153) foram negativas com o método por saponina/Chelex-100 (Tabela 1).

O desempenho da Nesfed-PCR comparada com o TS foi o seguinte: 1) método de lavagem - sensibilidade = 89,33%, especificidade = 100%, acurácia = 94,77%; 2) saponina/Chelex-100 - sensibilidade = 100%, especificidade = 94,87%, acurácia = 97,39%. A concordância entre esses métodos ea análise por microscopia foi quase perfeita [Kappa = 0,8952 e 0,9477, respectivamente; IC 95%: variação de 0,80 a 1,00]. O método de saponina/Chelex-100 fornece amostras de DNA de melhor qualidade do que o método de lavagem.
A frequência de espécies de Plasmodium e de infecções mistas detectadas pela Nesfed-PCR usando o DNA extraído pelos métodos de lavagem e saponina/Chelex-100 está demonstrada na Tabela 2. O método de saponina/Chelex-100 forneceu melhores amostras de DNA, o que é demonstrado pelo fato de que, com este método, a Nesfed-PCR pôde detectar tanto infecções simples como mistas por P. malariae, e detectou um número maior de outras infecções do que o método de lavagem.

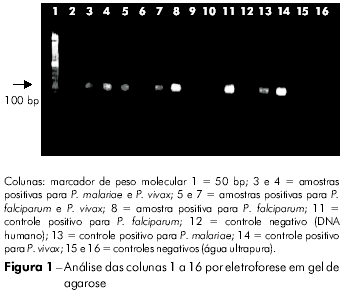
A figura 1 apresenta os resultados da análise por Nesfed-PCR usando DNA obtido pelo método de saponina/Chelex-100. As faixas 2, 3 e 4 contêm amostras de um único paciente; cada poço corresponde a reações para P. falciparum, P. malariae e P. vivax, respectivamente, demonstrando que o paciente apresenta uma infecção mista por P. malariae e P. vivax.
DISCUSSÃO
Apesar de o TS ainda ser o método de referência para o diagnóstico de malária, as técnicas moleculares são mais sensíveis e específicas para detectar parasitas e podem ser utilizadas para avaliar o desempenho das análises por microscopia20,28.
As amostras de sangue seco em papel-filtro constituem um método simples e acessível para coleta e armazenamento. Eles têm sido amplamente utilizados em triagem para diagnóstico, análise genética e estudos sobre epidemiologia molecular em áreas remotas com clima tropical, onde as condições de transporte e armazenamento não são ideais. A perda de sensibilidade de métodos baseados em PCR devido às condições de campo já foi reportada e é provavelmente causada pela baixa pureza, estabilidade e integridade do DNA obtido de amostras de sangue em papel-filtro. Um problema observado com a coleta em papel-filtro é a limitação no volume de amostras que podem ser utilizadas para a extração de DNA. Portanto, um bom isolamento do DNA é fundamental para assegurar a qualidade e eficácia de métodos moleculares7,20.
Vários estudos visaram reduzir os custos de protocolos baseados em PCR, bem como torná-los rápidos e precisos. Um fator essencial do protocolo para a aquisição de bons resultados é a obtenção de uma amostra de DNA de boa qualidade1,5,6,7.
Neste estudo realizamos uma análise comparativa de dois protocolos para a obtenção de DNA, levando em consideração o tempo necessário, seus custos e a qualidade do DNA extraído7. Os resultados demonstraram que a técnica de Nested-PCR com procedimento de lavagem não detectou oito amostras positivas (seis com P. vivax e dois com P. falciparum) identificadas por TS, enquanto o método de Nested-PCR com saponina/Chelex-100 detectou todas as amostras positivas identificadas por TS, bem como outras cinco amostras (três infecções simples por P. malariae e duas infecções mistas por P. vivax e P. malariae). Uma razão possível para a falha na detecção de algumas amostras positivas se deve ao fato de que o método de lavagem não eliminou inibidores de Nested-PCR adequadamente, como a hemoglobina. Os resultados demonstraram que o método de Nested-PCR com saponina/Chelex-100 se mostrou mais sensível e específico que o de lavagem, especialmente para a detecção de P. malariae.
Análises da extração de DNA com lavagem e com saponina/Chelex-100, como descrito por Warhurst et al26 e Wooden et al27, respectivamente, de 153 amostras de sangue seco em papel-filtro demonstraram que ambos os métodos são sensíveis e apresentam alta especificidade, além de serem de realização fácil e rápida. Além disso, pelo fato de esses métodos constituírem protocolos in house, seus custos foram mais baixos do que os kits comerciais disponíveis. No entanto, o método com saponina/Chelex-100 foi considerado mais sensível e, consequentemente, deveria ser o protocolo de escolha para a detecção de parasitas de malária com base na extração de DNA por Nested-PCR.
O protocolo de Nested-PCR desenvolvido por Kimura et al11 para espécies de Plasmodium que infectam humanos demonstrou ser útil, pois é bastante sensível, específico, é possível de ser reproduzido e não apresenta evidências de contaminação cruzada no estudo. Utilizando este protocolo, detectamos infecções mistas que haviam sido identificadas por TS como infecções simples, o que corrobora com os dados apresentados por Ebrahimzadeh et al5 , Scopel et al21 , Singh et al22, Snounou et al24 e Toma et al25.
Em casos de infecção mista, há predominância de uma espécie de Plasmodium, que normalmente é a detectada por ambas as técnicas, TS e PCR, enquanto que a espécie não dominante é identificada apenas por métodos baseados em análises de PCR. Além disso, um baixo nível de parasitemia de P. malariae é frequente principalmente em infecções mistas, provavelmente por causa das características desta espécie e do mecanismo de regulação dependente de densidade. Por isso, esta espécie poderia passar facilmente sem ser notada em análises por microscopia quando P. falciparum ou P. vivax são as espécies dominantes. Este fato também ajuda a explicar o maior número de infecções mistas detectadas por meio de métodos de biologia molecular, quando comparado com as ferramentas microscopias tradicionais27.
Os métodos de diagnóstico de malária baseados em PCR são bastante específicos e sensíveis; no entanto, não podem substituir as análises convencionais por microscopia para uso de rotina9, pois não permitem a quantificação das formas de parasitas. Ademais, eles são técnicas complexas, com várias etapas e alto custo. Portanto, eles podem ser úteis como ferramentas alternativas e complementares para o diagnóstico de malária humana em países em desenvolvimento4,13,19,22.
CONCLUSÃO
Os resultados sugerem a ocorrência de infecções mistas (P. falciparum + P. vivax; P. vivax + P. malariae) na área investigada, as quais não foram identificadas pelo método de microscopia convencional (TS). Além disso, os dois métodos para obtenção de DNA analisados, o de lavagem26 e o de extração com saponina/Chelex-10027, apresentaram sensibilidade e alta especificidade, bem como um custo mais baixo em comparação com os kits comerciais; eles também foram considerados de execução fácil e rápida. O segundo método, no entanto, foi considerado mais sensível: ele fornece um protocolo simples para a extração de DNA de amostras de sangue seco e poderia ser o protocolo de escolha para a obtenção do DNA.
AGRADECIMENTOS
A Ivana Pimentel e aos técnicos do Laboratório de Pesquisa em Malária do Instituto Evandro Chagas por seu apoio técnico.
REFERÊNCIAS
1 Alger J, Acosta MC, Lozano C, Velasquez C, Labrada LA. Stained smears as a source of DNA. Mem Inst Oswaldo Cruz. 1996;91(5):589-91.
2 Alves FP, Durlacher RR, Menezes MJ, Krieger H, Silva LH, Camargo EP. High prevalence of asymptomatic Plasmodium vivax and Plasmodium falciparum infections in native Amazonian populations. Am J Trop Med Hyg. 2002;66(6):641-8. [ Links ]
3 Ciceron L, Jaureguiberry G, Gay F, Danis M. Development of a Plasmodium for monitoring efficacy of antimalarial treatment. J Clin Microbiol. 1999;37(1):35-8. [ Links ]
4 Di Santi SM, Kirchgatter K, Brunialti KC, Oliveira AM, Ferreira SR, Boulos M. PCR - based diagnosis to evaluate the performance of malaria reference centers. Rev Inst Med Trop Sao Paulo. 2004 Jul-Aug;46(4):183-7. Doi:10.1590/S0036-46652004000400002 [ Links ]
5 Ebrahimzadeh A, Fouladi B, Fazaeli A. High rate of detection of mixed infections of Plasmodium vivax and Plasmodium falciparum in South-East of Iran, using nested PCR. Parasitol Int. 2007;56(1):61-4. Doi:10.1016/J.Parint.2006.12.001 [ Links ]
6 Edoh D, Steiger S, Genton B, Beck HP. PCR amplifications of DNA from malaria parasites on fixed and stained thick and thin blood films. Trans R Soc Trop Med Hyg. 1997 May-Jun;91:361-3. [ Links ]
7 Farnert A, Arez AP, Correia AT, Bjorkman A, Snounou G, Rosário V. Sampling and storage of blood and detection of malaria parasites by polymerase chain reaction. Trans R Soc Trop Med Hyg. 1999 Jan-Fev;93:50-3. [ Links ]
8 Gilles HM. The malaria parasite. In: Gilles HM, Warrell DA, editors. Essential Malariology. London: Edward Arnold. 1993. p. 12-34.
9 Haghdoost AA, Mazhari S, Bahadini K. Comparing the results of light microscopy with the results of PCR method in the diagnosis of Plasmodium vivax. J Vec Dis. 2006;43:53-7. [ Links ]
10 Imirzalioglu C, Soydan N, Schaller M, Bretzel RG, Chakraborty T, Domann E. Diagnosis of mixed Plasmodium malariae and P. vivax infection in a development aid volunteer by examination of bone-marrow specimens by real-time PCR. J Clin Microbiol. 2006 Jun;44(6):2307-10. Doi: 10.1128/JCM.02687-05 [ Links ]
11 Kimura M, Kaneko O, Liu Q, Zhou M, Kawamoto F, Wataya Y, et al. Identification of the four species of human malaria parasites by nested PCR that targets variant sequences in the small subunit rRNA gene. Parasitology Int. 1997;46:91-5.
12 Maeno Y, Nakazawa S, Dao le D, Yamamoto N, Giang ND, Van Hanh T, et al. A dried blood sample on filter paper is suitable for detecting Plasmodium falciparum gametocytes by reverse transcription polymerase chain reaction. Acta Trop. 2008;107(2):121-7. Doi:10.1016/j.actatropica.2008.05.001 [ Links ]
13 McNamara DT, Thomson JM, Kasehagen LJ, Zimmerman PA. Development of a multiplex PCR-ligase detection reaction assay for diagnosis of infection by the four parasite species causing malaria in humans. J Clin Microbiol. 2004 Jun;42(6):2403-10. Doi: 10.1128/JCM.42.6.2403-2410.2004 [ Links ]
14 Ministério da Saúde (BR). Manual de Diagnóstico Laboratorial da Malária. 2. ed. Brasília; 2009. 112 p.
15 Ministério da Saúde (BR), Secretaria de Vigilância em Saúde. Boletim epidemiológico da malária no 02. 2005.
16 Ministério da Saúde (BR). Situação Epidemiológica da Malária no Brasil, ano de 2008. Brasília, 2008. Disponível em: http://portal.saude.gov.br/portal/arquivos/pdf/folder_malaria_2009_web.pdf.
17 Organização Mundial de Saúde. revised July, 2003. Fact sheet no 94. Disponível em: http://www.who.int.dsa.
18 Pan American Health Organization. Regional Strategic Plan for Malaria in the Americas 2006-2010. Washington, D.C: PAHO; 2006. 85 p.
19 Rougemont M, Van Saanen M, Sahli R, Hinrikson HP, Bille J, Jaton K. Detection of Four Plasmodium Species in Blood from Humans by 18S rRNA Gene Subunit-Based and Species-Specific Real-Time PCR Assays. J Clin Microbiol. 2004 Dec;42(12):5636-43. [ Links ]
20 Schindler HC, Montenegro L, Montenegro R, Carvalho AB, Abath FG, Jaureguiberry G. Development and optimization of polymerase chain reaction-based malaria diagnostic methods and their comparison with quantitative buffy coat assay. Am J Trop Med Hyg. 2001 Oct;65(4):355-61. [ Links ]
21 Scopel KK, Fontes CJ, Nunes AC, Horta MF, Braga EM. High prevalence of Plasmodium malariae infections in a Brazilian Amazon endemic area (Apiacás - Mato Grosso State) as detected by polymerase chain reaction. Acta Trop. 2004 Mar;90(1):61-4. Doi:10.1016/J.Actatropica.2003.11.002 [ Links ]
22 Singh B, Bobogare A, Cox-Singh J, Snounou G, Abdullah MS, Rahman HA. A Genus - and species-specific nested polymerase chain reaction malaria detection assay for epidemoiologic studies. Am J Trop Med Hyg. 1999 Apr;60(4):687-92. [ Links ]
23 Snounou G, Viriyakosol S, Jarra W, Thaithong S, Brown KN. Identification of the four human malaria parasite species in field samples by the polymerase chain reaction and detection of a high prevalence of mixed infections. Mol Biochem Parasitol. 1993 Apr;58(2):283-92. [ Links ]
24 Snounou G, Viriyakosol S, Zhu XP, Jarra W, Pinheiro L, Rosario VE, et al. High sensitivity of detection of human malaria parasites by the use of nested polymerase chain reaction. Mol Biochem Parasitol. 1993 Oct;61(2):315-20. [ Links ]
25 Toma H, Kobayashi J, Bouakham V, Arakawa T, Sato Y, Nambanya S, et al. A field study on malaria prevalence in southeastern Laos by polymerase chain reaction assay. Am J Trop Med Hyg. 2001;64(5,6):257-61. [ Links ]
26 Warhurst DC, Awad el Kariem FM, Miles MA. Simplified preparation of malarial blood samples for polymerase chain reaction. Lancet. 1991 Feb;337(8736):303-4. [ Links ]
27 Wooden J, Kyes S, Silbley CH. PCR and strain identification in Plasmodium falciparum. Parasitol Today. 1993 Aug;9(8):303-5.
28 World Health Organization. Malaria diagnosis - new perspectives: report of a joint WHO/USAID informal consultation, 25-27 October 1999. Geneva: WHO; 2000. 1091 p.
 Correspondência / Correspondence /
Correspondencia:
Correspondência / Correspondence /
Correspondencia:
Giselle Maria Rachid Viana
Laboratório de Pesquisas Básicas
em Malária,
Seção
de Parasitologia Instituto Evandro Chagas
BR
316, km 7, s/n. 67030-000,
Levilândia. Ananindeua-Pará-Brasil
Phone: 55 91 3214-2148
E-mail:giselleviana@iec.pa.gov.br
Recebido em / Received / Recibido en: 23/7/2009
Aceito
em / Accepted / Aceito en: 21/9/2010










 texto em
texto em 
 Curriculum ScienTI
Curriculum ScienTI