Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Pan-Amazônica de Saúde
versión impresa ISSN 2176-6215versión On-line ISSN 2176-6223
Rev Pan-Amaz Saude v.1 n.3 Ananindeua sep. 2010
http://dx.doi.org/10.5123/S2176-62232010000300004
Epidemiologia molecular dos sorotipos 2 e 3 do vírus dengue, isolados no Brasil de 1991 a 2008
Ana Cecília Ribeiro CruzI; Ricardo GallerII; Eliana Vieira Pinto da SilvaI; Mayra de Oliveira e SilvaI; Adriana Ribeiro CarneiroI; Elizabeth Salbé Travassos da RosaI; Helena Baldez VasconcelosI; Eric Luiz Rodrigues de SáIII; Pedro Fernando da Costa VasconcelosI
ISeção de
Arbovirologia e Febres Hemorrágicas, Instituto Evandro Chagas/SVS/MS,
Ananindeua, Pará, Brasil
IIInstituto de Tecnologia em Imunobiológicos Bio-Manguinhos,
Fundação Oswaldo Cruz, Rio de Janeiro, Rio de Janeiro, Brasil
IIIFundação de Medicina Tropical do Tocantins,
Araguaina, Tocantins, Brasil
Endereço para correspondência
Correspondence
Dirección para correspondencia
Título original: Molecular epidemiology of dengue virus serotypes 2 and 3 isolated in Brazil from 1991 to 2008. Traduzido por: André Monteiro Diniz.
RESUMO
O vírus dengue (DENV1-4) causa a dengue clássica e a febre hemorrágica da dengue / síndrome de choque da dengue (FHD/SCD) em regiões tropicais e subtropicais. O objetivo deste estudo foi avaliar os genótipos circulantes de DENV2 e DENV3 obtidos em distintas regiões geográficas no período de 1991 a 2008. As análises filogenéticas de DENV2 demonstraram que o genótipo III (Sudeste da Ásia/América), apesar das diversas alterações nucleotídicas e de aminoácidos, foi o único a circular durante os últimos 19 anos. Desde a sua introdução no estudo, em 2000, todas as amostras isoladas de DENV3 analisadas foram agrupados no genótipo III (subcontinente indiano). Não foram encontradas evidências de que o DENV3 pertença a outros genótipos investigados.
Palavras-chave: Vírus da Dengue; Febre Hemorrágica da Dengue; Flavivirus.
INTRODUÇÃO
A dengue clássica (DC) e a febre hemorrágica da dengue/Síndrome de choque da dengue (FHD/SCD) são importantes problemas de saúde nas regiões tropicais e subtropicais. O número crescente de pacientes com manifestações clínicas graves e a expansão de áreas epidêmicas têm levado ao surgimento de muitas pesquisas acerca de seu agente causador, o vírus da dengue (DENV), que é o mais disseminado e importante vírus transmitido por mosquitos, em termos de morbidade e mortalidade1,2,3. Uma pandemia global de dengue, que teve início durante a Segunda Guerra Mundial, espalhou-se por todo o planeta e atingiu predominantemente os países tropicais. De acordo com a Organização Mundial de Saúde4, a DC causa aproximadamente 5 milhões de internações de crianças e, no mínimo, 50 mil mortes são causadas por FHD/SCD anualmente.
Os sorotipos 1 a 4 do DENV (DENV1 a DENV4) pertencem ao gênero Flavivirus, da família Flaviviridae. O genoma dos flavivírus caracteriza-se por ter um filamento único de RNA, de sentido positivo, e mede aproximadamente 11 Kb de comprimento, de onde é produzida uma única poliproteína. Esta poliproteína sofre proteólise por protease celular e viral, de modo a gerar 10 proteínas virais, e sua ordem é 5'-C-prM(M)-E-NS1-NS2A-NS2B-NS3-NS4A-NS4B-NS5-3'5,6.
As infecções pelo DENV podem se manifestar com quadros que variam de assintomáticos a subclínicos, passando por DC a FHD/CSD, os quais podem ser observados durante um surto de dengue. Entretanto, as infecções pelo DENV geralmente levam a duas síndromes bem conhecidas e definidas: DC e FHD. O mecanismo pelo qual o hospedeiro infectado pelo DENV desenvolve a doença benigna ou severa ainda não foi elucidado. O proposto mecanismo dependente de anticorpo, o qual precipita a exacerbação clínica, sugere que os anticorpos circulantes derivados de uma infecção primária aderem a um vírus heterólogo em uma infecção secundária, o que facilita a penetração de partículas do vírus em células mononucleares7,8. Outros estudos demonstraram que os mediadores do sistema imunológico do hospedeiro, como as citocinas e complemento, podem ter uma participação direta na patogênese do extravasamento de plasma, uma característica típica da FHD9,10,11,12.
No Brasil, o DENV2 foi isolado pela primeira vez em 1989, de um caso importado de Uganda, África. Antes deste caso, os sorotipos DENV1 e DENV4 já haviam causado uma epidemia em Boa Vista, Estado de Roraima, Brasil13. Em 1986, o DENV1 causou epidemias no Rio de Janeiro e em outras cidades14. Em 1990, foi observado o primeiro surto autóctone de DENV2 no Estado do Rio de Janeiro. A introdução do sorotipo DENV2 resultou em um aumento do número de casos graves da doença, com o surgimento de vários casos de FHD/SCD15,16. Além disso, o sorotipo DENV3 foi isolado no Estado de São Paulo a partir de um caso importado registrado em 200017. Um ano após a sua introdução no Brasil, o DENV3 causou um grande surto de dengue no Estado do Rio de Janeiro e rapidamente se espalhou para vários outros estados do Brasil18.
Neste estudo, efetuamos o sequenciamento dos genes C, prM/M e E dos sorotipos DENV2 e DENV3 em 27 cepas originadas de diferentes áreas geográficas do Brasil e coletadas em diferentes períodos entre 1991 e 2008, de modo a investigar a epidemiologia molecular dos sorotipos que atualmente circulam no país.
MATERIAIS E MÉTODOS
ISOLAMENTO VIRAL
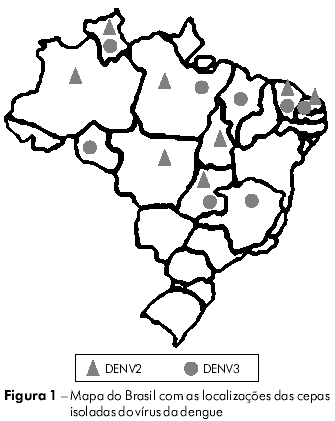
Vinte e sete cepas de DENV (14 de DENV2 e 13 de DENV3) foram obtidas a partir de uma coleta de vírus realizada pela Seção de Arbovirologia e Febres Hemorrágicas do Instituto Evandro Chagas (IEC). Os vírus foram isolados de amostras de soro coletadas de pacientes clinicamente diagnosticados como casos de DC, FHD/SCD e encefalite durante os surtos que ocorreram entre 1991 e 2008 e de mosquitos capturados na área endêmica (Figura 1). Os vírus foram cultivados em culturas de células de Aedesalbopictus (clone C6/36). As informações relevantes acerca de cada isolado estão resumidas na tabela 1.
EXTRAÇÃO DE RNA E AMPLIFICAÇÃO POR RT-PCR
As amostras de RNA genômico foram extraídas utilizando-se o reagente Trizol (Invitrogen, EUA), seguindo as instruções do fabricante. Os cDNAs foram sintetizados e amplificados por RT-PCR em duas etapas, com primers específicos para o DENV2 e o DENV3, os quais foram desenvolvidos para gerar produtos sobrepostos correspondentes à região genômica estrutural C/prM/M/E, conforme mencionado anteriormente. Inicialmente, 0,2-1 µg de RNA viral e 20 µM do primer complementar (antisense) foram aquecidos a 90 oC por 90 s e depois colocados em gelo. A mistura de RT contendo uma solução tampão de RT 1X (200 mM Tris-HCl [pH 8,4], 500 mM KCl), 0,2 mM de DNTP (deoxinucleotídeo), 100 mM DTT (ditiotreitol), 40 U de inibidor de RNAse (Invitrogen, EUA) e 200 U de transcriptase reversa Superscript II (Invitrogen, USA) foi adicionada ao RNA pré-aquecido e o volume foi ajustado para 20 µL. A reação RT foi realizada a uma temperatura de 45 oC por 1 h, seguida de um aquecimento a 94 oC por 10 min. Os produtos da RT foram utilizados como moldes para a amplificação por PCR. As reações foram compostas de 20 µM para cada um dos primers sense e antisense, tampão de PCR 1X, 1 mM de MgCl2, 0,2 µM de dNTP, 5 U/µL de Platinum Taq Polymerase (Invitrogen) e o volume final foi ajustado para 50 µL com água livre de RNAse. As amostras foram colocadas em um termociclador (Perkin Elmer 9600, EUA) a uma temperatura de 94 oC por 90 s (para uma desnaturação inicial); em seguida, foram realizados 35 ciclos de: 94 oC por 30 s, 55 oC por 30 s e 72 oC por 120 s, e uma etapa final de 72 oC por 5 min. Os produtos da RT-PCR foram visualizados em gel de agarose 1,5% corado com brometo de etídeo (5µg/ml).
SEQUENCIAMENTO NUCLEOTÍDICO E ANÁLISES SEQUENCIAIS
Os produtos da RT-PCR foram purificados com o QIAquick gel extraction kit (Qiagen, Inc., Valencia, CA). O cDNA purificado foi utilizado como molde para o sequenciamento com o kit Big Dye Terminator 3.0 (Applied Biosystems, Inc., EUA), seguindo as orientações do fabricante. O sequenciamento foi realizado utilizando o equipamento ABI Prism 377 (Applied Biosystems). As sequências nucleotídicas foram analisadas e editadas com o software SeqMan (DNASTAR, Lasergene software package). A análise filogenética foi realizada por meio de alinhamentos sequenciais progressivos múltiplos de pares, utilizando o CLUSTAL W (software Megalign; Lasergene software package DNASTAR).
Uma sequência de pares de base abrangendo os genes das proteínas estruturais C-prM-E foi utilizada para comparar os 14 isolados descritos e 22 sequências anteriormente publicadas de DENV2 coletadas do GenBank (www.ncbi.nlm.nih.gov). Um total de 1.977 bases dos genes M e E foram utilizadas para realizar a comparação dos 13 isolados com as 27 sequências previamente publicadas de DENV3. A sequência de DENV2 (cepa Jamaica, número de acesso M20558) foi usada como um outgroup.
As análises filogenéticas foram executadas com valores de bootstrap de mil pseudorréplicas, pelo método de neighbor-joining (NJ) Kimura-2-parameter {software MEGA 2.1) .
RESULTADOS
ANÁLISE DO DENV2
Determinamos a sequência nucleotídica completa do genoma estrutural de 14 isolados de DENV2 de pacientes com diferentes padrões de gravidade da doença (Tabela 1). As diferenças de números de nucleotídeos na comparação entre todos os vírus estudados e a cepa Jamaica (Jam83) do DENV2 revelaram uma grande semelhança (89,0 a 99,9%) de sequências nucleotídicas entre os nossos isolados. A semelhança média entre os subtipos de DENV2 foi de 89,5 e 99,8% para as sequências de nucleotídeos e aminoácidos, respectivamente.
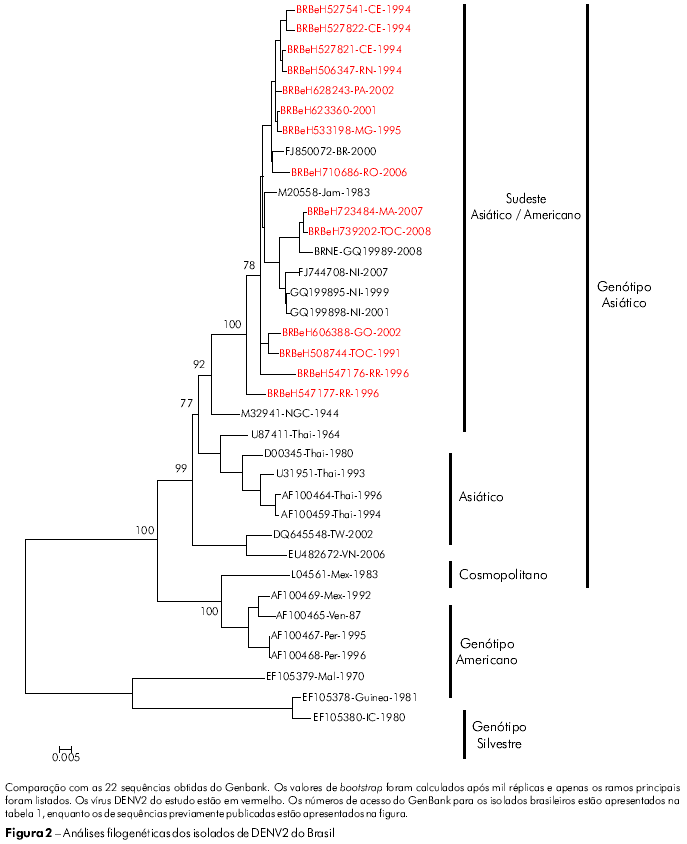
A classificação dos genótipos adotada foi a proposta por Rodriguez-Roche et al22 e Vasilaks e Weaver23 que utilizou o genótipo selvagem como outgroup. A árvore filogenética elaborada para as sequências nucleotídicas C/prM/M/E de nossos isolados de DENV2 agrupou todos os isolados no genótipo Jamaica (Figura 2; genótipo III). Além disso, nossos isolados de DENV2 foram mais proximamente relacionados com a cepa coletada no Brasil (RJ2000; FJ850072) e com uma cepa Jamaica de DENV2 (JAM1983; M20558). O genótipo asiático (III) foi dividido em quatro linhagens diferentes, que representam as cepas provenientes do sudeste da Ásia/América, Ásia I, Ásia II e Ásia III.
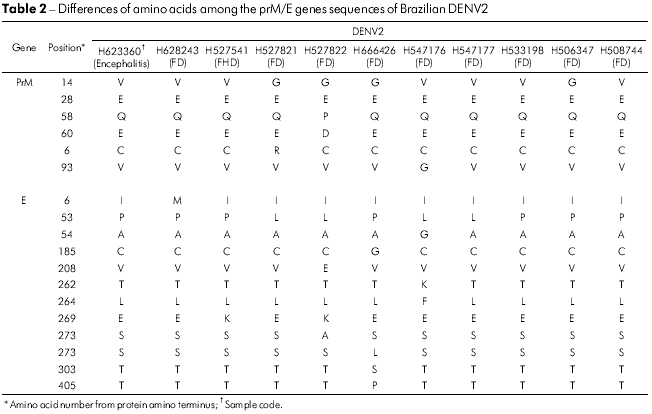
As sequências de aminoácidos prM/M/E de todos os 14 isolados brasileiros de DENV2 demonstraram que essas proteínas estavam bastante conservadas. Apenas cinco posições nos genes prM/M/E variaram nos cinco isolados, sendo que o aminoácido 14 do gene prM, o qual era uma glicina (V → G) em quatro cepas, foi o que mais variou. O isolado BeH527822 acumulou duas mudanças nas posições prM/M 58 (Q → P) e 60 (E → D), como demonstrado na tabela 2. No gene E, 12 posições de aminoácidos se mostraram variáveis, sendo que E53 (P → L) e E269 (E → K) foram observadas em quatro e dois isolados, respectivamente. Os isolados mais variáveis foram B eH 5 27 8 22, B eH 6 6 6 426 e BeH547176, que apresentaram mudanças em quarto posições. A única que foi observada em todos eles foi a E53 (P → L) (Tabela 2).
ANÁLISE DO DENV3
A homologia das sequências nucleotídicas entre as cepas do vírus DENV3 variou entre 89,7 e 100%. As sequências deduzidas de aminoácidos nas posições prM/M e E em todos os dez isolados de DENV3 mostraram que suas proteínas apresentavam-se bastante conservadas, com uma taxa de similaridade variando entre 93,2 e 100%. A homologia sequencial média foi de 94,8% e 96,6% para as sequências de nucleotídeos e aminoácidos, respectivamente.
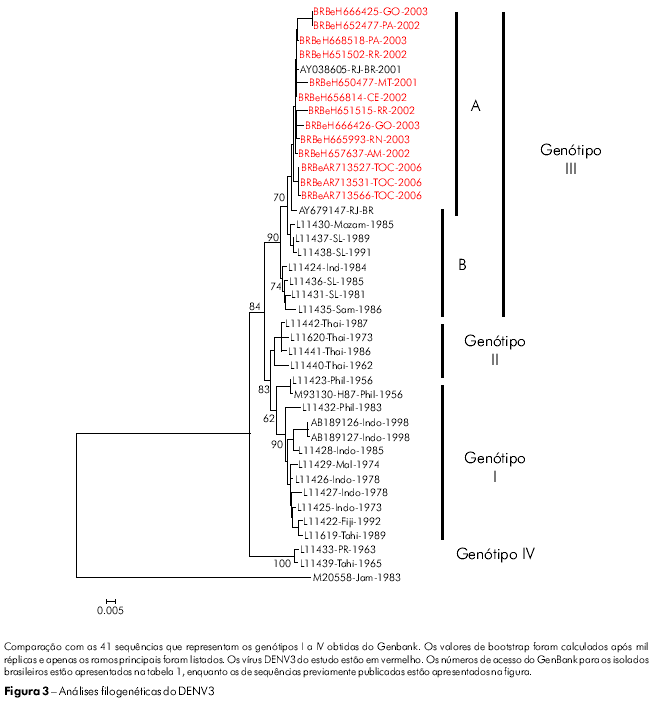
A árvore filogenética desenvolvida para os isolados de DENV3 teve por base o alinhamento de sequências observado na região E/NS1 (992-2550 nt), utilizando-se dos critérios de divisão dos genótipos descritos por Lanciotti24. Todos os isolados de DENV3 foram identificados como membros do genótipo III (subcontinente indiano) e foram agrupados em dois subclados (A e B). O subclado A contém as cepas latino-americanas e é considerado geneticamente mais próximo das cepas do subclado B isoladas em Sri Lanka (L11437, L11438) (Figura 3).
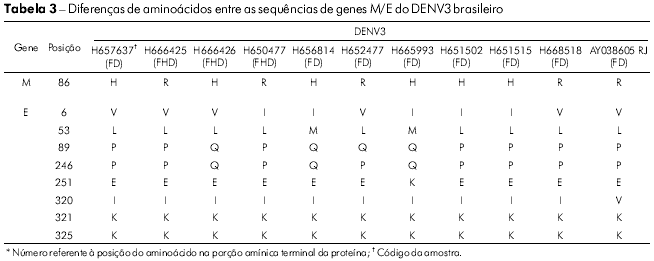
A variabilidade das sequências de aminoácidos para os isolados de DENV3 restringiu-se a uma posição no gene M (M86 H → R), enquanto que foram encontradas variações em 6 posições no gene E (Tabela 3). As posições que mais apresentaram variação foram E6 (I → V), E89 (Q → P) e E246 (Q → P). Os isolados BeH657637 e BeH665993 acumularam mudanças em três das seis posições variáveis dos aminoácidos (Tabela 3).
DISCUSSÃO
Adivergência entre sequências nucleotídicas (1 0,5%) para o DENV2 obtida neste estudo sugere um acúmulo de mutações durante o período de alta circulação deste sorotipo do DENV25. De fato, o DENV2 circula no Brasil por quase duas décadas; no entanto, surpreendentemente tem havido uma baixa incidência de casos de FHD se comparado com outros países. Desde a introdução do DENV3, em 2002, o número de casos de FHD tem aumentado drasticamente, o que sugere uma alta virulência deste sorotipo26. Estudos anteriores sobre o DENV2 nas Américas já haviam demonstrado que a emergência da FHD foi relacionada à introdução de um genótipo asiático no Caribe27.
Nossos dados confirmam que este genótipo do DENV2 tem sido predominante por todos os 19 anos de circulação do DENV no Brasil.
Segundo a classificação proposta por Rodriguez-Roche et al22 e Vasilaks & Weaver23, o DENV2 é agrupado em três genótipos, sendo que os seus genótipos III e IV circulam nas Américas. O genótipo III circulou em todos os países das Américas e teve como protótipo a cepa Jamaica, que tem sido associada a casos de FHD. O genótipo IV (americano) tem sido associado a cepas de virulência mais baixa e à DC, apenas. E importante observar que este genótipo tem circulado somente no subcontinente americano28. Em nosso estudo, nenhuma das sequências brasileiras analisadas foram incluídas no genótipo IV (americano) do DENV2. Na verdade, todas as sequências foram agrupadas no genótipo III (Figura 2).
Os primeiros estudos comparando a sequência nucleotídica do gene de 12 isolados de DENV2 não mostraram qualquer correlação entre a gravidade da doença e a sequência de nucleotídeos ou de aminoácidos29. Mesmo estudos mais detalhados sobre o genoma completo do genótipo do DENV2 do sudeste da Ásia não conseguiram identificar sítios específicos que determinassem sua virulência30,31. Leitmeyer et al28 conduziram uma análise mais consistente da diversidade genética do DENV2. Este estudo identificou uma substituição do aminoácido na posição E390 como determinante primário da dengue grave. No caso do genótipo americano, foi descoberto que os casos de DC eram associados à presença de ácido aspártico (Asp-D), que pode alterar as interações com os receptores celulares. No entanto, os resultados obtidos com 14 sequências de DENV2 em nosso estudo identificaram um Asn na posição E390 em todas as sequências analisadas quando comparadas às 14 cepas e às cepas dos genótipos asiático e americano28. Os resultados indicam que o DENV2 brasileiro analisado tem o potencial de causar FHD, o que se mostra consistente com os dados apresentados por pesquisas com isolados originados da Venezuela32.
A mudança do aminoácido E390 está localizada na região carboxílica terminal III (aa 303-395) na superfície lateral, que deve conter resíduos envolvidos no tropismo e na virulância de diferentes flavivírus33,34,35. No gene E de 14 vírus brasileiros analisados, 12 mudanças são observadas e apenas cinco substituições de aminoácidos foram associadas de forma significativa a uma mudança de caráter e polaridade. Essas posições foram: E208 (Val/V → Glu/E), 262 (Thr/T → Lys/K), 273 (Ser/S → Leu/L), 274 (Ser/S → Ala/A) e 405 (Thr/T → Pro/P). Todas as mudanças de aminoácidos são apresentadas na tabela 2. As mudanças foram observadas no ectodomínio III ou fora do ectodomínio da proteína, como no resíduo 405 da amostra H666426/Goi4191. Algumas mudanças apresentadas na tabela 2, como a E262 e 264, que são observadas em H547176/ROR1811 e na cepa viral originária da Venezuela (AF100466), sugerem que o vírus tenha se originado naquele país, confirmando então a existência de duas formas de introdução do DENV2 no Brasil.
Nas últimas duas décadas, o DENV3 tem causado epidemias de FHD no sudeste da Ásia, leste da África e América Latina. A primeira análise filogenética da proteína E deste sorotipo da dengue resultou na identificação de quatro genótipos distintos24. Em geral, os genótipos podem ser agrupados segundo a sua origem geográfica: Américas, subcontinente indiano, Tailândia e sudeste da Ásia/Pacífico Sul25.
O genótipo de DENV3 que circulou nas Américas até 1989 apresentava um baixo potencial epidêmico e foi isolado apenas de pacientes com DC. Entre 1980 e 1990, dois novos genótipos foram introduzidos na região do Pacífico Sul e nas Américas: o genótipo do sudeste da Ásia (genótipo I) é associado a grandes epidemias de FHD no Taiti e em Fiji36, e o genótipo do subcontinente indiano (genótipo III) foi introduzido na América Central na década de 1990 pela Nicarágua37,38,39,40.
Em 1994, o DENV3 ressurgiu nas Américas, causando um pequeno surto associado à DC no Panamá. O vírus se alastrou em direção ao norte da América Central até a Nicarágua e México41,2. Sete anos após a sua introdução nas Américas, o DENV3 se espalhou pela América do Sul até a Venezuela, Paraguai e Brasil, causando grandes epidemias42,43,17,44,45.
Os 13 isolados brasileiros de DENV3 em nosso estudo foram coletados em diferentes regiões, e, juntamente com os isolados obtidos em países vizinhos45,46,40, foram todos agrupados no genótipo III (subcontinente indiano). Eles são geneticamente diferentes da cepa de DENV3 que circulou nas Américas na década de 1960, que pertencia ao genótipo V47. É possível que esta variabilidade acumulada seja o resultado de sua rápida dispersão e do aumento em sua replicação viral. Esta hipótese é corroborada pelos valores de bootstrap de aproximadamente 84% mostrados pela árvore filogenética desenvolvida segundo o método neighbor-joining (Figura 3). A variabilidade da cepa de DENV3 (genótipo III) pode ser creditada às suas múltiplas introduções no Brasil, que deu ensejo à classificação em subgrupos de acordo com sua origem e ano de isolamento de sua cepa viral (Figura 3).
A transmissão local do DENV3 foi detectada inicialmente no Estado do Rio de Janeiro em dezembro de 2000. No verão de 2001, ocorreu a primeira epidemia autóctone na Cidade do Rio de Janeiro, com um alto número de casos de FHD. Em seguida, houve uma disseminação deste genótipo por todo o Brasil. Todos os vírus isolados pertenciam ao genótipo III, semelhante ao que circulava no Sri Lanka, corroborando o que já havia sido descrito em estudos anteriores47,48,49. Com base na cronologia do isolamento do vírus, nossos resultados demonstram que a dispersão do genótipo III do vírus DENV3 no Brasil ocorreu no sentido Sudeste-Norte. Esses achados diferem daqueles apresentados por Figueiredo et al50, que apontavam para a circulação do genótipo I no Estado de Minas Gerais, Sudeste do Brasil, e por Araújo et al51 que sugeria a formação de um novo genótipo (V), que agruparia as cepas de DENV3 do Brasil, China e Japão. As diferenças nos resultados mostram que a circulação de novos genótipos pareceu se restringir a uma região específica do Brasil ou a um certo período de tempo. Este fato faz surgir algumas questões importantes: Por que outras cepas destes genótipos não puderam ser identificadas? Esta cepa reserva um potencial de transmissão? Portanto, a emergência e o surgimento de outros ou novos genótipos devem ser mais investigados. Estes estudos devem incluir a análise as sequências completas do genoma do vírus DENV3 e de seu perfil epidemiológico.
CONCLUSÃO
Nosso estudo sugere que a baixa variabilidade genética do DENV2 circulante no Brasil pode explicar a baixa incidência de casos graves da doença registrados até o presente momento. No entanto, a disseminação em massa do genótipo III do DENV2, o qual apresentou uma maior variabilidade que o DENV3, pode ter ocasionado um acúmulo de mudanças genéticas associadas ao aumento da virulência, ao status imunológico e outros fatores importantes descritos. Este fato poderia explicar o grande número de casos graves de dengue deste a introdução de DENV2 e DENV3 no Brasil.
AGRADECIMENTOS
Agradecemos a valiosa contribuição dos Drs. Márcio Roberto Teixeira Nunes, Ralph Lainson e Elena Caride, Creuza Lima Carvalho e Maria Natividade.
APOIO FINANCEIRO
Esta pesquisa foi parcialmente financiada pelo Conselho Nacional de Desenvolvimento Científico e Tecnológico, Brasil (CNPq) (bolsas 300460/2005-8 e 501558/2003-9).
REFERÊNCIAS
1 Monath TP, Heinz FX. Flaviviruses. In: Fields BN, Knipe DM, Howley PM, editors. Field's Virology. Philadelphia: Lippincott Williams Wilkins; 1996. p. 961-1034.
2 Guzmán MG, Kouri G. Dengue: an update. Lancet Infect Dis. 2002 Jan;2(1):33-42.
3 Kourí G. El dengue, un problema cresciente de salud en las Américas. Rev Panam Salud Publica. 2006;19(3):143-5. [ Links ]
4 World Health Organization. Bulletin of Dengue. 1986
5 Chambers TJ, Hahn CS, Galler R, Rice CM. Flavivirus genome organization, expression, and replication. Annu Rev Microbiol. 1990;44:649-88.
6 Rice CM. Flaviviridae: the viruses and their replication. In: Fields BN, Knipe DM, Howley PM, editors. Fields Virology. Philadelphia: Lippincott Williams Wilkins; 1996. p. 931-60.
7 Halstead SB. Pathogenesis of dengue: challenges to molecular biology. Science. 1988 Jan;239(4839):476-81. [ Links ]
8 Littaua R, Kurane I, Ennis FA. Human IgG Fc receptor II mediates antibody-dependent enhancement of dengue virus infection. J Immunol. 1990 Apr;144(8):3183-6. [ Links ]
9 Kurane I, Ennis FE. Immunity and immunopathology in dengue virus infections. Semin Immunol. 1992 Apr;4(2):121-7. [ Links ]
10 Rothman AL, Ennis FA. Imunnopathogenesis of dengue haemorrhagic fever. Virology. 1999;257:1-6.
11 Chaturvedi UC, Argwal R, Elbishbishi EA, Mustafá AS. Cytokine cascade in dengue haemorrhagic fever: inplications for pathgenesis. FEMS Immunol Med Microbiol. 2000 Jul;28(3):183-8. [ Links ]
12 Rothman AL. Dengue: defining protective versus pathologic immunity. J Clin Invest. 2004 Apr;113(7):946-51.
13 Travassos da Rosa APA, Travassos da Rosa JFS, Pinheiro FP, Vasconcelos PFC. Arboviroses. In: Leão RNQ, editor. Doenças Infecciosas e Parasitárias. Belém: CEJUP; 1997. p. 207-26.
14 Schatzmayr HG, Nohueira RM, Travassos da Rosa AP. An outbreak of dengue virus at Rio de Janeiro – 1986. Mem Inst Oswaldo Cruz. 1986 Apr-Jun;81(2):245-6. [ Links ]
15 Nogueira RM, Miagostovich MP, Lampe E, Souza RW, Zagne SM, Schatzmayr HG. Dengue epidemic in the stage of Rio de Janeiro, Brazil, 1990-1: co-circulation of dengue 1 and dengue 2 serotypes. Epidemiol Infect. 1993 Aug;111(1):163-70. [ Links ]
16 Zagne SM, Alves VG, Nogueira RM, Miagostovich MP, Lampe E, Tavares W. Dengue haemorrhagic fever in the state of Rio de Janeiro, Brazil: a study of 56 confirmed cases. Trans R Soc Trop Med Hyg. 1994 Nov-Dec;88(6):677-9.
17 Rocco IM, Kavakama BB, Santos CL. First isolation of dengue 3 in Brazil from an imported case. Rev Inst Med Trop Sao Paulo. 2001 Jan-Feb;43(1):55-7.
18 Siqueira Jr JB, Martelli CMT, Coelho GE, Simplício ACR, Hatch DL. Dengue and dengue hemorrhagic fever, Brazil, 1981-2002. Emerg Infect Dis. 2005 Jan;11(1):48-53.
19 Kumar S, Tamura K, Nei M. MEGA3: integrated software for molecular evolutionary genetics analysis and sequence alignment. Brief Bioinform. 2004 Jun;5(2):150-63. [ Links ]
20 Saitou N, Nei M. The neighbor-joining method: a new method for reconstructing phylogenetic trees. Mol Biol Evol. 1987 Jul;4(4):406-25. [ Links ]
21 Kimura M. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequence. J Mol Evol. 1980 Dec;16(2):111-20.
22 Rodriguez-Roche R, Alvarez M, Gritsun T, Halstead S, Kouri G, Gould EA, et al. Virus evolution during a severe dengue epidemic in Cuba, 1997. Virology. 2005;334(2):154-9.
23 Vasilakis N, Weaver SC. History and evolution of human dengue emergence. In: Maramorosch K, Shatkin AS, Murphy FA, editors. Advances in Virus Research. California: Academic Press; 2008. p. 1-76.
24 Lanciotti RS, Lewis JG, Gubler D, Trent DW. Molecular evolution and epidemiology of dengue 3 viruses. J Gen Virol. 1994 Jan;75(1):65-75. [ Links ]
25 Rico-Hesse R. Microevolution and virulence of dengue virus. In: Chambers TJ, Monath TP, editors. The Flaviviruses: structure, replication and evolution. California: Academic Press; 2003. p. 316-41.
26 Secretaria de Vigilância em Saúde. Plano Nacional de Combate a Dengue. Brasília: Ministério da Saúde; 2006.
27 Rico-Hesse R, Harrison LM, Salas RA, Tovar D, Nisalak A, Ramos C, et al. Origins of dengue type 2 viruses associated with increased pathogenicity in the Americas. Virology. 1997 Apr;230(2):224-51.
28 Leitmeyer KC, Vaughn DW, Watts DG, Salas R, Chacon IV, Ramos C, et al. Dengue virus structural differences that correlate with pathogenesis. J Virol. 1999 Jun;73(6):4738-47. [ Links ]
29 Block J, Samuel S, Gibbs AJ, Vitarana UT. Variation of the nucleotide and encoded amino acid sequences of the envelope gene from eight dengue 2 viruses. Arch Virol. 1989;105(1-2):39-53. [ Links ]
30 Mangada MN, IgarashI A. Molecular and in vitro analysis of eigth dengue type 2 viruses isolated from patients exhibiting different disease severities. Virology. 1998 May;244(2):458-66.
31 Pandey BD, Igarashi A. Severity-related molecular differences among nineteen strains of dengue type 2 viruses. Microbiol Immunol. 2000 Feb;44(3):179-88. [ Links ]
32 Uzcategui NY, Camacho D, Comach G, Cuello de Uzcategui R, Holmes EC, Gould EA. Molecular epidemiology of dengue type 2 virus in Venezuela: evidence for in situ virus evolution and recombination. J Gen Virol. 2001 Dec;82(12):2945-53. [ Links ]
33 Hasegawa HM, Yoshida T, Shiosaka S, Kobayashi Y. Mutations in the envelope protein of Japanese encephalitis virus affect entry into cultured cells and virulence in mice. Virology. 1992 Nov;191(1):158-65. [ Links ]
34 Jiang WR, Lowe A, Higgs S, Reid S, Gould EA. Single amino acid codon changes detected in louping ill virus antibody-resistant mutants with reduced neurovirulence. J Gen Virol. 1993 May;74(5):931-5. [ Links ]
35 Mandl CW, Guirakhoo F, Holzmann H, Heinz FX, Kunz C. Antigenic structure of the flavivirus envelope protein E at the molecular level, using tick-borne encephalitis virus as a model. J Virol. 1989 Feb;63(2):564-71. [ Links ]
36 Chungue E, Deubel V, Cassar O, Laille M, Martin PMV. Molecular epidemiology of dengue 3 viruses and genetic relatedness among dengue 3 strains isolated from patients with wild or severe form for dengue fever in French Polynesia. J Gen Virol. 1993 Dec;74(12):2765-70.
37 Balmaseda A, Sandoval E, Perez L, Gutierrez CM, Harris E. Application of molecular typing techniques in the 1998 dengue epidemic in Nicaragua. Am J Trop Med Hyg. 1999 Dec;61(6):893-7.
38 Harris E, Roberts TG, Smith L, Selle J, Kramer LD, Valle S, et al. Typing of dengue viruses in clinical specimens and mosquitoes by single-tube multiplex reverse transcriptase PCR. J Clin Microbiol. 1998 Sep;36(9):2634-9. [ Links ]
39 Harris E, Videa E, Perez L, Sandoval E, Tellez Y, Perez ML, et al. Clinical, epidemiologic, and virologic features of dengue in the 1998 epidemic in Nicaragua. Am J Trop Med Hyg. 2000 Jul-Aug;63(1- 2):5-11. [ Links ]
40 Usuku S, Castillo L, Sugimoto C, Noguchi Y, Yogo Y, Kobayashi N. Phylogenetic analysis of dengue 3 viruses prevalent in Guatemala during 1996-1998. Arch Virol. 2001 Jul;146(7):1381-90. DOI:10.1007/s007050170098 [ Links ]
41 Centers for Disease Control and Prevention. Dengue type 3 infection – Nicaragua and Panama, October-November 1994. MMWR Morb Mortal Wkly Rep. 1995 Jan;44(2):21-4. [ Links ]
42 Halstead SB. Dengue in the Americas and Southeast Asia: do they differ? Rev Panam Salud Publica. 2006 Dec;20(6):407-15. [ Links ]
43 Nogueira RMR, Miagostovich MP, Filippis AM, Pereira MA, Schatzmayr HG. Dengue virus type 3 in Rio de Janeiro, Brazil. Mem Inst Oswaldo Cruz. 2001 Oct;96(7):925-6. DOI:10.1590/S0074-02762001000700007 [ Links ]
44 Simone TS, Nogueira RM, Araújo ES, Guimarães FR, Santos FB, Schatzmayr HG, et al. Dengue virus surveillance: the co-circulation of DENV1, DENV2 and DENV3 in the State of Rio de Janeiro, Brazil. Trans R Soc Trop Med Hyg. 2004 Sep;98(9):553-62. [ Links ]
45 Aquino VH, Anatriello E, Gonçalves PF, Silva EV, Vasconcelos PF, Vieira DS, et al. Molecular Epidemiology of dengue type 3 virus in Brazil and Paraguay, 2002-2004. Am J Trop Med Hyg. 2006;75(4):710-5. [ Links ]
46 Alvarez M, Rodriguez-Roche R, Bernardo L, Vázquez S, Morier L, Gonzalez D, et al. Dengue Hemorrhagic Fever caused by sequential dengue 1-3 virus infections over a long time interval: Havana epidemic, 2001-2002. Am J Trop Med Hyg. 2006;75(6):1113-7. [ Links ]
47 Uzcategui NY, Comach G, Camacho D, Salcedo M, Cabello de Quintana M, Jimenez M, et al. Molecular epidemiology of dengue virus type 3 in Venezuela. J Gen Virol. 2003 Jun;84(6):1569-75. [ Links ]
48 Messer WB, Gubler DJ, Harris E, Sivananthan K, Silva AM. Emergence and global spread of a dengue serotype 3, subtype III virus. Emerg Infect Dis. 2003 Jul;9(7):800-9. [ Links ]
49 Nogueira RM, Schatzmayr HG, Filippis AM, Santos FB, Cunha RV, Coelho JO, et al. Dengue virus type 3, Brazil, 2002. Emerg Infect Dis. 2005 Sep;11(9):1376-81. [ Links ]
50 Figueiredo LB, Cecílio AB, Ferreira GP, Drumond BP, Oliveira JG, Bonjardim CA, et al. Dengue virus 3 genotype 1 associated with dengue fever and dengue hemorrhagic fever, Brazil. Emerg Infect Dis. 2008 Feb;14(2):314-6. [ Links ]
51 Araújo JMG, Bello G, Schatzmayr HG, Santos FB, Nogueira RMR. Dengue virus type 3 in Brazil: a phylogentic perspective. Mem Inst Oswaldo Cruz. 2009;104(3):526-9.
 Correspondência
/ Correspondence / Correspondencia:
Correspondência
/ Correspondence / Correspondencia:
Ana Cecília Ribeiro Cruz
Instituto Evandro Chagas,
Seção de Arbovirologia e Febres Hemorrágicas
Rodovia BR 316, km 7, s/nº. Bairro: Levilândia
CEP: 67030-000 Ananindeua-Pará-Brasil
Tel.: 55 (91) 3217-3199 | Fax: 55 (91) 3226-5262
E-mail:anacecilia@iec.pa.gov.br
Recebido em / Received / Recibido en: 31/7/2009
Aceito em / Accepted / Aceito en: 1/2/2010










 texto en
texto en 
 Curriculum ScienTI
Curriculum ScienTI