Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista Pan-Amazônica de Saúde
versão impressa ISSN 2176-6215versão On-line ISSN 2176-6223
Rev Pan-Amaz Saude v.1 n.3 Ananindeua set. 2010
http://dx.doi.org/10.5123/S2176-62232010000300005
ARTIGO ORIGINAL | ORIGINAL ARTICLE | ARTÍCULO ORIGINAL
Identificação genotípica de membros do complexo Mycobacterium avium isolados de infecções pulmonares no Estado do Pará, Brasil
Genotypic identification of members of the Mycobacterium avium complex isolated from pulmonary infections in Pará State, Brazil
Identificación genotípica de miembros del complejo Mycobacterium avium aislados de infecciones pulmonares en el Estado de Pará, Brasil
Ana Roberta Fusco da CostaI; Maria Luíza LopesI; Jeann Ricardo da Costa BahiaII; Emilyn da Costa ConceiçãoII; Karla Valéria Batista LimaI
ISeção de Bacteriologia e Micologia, Instituto Evandro Chagas/SVS/MS, Ananindeua, Pará, Brasil
IIInstituto Evandro Chagas/SVS/MS, Ananindeua, Pará, Brasil. Universidade Federal do Pará, Belém, Pará, Brasil
Endereço para correspondência
Correspondence
Dirección para correspondencia
RESUMO
INTRODUÇÃO: O complexo Mycobacterium avium (MAC) compreende micobactérias de crescimento lento, naturalmente encontradas no meio ambiente, capazes de causar infecções em diversas espécies de seres vivos, incluindo aves, suínos e humanos. Tais infecções podem ser assintomáticas, clinicamente significantes e, em alguns casos, fatais. No entanto, existe a necessidade de se disponibilizarem técnicas que ofereçam uma identificação conclusiva de bactérias estreitamente relacionadas. As técnicas de biologia molecular se apresentam como ferramentas promissoras para uma identificação mais precisa.
MATERIAIS E MÉTODOS: Neste trabalho foram avaliados os marcadores moleculares RNAr 16S, hsp65 e rpoB aplicados à distinção de membros do MAC isolados no Laboratório de Micobactérias do Instituto Evandro Chagas.
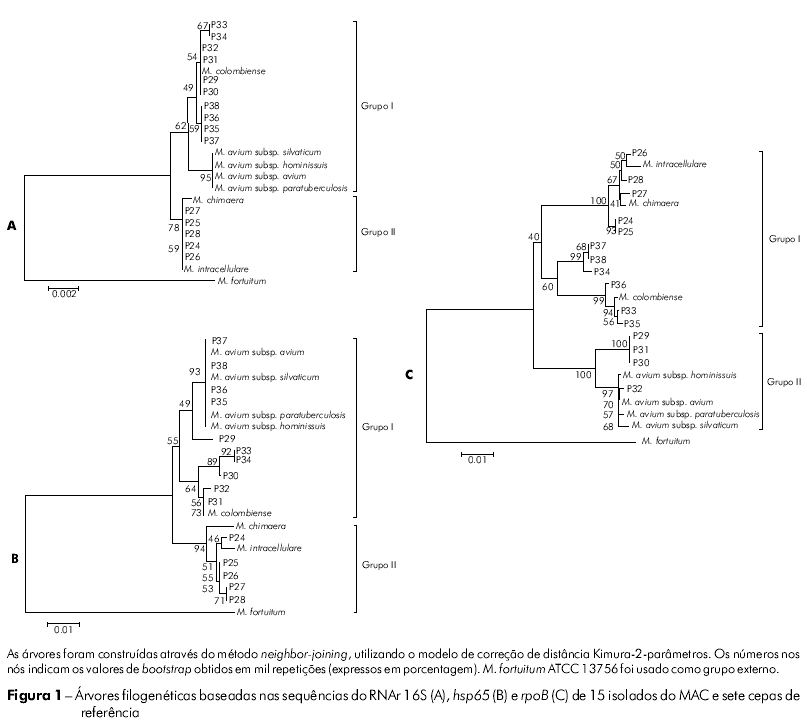
RESULTADOS: Amostras de MAC colhidas em 15 pacientes foram previamente avaliadas pelo método de análise de polimorfismo de fragmentos de restrição do gene hsp65 (PRA-hsp65), que forneceu três diferentes perfis: (I) BstEII: 235/115/100, HaeIII: 145/130/60; (II) BstEII: 235/210, HaeIII: 130/105; e (III) BstEII: 235/210, HaeIII:145/130. Árvores filogenéticas foram construídas após análise do RNAr 16S, em que as amostras foram distribuídas em dois grupos, semelhante ao encontrado na análise de hsp65. Entretanto, os resultados de rpoB foram discordantes daqueles das outras árvores, devido à modificação de topologia.
CONCLUSÃO: Os achados deste estudo sugerem que diferentes forças evolucionárias podem estar atuando sobre o gene rpoB, e, desta forma, é necessário que se tenha precaução ao estabelecer esse alvo para fins taxonômicos. Adicionalmente, recomenda-se que mais de um marcador, incluindo o RNAr 16S, seja avaliado para a identificação micobacteriana.
Palavras-chave: Complexo Mycobacterium avium; Polimorfismo de Fragmento de Restrição; Análise de Sequência de DNA; Filogenia.
ABSTRACT
INTRODUCTION: The Mycobacterium avium complex (MAC) is a group of slow-growing mycobacteria naturally found in the environment capable of causing infections in a wide variety of living species, including birds, swines and humans. These infections can be asymptomatic, clinically significant and, in some cases, fatal. There is a demand for techniques that are capable of conclusively identifying closely related bacteria. Molecular biological techniques are promising tools for a more precise identification.
MATERIAL AND METHODS: In this study, we evaluated the ability of 16S rRNA, hsp65 and rpoB molecular markers to distinguish between members of the MAC isolated in the Mycobacteria Laboratory at the Instituto Evandro Chagas.
RESULTS: MAC samples collected from 15 patients were previously evaluated using an hsp65 gene restriction fragment length polymorphism analytical method (RFLP-hsp65), and they showed three different profiles: (I) BstEII: 235/115/100, HaeIII: 145/130/60; (II) BstEII: 235/210, HaeIII: 130/105; and (III) BstEII: 235/210, HaeIII: 145/130. We constructed phylogenetic trees using 16S rRNA analysis in which the samples were distributed into two groups, similarly to those found in the hsp65 analysis. However, the results from the rpoB analysis disagreed with those of the other trees due to changes in topology.
CONCLUSION: The findings from this study warrant that various evolutionary forces may be acting on the rpoB gene. Thus, it is necessary to be cautious when using this target for taxonomic analyses. Additionally, we recommend that multiple markers (including16S rRNA) be evaluated when identifying mycobacteria.
Keywords: Mycobacterium avium Complex; Polymorphism, Restriction Fragment Length; Sequence Analysis, DNA; Phylogeny.
RESUMEN
INTRODUCCIÓN: El complejo Mycobacterium avium (MAC) comprende micobacterias de crecimiento lento, naturalmente encontradas en el medio ambiente, capaces de causar infecciones en diversas especies de seres vivos, incluyendo aves, porcinos y humanos. Tales infecciones pueden ser asintomáticas, clínicamente significantes y, en algunos casos, fatales. Sin embargo, existe la necesidad de poner a disposición técnicas que ofrezcan una identificación conclusiva de bacterias estrechamente relacionadas. Las técnicas de biología molecular se presentan como herramientas prometedoras para una identificación más precisa.
MATERIALES Y MÉTODOS: En este trabajo fueron evaluados los marcadores moleculares RNAr 16S, hsp65 y rpoB aplicados a la distinción de miembros del MAC, aislados en el Laboratorio de Micobacterias del Instituto Evandro Chagas.
RESULTADOS: Muestras del MAC colectadas en 15 pacientes fueron previamente evaluadas por el método de análisis de polimorfismo de fragmentos de restricción del gen hsp65 (PRA-hsp65), que suministró tres perfiles diferentes: (I) BstEII: 235/115/100, HaeIII: 145/130/60; (II) BstEII: 235/210, HaeIII: 130/105; y (III) BstEII: 235/210, HaeIII: 145/130. Se construyeron árboles filogenéticos luego del análisis del RNAr 16S, en el que las muestras fueron distribuidas en dos grupos, semejante al encontrado en el análisis de hsp65. Sin embargo, los resultados del rpoB fueron discordantes de los otros árboles, debido a la modificación de la topología.
CONCLUSIÓN: Los hallazgos de este estudio sugieren que distintas fuerzas evolutivas pueden estar actuando sobre el gen rpoB, y así, es necesario tener precaución al establecer esa meta para fines taxonómicos. Adicionalmente, se recomienda que más de un marcador, incluyendo el RNAr 16S, sea evaluado para la identificación micobacteriana.
Palabras clave: Complejo Mycobacterium avium; Polimorfismo de Longitud del Fragmento de Restricción; Análisis de Secuencia de ADN; Filogenia.
INTRODUÇÃO
O complexo Mycobacterium avium (MAC), juntamente com outras espécies micobacterianas atípicas, denominadas de micobactérias não tuberculosas (MNT), compreende micobactérias de crescimento lento, naturalmente encontradas no meio ambiente1, capazes de causar infecções em diversas espécies de seres vivos, incluindo aves, suínos e humanos, podendo se apresentar como assintomáticas, clinicamente significantes e, em alguns casos, fatais2. Esse complexo é constituído pelas espécies M. avium, M. intracellulare, M. colombiense e M. chimaera3,1.
O MAC representa um importante grupo de micobactérias associado a várias patologias humanas, especialmente às infecções disseminadas em pacientes com aids4. Além dessas, recentes evidências sugerem que a taxa de infecção em pacientes imunocompetentes esteja em elevação nos EUA5,6,7.
Várias metodologias são aplicadas à diferenciação de membros do MAC, dentre as quais estão incluídas a soroaglutinação8, a hibridização DNA-RNAr com sondas comerciais (Accuprobe; GenProbe), a análise do polimorfismo de restrição do gene hsp65 (65-kDa heat 9 shock protein)9, a análise de ITS (16S-23S rDNA internal transcribed spacer)10, a RNAr 16S (RNA ribossômico 16S)11, a hsp6512 e, recentemente, a região V do gene rpoB (subunidade beta da RNA polimerase)13. Este último alvo foi proposto como alternativa para identificação de MAC em uma única etapa14, mas há poucos estudos utilizando esse marcador.
Assim, o objetivo do presente estudo é avaliar a aplicação deste marcador, bem como o RNAr 16S e hsp65, para distinguir espécies membros do MAC isoladas no Laboratório de Micobactérias da Seção de Bacteriologia e Micologia (SABMI) do Instituto Evandro Chagas (IEC), contribuindo desta forma para o aprimoramento da identificação desse grupo de micobactérias, ainda pouco conhecido na Região Amazônica.
MATERIAIS E MÉTODOS
CASUÍSTICA
Trata-se de um estudo retrospectivo de identificação de 60 amostras do MAC isoladas de espécimes clínicos pulmonares provenientes de 15 pacientes das cidades de Belém, Ananindeua, Parauapebas e Marabá, Estado do Pará. As amostras foram isoladas no Laboratório de Referência Regional de Micobactérias, SABMI/IEC, no período de janeiro de 2004 a dezembro de 2007. A infecção por MNT foi definida segundo os critérios da American Thoracic Society15 e Ministério da Saúde16, que consideraram de importância bacteriológica as MNT de origem pulmonar isoladas pelo menos duas vezes consecutivas de escarro ou lavado bronco-alveolar. Foram excluídos os pacientes com mais de uma espécie de micobactéria isolada dos espécimes clínicos. O projeto que deu origem a este trabalho foi aprovado pelo Comitê de Ética em Pesquisa com Seres Humanos do IEC, sob parecer no 0010/2008, CAAE: 0001.0.072.073-08.
PROCEDIMENTOS LABORATORIAIS
Cultivo micobacteriano
Todas as amostras foram processadas em cabine de segurança biológica classe II B2, na SABMI/IEC, no período de janeiro de 2004 a dezembro de 2007. Os espécimes clínicos foram semeados em meio sólido Löwenstein-Jensen (LJ-Difco, São Paulo, Brasil) e incubados a 35-37 °C na ausência de luz. A distinção presuntiva entre espécies do complexo M. tuberculosis e MNT foi obtida por meio da análise macro e microscópica da cultura, pesquisa do fator corda e teste de inibição de crescimento em meio LJ contendo 0,5 mg/mL de ácido para-nitrobenzóico (MP Biomedicals, Sólon, EUA)16.Todas as culturas com características sugestivas de MNT foram submetidas à identificação molecular.
Extração do DNA
Duas alçadas de crescimento micobacteriano em meio sólido foram suspensas em 300 µL de água MilliQ estéril. Essa suspensão foi transferida para microtubos contendo 600 µL de tampão de lise (Tris HCl pH 9,0 300 mM, EDTA 100 mM, sucrose 4,8%, SDS 1,25%), 600 µL de tampão de homogeneização (Tris HCl pH8 10 mM, NaCl 60 mM, EDTA pH8 10 mM, sucrose 5%), 15 µL de proteinase K (20 mg/mL) e incubada a 56 oC por 12 h. Os detritos celulares foram removidos com 600 µL de solução de fenolclorofórmio-álcool isoamil 25:24:1 (Invitrogen, São Paulo, Brasil). O DNA foi precipitado com 100 µL de acetato de sódio 3M, pH 4,8 (Sigma, São Paulo, Brasil) e 500 µL de isopropanol (Merck, Steinheim, Alemanha), centrifugado a 13.000 rpm por 10 min e tratado com 500 µL de etanol 70% (Merck, Steinheim, Alemanha). Após centrifugação a 13.000 rpm (16.845 xg), a 4 oC, o álcool foi removido por evaporação e o DNA precipitado foi ressuspenso em 100 µL de tampão TE 1x (Tris HCl pH8 10 mM, EDTA pH8 1 mM). A concentração da solução de DNA foi determinada usando o Kit Qubit Fluorometer (Invitrogen, São Paulo, Brasil), segundo as recomendações do fabricante.
Identificação genotípica
As amostras foram identificadas inicialmente pelo método de PRA-hsp65, segundo o qual uma sequência de 441 pb foi amplificada pela reação em cadeia da polimerase (PCR), a partir do gene alvo hsp65, utilizando os primers TB11 (5' ACCAACGATGGTGTGTCCAT 3') e TB12 (5' CTTGTCGAACCGCATACCCT 3')9. Os produtos da PCR foram digeridos separadamente com 10 U das enzimas BstEII (New England Biolabs, Ipswich, EUA) e HaeIII (Invitrogen, São Paulo, Brasil). Os fragmentos assim obtidos foram separados por eletroforese em gel de poliacrilamida 8% (Biosolve LTD, Valkenswaard, Holanda) contendo marcadores de peso molecular de 25 pb e 50 pb (Invitrogen, São Paulo, Brasil). Posteriormente, uma amostra de cada um dos 15 pacientes foi submetida ao sequenciamento dos genes RNAr 16S (RNAf/RNAr), hsp65 (HSPF3/HSPR4) e rpoB (MycoF/MycoR), de acordo com condições previamente descritas13,17,18.
Após a verificação dos produtos da PCR em gel de agarose Seakem LE 1% (Cambrex, Rockland, EUA), estes foram purificados usando o sistema de purificação S.N.A.P.™ Gel Purification Kit (Invitrogen, São Paulo, Brasil). Os produtos amplificados foram diretamente sequenciados com os primers forward e reverse usando o sistema BigDye Terminator v3.1 Cycle Sequencing Kit (Applied Biosystems, Foster City, EUA) e purificados com BigDye XTerminator Purification Kit (Applied Biosystems, Foster City, EUA), conforme as recomendações do fabricante. Posteriormente, foram submetidos ao analisador genético ABI3130 (Applied Biosystems, Tóquio, Japão).
Análises
Os perfis de PRA foram analisados por simples comparação visual com os marcadores de peso molecular, sendo interpretados com algoritmos publicados19,20,21,16 e PRASITE (http://app.chuv.ch/prasite/index.html). As sequências nucleotídicas obtidas para RNAr 16S, hsp65 e rpoB foram alinhadas com sequências de espécies relacionadas depositadas no GenBank (http://www.ncbi.nlm.nih.gov/BLAST/) usando o algoritmo de múltiplo alinhamento no programa BioEdit (v 7.0.9; TomHal [http://www.mbio.ncsu.edu/BioEdit/bioedit.html]). As árvores filogenéticas foram construídas usando o método de neighbor-joining, com modelo de correção Kimura-2-parâmetros no programa MEGA (v 4.0; Tamura, Dudley, Nei and Kumar [http://www.megasoftware.net/]), tomando M. fortuitum ATCC 13756 como grupo externo. A análise de bootstrap com mil replicações foi realizada. As sequências do GenBank utilizadas para comparações compreenderam: M. avium subsp. avium (RNAr 16S: EF521895, hsp65: AF547808, rpoB: EF521907), M. avium subsp. paratuberculosis (RNAr 16S: EF521896, hsp65: AF547809, rpoB: EF521906), M. avium subsp. silvaticum (RNAr 16S: EF521891, hsp65: AF547810, rpoB: EF521905), M. avium subsp. hominissuis (RNAr 16S: Ef521892, hsp65: EU239782, rpoB: EF521911), M. intracellulare (RNAr 16S: X52927, hsp65: EU239784, rpoB: EF521909), M. chimaera (RNAr 16S: EF521894, hsp65: EU239783, rpoB: EF521908) e M. colombiense (RNAr 16S: AM062764, hsp65: EU239785, rpoB: EF521910).
RESULTADOS
CARACTERIZAÇÃO POR PRA-hsp65
O PRA-hsp65 realizado nas amostras de MAC caracterizou-as em três diferentes perfis alélicos associados às espécies M. intracellulare e M. avium (Tabela 1).

ANÁLISE DO SEQUENCIAMENTO DO RNAr 16S, hsp65 E rpoB
As amostras apresentaram cinco genótipos distribuídos em dois grupos, cujos valores de similaridade variaram de 98,3% a 100% com sequências do RNAr 16S de cepas de MAC disponíveis no GenBank. Apesar das subespécies de M. avium não serem diferenciadas entre si, elas exibiram divergências de 2,1% com M. chimaera, 1,7% com M. intracellulare e 0,9% com M. colombiense. As amostras apresentaram cinco genótipos distribuídos em dois grupos, cujos valores de bootstrap indicavam baixo poder discriminatório para esse marcador. O grupo I apresentou sequências de táxons relacionados à espécie M. avium, que estavam subdivididos em três subgrupos, dos quais apenas um foi sustentado por valor de bootstrap de 95%, compreendendo aquele com subespécies de M. avium obtidas do GenBank. A diversidade nucleotídica para este subgrupo com relação às amostras isoladas de espécimes clínicos deste estudo foi atribuída às substituições nas posições 154 (C→T), 155 (A→T) e 169 (C→T). O grupo II comportou amostras estreitamente relacionadas ao M. intracellulare e ao M. chimaera, compartilhando similaridades de 99,5% a 100% (Figura 1A).
Na análise das sequências do hsp65 das amostras clínicas observaram-se nove genótipos com similaridades de 97% a 100%. As similaridades entre as amostras e as cepas de referência são apresentadas na tabela 2. Os táxons foram alocados em dois grupos, sendo o grupo I dividido em um subgrupo com M. avium sp., cujo valor de bootstrap foi de 93%, e um outro com amostras relacionadas ao M. colombiense. O grupo II apresentou amostras relacionadas ao M. intracellulare e M. chimaera, resultado semelhante ao obtido na análise de RNAr 16S (Figura 1B).
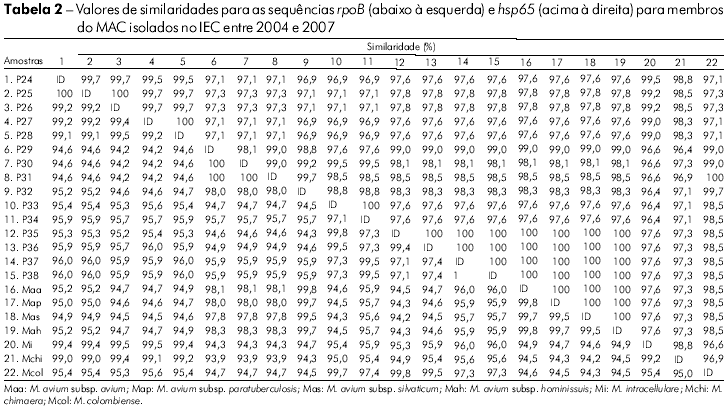
Na análise do gene rpoB, verificou-se similaridade de 95% a 100% entre as amostras, com divergências de 0,2% – 0,3% entre as subespécies M. avium e a clara distinção de M. avium dos demais membros do MAC (Tabela 2). Entretanto, observou-se mudança na topologia dos isolados, comparada com aquelas obtida para RNAr 16S e hsp65, sendo que amostras antes filogeneticamente relacionadas ao M. colombiense foram agrupadas com M. avium (P30 – P32) e vice-versa (P35– P38). Um achado importante foi a modificação dos membros nos agrupamentos: o grupo I foi formado por M. intracellulare com espécies relacionadas e M. colombiense, e o grupo II por subespécies de M. avium (Figura 1C). Onze genótipos foram encontrados na análise desse alvo molecular.
DISCUSSÃO
O estudo de membros do MAC tem se tornado importante pela prevalência em infecções disseminadas em pacientes com aids e pelo aumento do número de infecções em pacientes sem aids. Estudos têm sugerido que as infecções por MAC em pacientes com aids possam ser causadas por um limitado número de cepas relacionadas ao M. avium22,23. No entanto, as espécies e subespécies pertencentes ao MAC não são facilmente diferenciadas por testes fenotípicos, sendo os métodos moleculares, portanto, a melhor opção atualmente para identificação dessas micobactérias3.
O PRA-hsp65 é considerado um método simples, de fácil implantação nos laboratórios clínicos e que permite a identificação de uma ampla variedade de espécies detectadas na rotina laboratorial19,24,21. Entretanto, ele não possibilita a diferenciação de espécies recentemente descritas, como M. colombiense e M. chimaera, que compartilham perfis com as variantes alélicas M. avium I e M. intracellulare I, respectivamente19,25. Em virtude das diferentes significâncias clínicas entres as espécies de MAC, é necessário que se faça o esclarecimento taxonômico3,263,26.
O sequenciamento do RNAr 16S evidenciou a distribuição dos isolados em dois principais agrupamentos: um com táxons relacionados ao M. avium e outro ao M. intracellulare, porém os baixos valores de bootstrap encontrados na árvore demonstram o pequeno poder discriminatório para este alvo. Embora o RNAr 16S tenha um importante valor para a taxonomia das micobactérias, definindo a classificação das mesmas em grupos sistemáticos, a pequena variabilidade encontrada entre membros pertencentes ao MAC não permite distinção em nível de espécie1.
As sequências hsp65 apresentaram maior polimorfismo quando comparadas ao RNAr 16S. As similaridades variaram de 97% a 100%. No grupo I, que reuniu espécies relacionadas ao M. avium e M. colombiense, não foi possível realizar uma clara diferenciação entre os isolados. Ou seja, quando adotado o valor de cut-off acima de 97% para diferenciação de espécies por hsp65, proposto por McNabb et al12, não foi possível diferenciar M. avium de M. colombiense, cujas sequências de referência compartilharam similaridades de 98,5%. No entanto, ao considerarmos o maior escore de similaridade, conseguimos identificar 90% (9/10) dos isolados deste grupo, representados por M. colombiense (P30 – P34) e M. avium (P35 – P38), excluindo apenas P29, que exibiu 99% de similaridade com ambas as espécies. Esse achado confirma a estreita relação entre M. avium e M. colombiense, como demonstrado por Murcia et al25. Os isolados clínicos P24 a P28 exibiram maior similaridade com M. intracellulare, embora compartilhassem 98,3% – 98,8% de similaridade com M. chimaera. Tal dado demonstra a necessidade de busca por novos alvos moleculares, capazes de distinguir uma ampla variedade de espécies, incluindo aquelas que exibem estreitas relações filogenéticas.
Um segmento da região V do gene rpoB, inicialmente usado para identificação de micobactérias de crescimento rápido13 e recentemente proposto para identificação de espécies do MAC14, foi avaliado neste estudo. Na análise das sequências foram observadas incongruências de resultados quando comparados ao sequenciamento dos genes RNAr 16S e hsp65. Como observado na figura 2A, M. colombiense mostrou-se estreitamente relacionado ao M. avium, o que também se estendeu à análise de hsp65 (Figura 2B). Este achado é equivalente ao descrito por Murcia et al25. No entanto, na análise do rpoB verificou-se que M. colombiense se apresentou no grupo relacionado ao M. chimaera e M. intracellulare, ocorrendo, portanto, mudança na topologia da árvore. Além disso, houve uma inversão na alocação dos isolados dentro dos grupos, sendo que alguns isolados filogeneticamente relacionados ao M. colombiense foram reunidos com M. avium e vice-versa.
A disposição de táxons na árvore de rpoB deste estudo também foi observada em trabalho prévio que demonstrou a utilidade deste marcador para identificação de MAC, segundo o qual divergência de 0,7% possibilitou a diferenciação de 93% dos isolados clínicos14.
Há um consenso a respeito de que o conhecimento preciso de isolados de MAC envolvidos em infecções possa fornecer importantes dados sobre a heterogeneidade clínica e patogenicidade destes membros, bem como sobre o estabelecimento de terapia baseada na diversidade genotípica27,28,3.
CONCLUSÃO
Embora o gene rpoB tenha sido proposto como marcador molecular adicional para diferenciação de micobactérias, inclusive de isolados pertencentes ao MAC, os achados deste estudo demonstraram que os resultados da análise do gene rpoB foram discordantes daqueles encontrados para as sequências do RNAr 16S e hsp65, sugerindo que diferentes forças evolucionárias atuam sobre marcadores individuais. Diante do exposto, alertamos que a seleção de alvos moleculares para fins taxonômicos deve ser baseada em marcadores conservados, onde se observe a manutenção da relação das espécies com seus grupos sistemáticos conhecidos e que exibam maior poder discriminatório que o gene do RNAr 16S. Adicionalmente, recomendamos que mais de um marcador, incluindo o RNAr 16S, seja avaliado para a identificação das espécies micobacterianas.
REFERÊNCIAS
1 Tortoli E. Impact of genotypic studies on mycobacterial taxonomy: the new mycobacteria of the 1990. Clin Microbiol Rev. 2003 Apr;16(2):319-54. [ Links ]
2 Smole SC, McAleese F, Ngampasutadol J, von Reyn CF, Arbeit RD. Clinical and epidemiological correlates of genotypes within the Mycobacterium avium complex defined by restriction and sequence analysis of hsp65. J Clin Microbiol. 2002 Sep;40(9):3374-80. DOI:10.1128/JCM.40.9.3374-3380.2002 [ Links ]
3 Tortoli E, Rindi L, Garcia MJ, Chiaradonna P, Dei R, Garzelli C, et al. Proposal to elevate the genetic variant MAC-A, included in the Mycobacterium avium complex, to species rank as Mycobacterium chimaera sp. nov. Int J Syst Evol Microbiol. 2004;54:1277-85. DOI:10.1099/ijs.0.02777-0 [ Links ]
4 von Reyn CF, Arbeit RD, Horsburgh CR, Ristola MA, Waddell RD, Tvaroha SM, et al. Sources of disseminated Mycobacterium avium infection in AIDS. J Infect. 2002 Apr;44(3):166-70. [ Links ]
5 Cappelluti E, Fraire AE, Schaefer OP. A case of 'hot tub lung' due to Mycobacterium avium complex in an immunocompetent host. Arch Intern Med. 2003 Apr;163(7):845-8. [ Links ]
6 Lumb R, Stapledon R, Scroop A, Bond P, Cunliffe D, Goodwin A, et al. Investigation of spa pools associated with lung disorders caused by Mycobacterium avium complex in immunocompetent adults. Appl Environ Microbiol. 2004;70(8):4906-10.
7 Myojo M, Fujiuchi S, Matsumoto H, Yamazaki Y, Takahashi M, Satoh K, et al. Disseminated Mycobacterium avium complex (DMAC) in an immunocompetent adult. Int J Tuberc Lung Dis. 2003 May;7(5):498-501. [ Links ]
8 Tsang AY, Denner JC, Brennan PJ, McClatchy JK. Clinical and epidemiological importance of typing of Mycobacterium avium complex isolates. J Clin Microbiol. 1992 Feb;30(2)479-84. [ Links ]
9 Telenti A, Marchesi F, Balz M, Bally F, Bottger EC, Bodmer T. Rapid identification of mycobacteria to the species level by polymerase chain reaction, restriction enzyme analysis. J Clin Microbiol. 1993 Feb;31:175-8. [ Links ]
10 Frothingham R, Wilson KH. Sequence-based differentiation of strains in the Mycobacterium avium complex. J Bacteriol. 1993 May;175(10):2818-25. [ Links ]
11 Turenne CY, Tschetter L, Wolfe J, Kabani A. Necessity of quality-controlled 16S rRNA gene sequence databases: identifying nontuberculous Mycobacterium species. J Clin Microbiol. 2001;39(10):3637-48. DOI:10.1128/JCM.39.10.3638-3648.2001 [ Links ]
12 McNabb A, Eisler D, Adie K, Amos M, Rodrigues M, Stephens G, et al. Assessment of partial sequencing of the 65-kilodalton heat shock protein gene (hsp65) for routine identification of Mycobacterium species isolated from clinical sources. J Clin Microbiol. 2004 Jul;42(7):3000-11. DOI:10.1128/JCM.42.7.3000-3011.2004 [ Links ]
13 Adékambi T, Colson P, Drancourt M. rpoB-based identification of nonpigmented and late-pigmenting rapidly growing mycobacteria. J Clin Microbiol. 2003 Dec;41(12):5699-708. DOI: 10.1128/JCM.41.12.5699-5708.2003 [ Links ]
14 Ben Salah I, Adékambi T, Raoult D, Drancourt M. rpoB sequence-based identification of Mycobacterium avium complex species. Microbiology. 2008 Dec;154(12):3715-23. DOI:10.1099/mic.0.2008/020164-0 [ Links ]
15 Griffith DE, Aksamit T, Brown-Elliott BA, Catanzaro A, Daley C, Gordin F, et al. An official ATS/IDSA statement: diagnosis, treatment, and prevention of nontuberculous mycobactecterial diseases. Am J Respir Crit Care Med. 2007;175:367-416. DOI:10.1164/RCCM.200604-571ST [ Links ]
16 Secretaria de Vigilância em Saúde. Manual Nacional de Vigilância Laboratorial da Tuberculose e outras Micobactérias. Brasília: Ministério da Saúde; 2008.
17 Kim H, Kim SH, Shim TS, Kim MN, Bai GH, Park YG, et al. PCR restriction fragment length polymorphism analysis (PRA)-algorithm targeting 644 bp Heat Shock Protein 65 (hsp65) gene for differentiation of Mycobacterium spp. J Microbiol Methods. 2005 Aug;62(2):199-209. DOI:10.1016/J.MIMET.2005.02.010 [ Links ]
18 Shin S, Kim EC, Yoon JH. Identification of nontuberculous mycobacteria by sequence analysis of the 16S ribosomal RNA, the heat-shock protein 65 and the RNA polymerase b-subunit genes. Korean J Lab Med. 2006 Jun;26(3):153-60. [ Links ]
19 Brunello F, Ligozzi M, Cristelli E, Bonora S, Tortoli E, Fontana R. Identification of 54 mycobacterial species by PCR-restriction fragment length polymorphism analysis of the hsp65 gene. J Clin Microbiol. 2001 Aug;39(8):2799-806. DOI:10.1128/JCM.39.8.2799-2806.2001 [ Links ]
20 Devallois A, Goh KS, Rastogi N. Rapid identification of mycobacteria to species level by PCR-restriction fragment length polymorphism analysis of the hsp65 gene and proposition of an algorithm to differentiate 34 mycobacterial species. J Clin Microbiol. 1997 Nov;35(11):2969-73. [ Links ]
21 Leão SC, Bernadelli A, Cataldi A, Zumarraga M, Robledo J, Realpe T, et al. Multicenter evaluation of mycobacteria identification by PCR restriction enzyme analysis in laboratories from Latin America and the Caribbean. J Microbiol Methods. 2005 May;61(2):193-9. DOI:10.1016/J.MIMET.2004.11.015 [ Links ]
22 Ritacco V, Kremer K, Van der Laan T, Pijnenburg JE, Haas PE, van Soolingen D. Use of IS901 and IS1245 in RFLP typing of Mycobacterium avium complex: relatedness among serovar reference strains, human and animal isolates. Int J Tuberc Lung Dis. 1998 Mar;2(3):242-51. [ Links ]
23 Yakrus MA, Good RC. Geographic distribution, frequency, and specimen source of Mycobacterium avium complex serotypes isolated from patients with acquired immunodeficiency syndrome. J Clin Microbiol. 1990 May;28(5):926-9.
24 Chimara E, Ferrazoli L, Ueky SY, Martins MC, Durham AM, Arbeit RD, et al. Reliable identification of mycobacterial species by PCR-restriction enzyme analysis (PRA)-hsp65 in a reference laboratory and elaboration of a sequence-based extended algorithm of PRA-hsp65 patterns. BMC Microbiology. 2008;8(48):1-12. DOI:10.1186/1471-2180-8-48 [ Links ]
25 Murcia MI, Tortoli E, Menendez MC, Palenque E, Garcia MJ. Mycobacterium colombiense sp. nov., a novel member of the Mycobacterium avium complex and description of MAC-X as a new ITS genetic variant. Int J Syst Evol Microbiol. 2006;56(9):2049-54. DOI:10.1099/ijs.0.64190-0 [ Links ]
26 Turenne CY, Wallace R Jr, Behr MA. Mycobacterium avium in the postgenomic era. Clin Microbiol Rev. 2007 Apr;20(2):205-29. DOI:10.1128/CMR.00036-06 [ Links ]
27 Frothingham R, Wilson KH. Molecular phylogeny of the Mycobacterium avium complex demonstrates clinically meaningful divisions. J Infect Dis. 1994 Feb;169(2):305-12. [ Links ]
28 Maekura R, Okuda Y, Hirotani A, Kitada S, Hiraga T, Yoshimura K, et al. Clinical and prognostic importance of serotyping Mycobacterium avium-Mycobacterium intracellulare complex isolates in human immunodeficiency virus-negative patients. J Clin Microbiol. 2005 Jul;43(7):3150-8. DOI:10.1128/JCM.43.7.3150-3158.2005 [ Links ]
 Correspondência / Correspondence/ Correspondencia:
Correspondência / Correspondence/ Correspondencia:
Ana Roberta Fusco da Costa
Instituto Evandro Chagas,
Seção de Bacteriologia e Micologia,
Laboratório de Biologia Molecular
Rodovia BR 316, km 07, s/no. Bairro: Levilândia
CEP:67030-000 Ananindeua-Pará-Brasil
E-mail:anacosta@iec.pa.gov.br/robertafusco@gmail.com
Tel./Fax: +55 (91) 3214-2116 / +55 (91) 3214-2129
Recebido em / Received / Recibido en: 31/7/2010
Aceito em / Accepted / Aceito en: 24/8/2010










 texto em
texto em