Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Pan-Amazônica de Saúde
Print version ISSN 2176-6215On-line version ISSN 2176-6223
Rev Pan-Amaz Saude vol.1 no.3 Ananindeua Sept. 2010
http://dx.doi.org/10.5123/S2176-62232010000300005
ARTÍCULO ORIGINAL
Identificación genotípica de miembros del complejo Mycobacterium avium aislados de infecciones pulmonares en el Estado de Pará, Brasil
Ana Roberta Fusco da CostaI; Maria Luíza LopesI; Jeann Ricardo da Costa BahiaII; Emilyn da Costa ConceiçãoII; Karla Valéria Batista LimaI
ISeção de Bacteriologia e Micologia,
Instituto Evandro Chagas/SVS/MS, Ananindeua, Pará, Brasil
IIInstituto
Evandro Chagas/SVS/MS, Ananindeua, Pará, Brasil. Universidade Federal
do Pará, Belém, Pará, Brasil
Endereço para correspondência
Correspondence
Dirección para correspondencia
Título original: Identificação genotípica de membros do complexo Mycobacterium avium isolados de infecções pulmonares no Estado do Pará, Brasil. Traducido por: Lota Moncada
RESUMEN
INTRODUCCIÓN: El complejo Mycobacterium avium (MAC) comprende micobacterias de crecimiento lento, naturalmente
encontradas en el medio ambiente, capaces de causar infecciones en diversas
especies de seres vivos, incluyendo aves, porcinos y humanos. Tales infecciones
pueden ser asintomáticas, clínicamente significantes y,
en algunos casos, fatales. Sin embargo, existe la necesidad de poner a
disposición técnicas
que ofrezcan una identificación conclusiva de bacterias estrechamente
relacionadas. Las técnicas de biología molecular se presentan
como herramientas prometedoras para una identificación más
precisa.
MATERIALES Y MÉTODOS: En este trabajo fueron evaluados
los marcadores moleculares RNAr 16S, hsp65 y rpoB aplicados a la distinción
de miembros del MAC, aislados en el Laboratorio de Micobacterias del Instituto
Evandro Chagas.
RESULTADOS: Muestras del MAC colectadas en 15 pacientes
fueron previamente evaluadas por el método de análisis de polimorfismo
de fragmentos de restricción del gen hsp65 (PRA-hsp65), que
suministró tres
perfiles diferentes: (I) BstEII: 235/115/100, HaeIII: 145/130/60;
(II) BstEII:
235/210, HaeIII: 130/105; y (III) BstEII: 235/210, HaeIII:
145/130. Se construyeron
árboles filogenéticos luego del análisis del RNAr 16S,
en el que las muestras fueron distribuidas en dos grupos, semejante al encontrado
en el análisis de hsp65. Sin embargo, los resultados del rpoB fueron
discordantes de los otros árboles, debido a la modificación
de la topología.
CONCLUSIÓN: Los hallazgos de este
estudio sugieren que distintas fuerzas evolutivas pueden estar actuando
sobre el gen rpoB, y así, es necesario tener precaución al
establecer esa meta para fines taxonómicos. Adicionalmente, se recomienda
que más
de un marcador, incluyendo el RNAr 16S, sea evaluado para la identificación
micobacteriana.
Palabras clave: Complejo Mycobacterium avium; Polimorfismo de Longitud del Fragmento de Restricción; Análisis de Secuencia de ADN; Filogenia.
INTRODUCTION
El complejo Mycobacterium avium (MAC), junto con otras especies mico bacterianas atípicas, denominadas de micobacterias no tuberculosas (MNT), comprende micobacterias de crecimiento lento, naturalmente encontradas en el medio ambiente1, capaces de causar infecciones en diversas especies de seres vivos, incluyendo aves, porcinos y humanos, pudiendo presentarse como asintomáticas, clínicamente significantes y, en algunos casos, fatales2. Este complejo está constituido por las especies M. avium, M. intracellulare, M. colombiense y M. chimaera3,1.
El MAC representa un importante grupo de micobacterias asociado a varias patologías humanas, especialmente a las infecciones diseminadas en pacientes con SIDA4. Además de esas, recientes evidencias sugieren que la tasa de infección en pacientes inmunocompetentes esté en elevación en los EUA5,6,7.
Varias metodologías se aplican a la diferenciación de miembros del MAC, entre las que están incluidas la seroaglutinación8. La hibridación ADN-ARNr con sondas comerciales (Accuprobe; GenProbe), el análisis del polimorfismo de restricción del gen hsp65 (65-kDa heat shock protein)9, el análisis de ITS (16S-23S rADN internal transcribed spacer)10, el ARNr 16S (ARN ribosómico 16S)11, el hsp6512 y, recientemente, la región V del gen rpoB (subunidad beta del ARN polimerasa)13. Este último blanco fue propuesto como alternativa para identificación de MAC en una única etapa14, pero hay pocos estudios utilizando ese marcador.
Así, el objetivo del presente estudio es evaluar la aplicación de este marcador, bien como el ARNr 16S y hsp65, para distinguir especies miembros del MAC aisladas en el Laboratorio de Micobacterias de la Sección de Bacteriología y Micología (SABMI) del Instituto Evandro Chagas (IEC), contribuyendo de esta forma para el perfeccionamiento de la identificación de este grupo de micobacterias, todavía poco conocido en la Región Amazónica.
MATERIALES Y MÉTODOS
CASUÍSTICA
Se trata de un estudio retrospectivo de identificación de 60 muestras del MAC aisladas de especímenes clínicos pulmonares provenientes de 15 pacientes de las ciudades de Belém, Ananindeua, Parauapebas y Marabá, Estado de Pará. Las muestras fueron aisladas en el Laboratorio de Referencia Regional de Micobacterias, SABMI/IEC, en el período de enero de 2004 a diciembre de 2007. La infección por MNT se definió según los criterios de la American Thoracic Society15 y el Ministerio de Salud16, que consideraron de importancia bacteriológica las MNT de origen pulmonar aisladas por lo menos dos veces consecutivas de esputo o lavado broncoalveolar. Fueron excluidos los pacientes con más de una especie de micobacteria aislada de los especímenes clínicos.
El proyecto que dio origen a este trabajo fue aprobado por el Comité de Ética en Investigación con Seres Humanos del IEC, bajo el parecer no 0010/2008, CAAE: 0001.0.072.073-08.
PROCEDIMIENTOS DE LABORATORIO
Cultivo micobacteriano
Todas las muestras fueron procesadas en cabina de seguridad biológica clase II B2, en la SABMI/IEC, en el período de enero de 2004 a diciembre de 2007.
Los especímenes clínicos fueron sembrados en medio sólido Löwenstein-Jensen (LJ-Difco, São Paulo, Brasil) e incubados a 35-37o C en ausencia de luz. La distinción presuntiva entre especies del complejo M. tuberculosis y MNT se obtuvo por medio del análisis macro y microscópico del cultivo, investigación del factor corda y test de inhibición de crecimiento en medio LJ conteniendo 0,5 mg/mL de ácido paranitrobenzoico (MP Biomedicals, Solon, EUA)16. Todos los cultivos con características sugestivas de MNT fueron sometidas a identificación molecular.
Extracción del ADN
Dos cultivos de crecimiento micobacteriano en medio sólido fueron suspendidas en 300 µL de agua MilliQ estéril. Esa suspensión se transfirió a microtubos conteniendo 600 µL de tampón de lisis (Tris HCl pH 9,0 300 mM, EDTA 100 mM, sucrose 4,8%, SDS 1,25%), 600 µL de solución homogeneizadora (Tris HCl pH8 10 mM, NaCl 60 mM, EDTA pH8 10 mM, sucrose 5%), 15 µL de proteinasa K (20 mg/mL) y incubada en 56o C por 12 h. Los detritos celulares se removieron con 600 µL de solución de fenol-cloroformo-alcohol isoamílico 25:24:1 (Invitrogen, São Paulo, Brasil). El DNA fue precipitado con 100 µL de acetato de sodio 3M, pH 4,8 (Sigma, São Paulo, Brasil) y 500 µL centrifugado a 13.000 rpm por 10 min y tratado con 500 µL de etanol 70% (Merck, Steinheim, Alemanha). Luego de la centrifugación a 13.000 rpm (16.845 xg), a 4o C, el alcohol se removió por evaporación y el ADN precipitado se resuspendió en 100 µL de tampón TE 1x (Tris HCl pH8 10 mM, EDTA pH8 1 mM). La concentración de la solución de ADN se determina usando el Kit Qubit Fluorometer (Invitrogen, São Paulo, Brasil), según las recomendaciones del fabricante.
Identificación genotípica
Las muestras fueron identificadas inicialmente por el método de PRA-hsp65, según el cual una secuencia de 441 pb fue amplificada por la reacción en cadena de la polimerasa (PCR), a partir del gen objetivo hsp65, utilizando los primers TB11 (5' ACCAACGATG GTGTGTCCAT 3') y TB12 (5' CTTGTCGAACCGCATACC CT 3')9. Los productos de PCR fueron digeridos separadamente con 10 U de las enzimas 8stEII (New England Biolabs, Ipswich, EUA) y HaeIII (Invitrogen, São Paulo, Brasil). Los fragmentos así obtenidos fueron separados por electroforesis en gel de poliacrilamida 8% (Biosolve LTD, Valkenswaard, Holanda) conteniendo marcadores de peso molecular de 25 pb y 50 pb (Invitrogen, São Paulo, Brasil). Posteriormente, una muestra de cada uno de los 15 pacientes fue sometida a la secuenciación de los genes ARNr 16S (RNAF/RNAR), hsp65 (HSPF3/HSPR4) y rpoB (MycoF/MycoR), de acuerdo a condiciones previamente descritas13,17,18.
Luego de la verificación de los productos de la PCR en gel de agarosa Seakem LE 1% (Cambrex, Rockland, EUA), estos fueron purificados usando el sistema de purificación S.N.A.P™ Gel Purification Kit (Invitrogen, São Paulo, Brasil). Los productos amplificados fueron secuenciados directamente con los primers forward y reverse usando el sistema BigDye Terminator v3.1 Cycle Sequencing Kit (Applied Biosystems, Foster City, EUA) y purificados con BigDye XTerminator Purification Kit (Applied Biosystems, Foster City, EUA), conforme las recomendaciones del fabricante. Posteriormente, fueron sometidos al analizador genético ABI3130 (Applied Biosystems, Tokio, Japón).
Análisis
Los perfiles de PRA fueron analizados por simple comparación visual con los marcadores de peso molecular, siendo interpretados con algoritmos publicados19,20,21,16 y PRASITE (http://app.chuv.ch/prasite/index.html). Las secuencias de nucleótidos obtenidas para RNAr 16S, hsp65 y rpoB fueron alineadas con secuencias de especies relacionadas depositadas en el GenBank (http://www.ncbi.nlm.nih.gov/BLAST/) usando el algoritmo de múltiple alineamiento en el programa BioEdit (versión 7.0.9; Tom Hall [http://www.mbio.ncsu.edu/BioEdit/bioedit.html]). Los árboles filogenéticos se construyeron usando el método de neighbor-joining, con el modelo de corrección Kimura-2-parámetros en el programa MEGA (versión 4.0; Tamura, Dudley, Nei and Kumar [http://www.megasoftware.net/]). El análisis de bootstrap con mil replicaciones se realizó tomando M. fortuitum ATCC 13756 como grupo externo. Las secuencias del GenBank utilizadas para comparaciones comprendieron: M. avium subsp. avium (ARNr 16S: EF521895, hsp65: AF547808, rpoB: EF521907), M. avium subsp. paratuberculosis (ARNr 16S: EF521896, hsp65: AF547809, rpoB: EF521906), M. avium subsp. silvaticum (ARNr 16S: EF521891, hsp65: AF547810, rpoB: EF521905), M. avium subsp. hominissuis (ARNr 16S: EF521892, hsp65: EU239782, rpoB: EF521911), M. intracellulare (ARNr 16S: X52927, hsp65: EU239784,rpoB: EF521909), M. chimaera (ARNr 16S: EF521894,hsp65: EU239783, rpoB: EF521908) y M. colombiense (ARNr 16S: AM062764, hsp65: EU239785, rpoB: EF521910).
RESULTADOS
CARACTERIZACIÓN POR PRA-hsp65
El PRA-hsp65 realizado en las muestras de MAC se caracterizó en tres diferentes perfiles alélicos asociados a las especies M. intracellulare y M. avium (Tabla 1).

ANÁLISIS DE LA SECUENCIACIÓN DEL ARNr 16S, hsp65 y rpoB
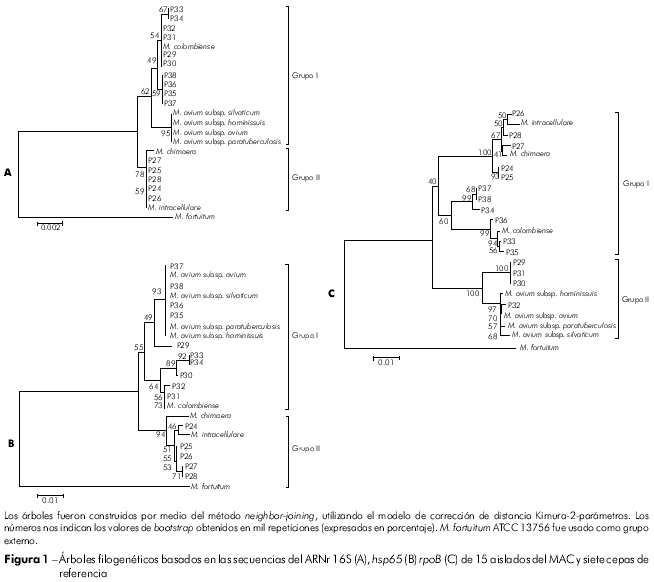
Las muestras presentaron cinco genotipos distribuidos en dos grupos, cuyos valores de similitud variaron de 98,3% a 100% con secuencias del ARNr 16S de cepas de MAC disponibles en el GenBank. A pesar de las subespecies de M. avium no ser diferenciadas entre si, ellas exhibieron divergencias de 2,1% con M. chimaera, 1,7% con M. intracellulare y 0,9% con M. colombiense. Las muestras presentaron cinco genotipos distribuidos en dos grupos, cuyos valores de bootstrap indicaban bajo poder discriminatorio para este marcador. El grupo I presentó secuencias de taxones relacionados a la especie M. avium, que estaban subdivididos en tres subgrupos, de los cuales apenas uno fue sostenido por valor de bootstrap de 95%, comprendiendo los con subespecies de M. avium obtenidas del GenBank. La diversidad nucleotídica para este subgrupo con relación a las muestras aisladas de especímenes clínicos de este estudio se atribuye a las sustituciones en las posiciones 154 (C →T), 155 (A →T) e 169 (C →T). El grupo II comprendió muestras estrechamente relacionadas al M. intracellulare y al M. chimaera, compartiendo similitudes de 99,5% a 100% (Figura 1A).
En el análisis de las secuencias del hsp65 de las muestras clínicas se observaron nueve genotipos con similitudes de 97% a 100%. Las similitudes entre las muestras y las cepas de referencia se presentan en la tabla 2. Los taxones fueron distribuidos en dos grupos, siendo el grupo I dividido en un subgrupo con M. avium sp., cuyo valor de bootstrap fue de 93%, y otro con muestras relacionadas al M. colombiense. El grupo II presentó muestras relacionadas al M. intracellulare y M. chimaera, resultado semejante al obtenido en el análisis de ARNr 16S (Figura 1B).
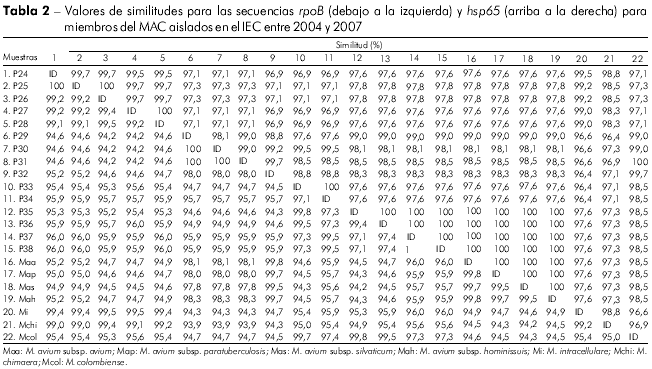
En el análisis del gen rpoB, se verificó similitud de 95% a 100% entre las muestras, con divergencias de 0,2% - 0,3% entre las subespecies M. avium y la clara distinción de M. avium de los demás miembros del MAC (Tabla 2). Sin embargo, se observó cambio en la topología de los aislados, comparada con la obtenida para ARNr 16S y hsp65, siendo que muestras antes filogenéticamente relacionadas al M. colombiense fueron agrupadas con M. avium (P30 - P32) y viceversa (P35 - P38). Un hallazgo importante fue la modificación en los agrupamientos: el grupo I fue formado por M. intracellulare con especies rel acionadas y M. colombiense, y el grupo II por subespecies de M. avium (Figura 1C). Se encontraron 11 genotipos en el análisis de ese objetivo molecular.
DISCUSIÓN
El estudio de miembros del MAC se ha tornado importante por la prevalencia en infecciones diseminadas en pacientes con SIDA y por el aumento del número de infecciones en pacientes sin SIDA. Estudios ha sugerido que las infecciones por MAC en pacientes con SIDA puedan ser causadas por un limitado número de cepas relacionadas al M. avium22,23. Sin embargo, las especies y subespecies que pertenecen al MAC no se diferencian fácilmente por análisis fenotípicos, siendo los métodos moleculares, por lo tanto, la mejor opción actualmente para la identificación de esas micobacterias3.
El PRA-hsp65 se considera un método simple, de fácil implantación en los laboratorios clínicos y que permite la identificación de una amplia variedad de especies detectadas en la rutina laboratorial19,24,21. Sin embargo, no posibilita la diferenciación de especies recientemente descritas, como M. colombiense y M. chimaera, que comparten perfiles con las variantes alélicas M. avium I y M. intracellulare I, respectivamente19, 25. En virtud de las diferentes significaciones clínicas entres las especies de MAC, es necesario que se realice la aclaración taxonómica3,26.
La secuenciación del ARNr 16S evidenció la distribución de los aislados en dos principales agrupamientos: uno con taxones relacionados al M. avium y otro al M. intracellulare, pero los bajos valores de bootstrap encontrados en el árbol demuestran el pequeño poder discriminatorio para este objetivo. Aunque el ARNr 16S tenga un importante valor para la taxonomía de las micobacterias, definiendo la clasificación de las mismas en grupos sistemáticos, la pequeña variabilidad encontrada entre miembros pertenecientes al MAC no permite distinción a nivel de especie1.
Las secuencias hsp65 presentaron un mayor polimorfismo cuando comparadas al ARNr 16S. Las similitudes variaron de 97% a 100%. En el grupo I, que reunió especies relacionadas al M. avium y M. colombiense, no fue posible realizar una diferenciación clara entre los aislados. O sea, cuando se adoptó el valor de cut-off superior a 97% para diferenciación de especies por hsp65, propuesto por McNabb et al12, no fue posible diferenciar M. avium de M. colombiense, cuyas secuencias de referencia compartieron similitudes de 98,5%. Sin embargo, al considerar la mayor puntuación de similitud, conseguimos identificar un 90% (9/10) de los aislados de este grupo, representados por M. colombiense (P30 - P34) y M. avium (P35 - P38), excluyendo apenas P29, que exhibió un 99% de similitud con ambas especies. Este hallazgo confirma la estrecha relación entre M. avium y M. colombiense, como demostrado por Murcia et al25. Los aislados clínicos P24 a P28 exhibieron mayor similitud con M. intracellulare, aunque compartieran un 98,3% - 98,8% de similitud con M. chimaera. Tal dato demuestra la necesidad de búsqueda de nuevos objetivos moleculares, capaces de distinguir una amplia variedad de especies, incluyendo aquellas que exhiben estrechas relaciones filogenéticas.
Un segmento de la región V del gen rpoB, inicialmente usado para identificación de micobacterias de crecimiento rápido13 y recientemente propuesto para identificación de especies del MAC14, fue también evaluado en este estudio. En el análisis de las secuencias se observaron incongruencias de resultados cuando comparados a la secuenciación de los genes ARNr 16S y hsp65. Como observado en el árbol 2A, M. colombiense se mostró estrechamente relacionado al M. avium, que también se extendió al análisis de hsp65 (Figura 2B). Este hallazgo es equivalente al descrito por Murcia et al25. Sin embargo, en el análisis del rpoB se comprobó que M. colombiense se presentó en el grupo relacionado al M. chimaera y M. intracellulare, sucediendo, por lo tanto, un cambio en la topología del árbol. Además de eso, hubo una inversión e la ubicación de los aislados dentro de los grupos, siendo que algunos aislados filogenéticamente relacionados al M. colombiense fueron reunidos con M. avium y viceversa.
La disposición de taxones en el árbol de rpoB de este estudio también fue observada en trabajo previo, lo que demostró la utilidad de este marcador para la identificación del MAC, según el cual, una divergencia de 0,7% posibilitó la diferenciación de 93% de los aislados clínicos14.
Hay consenso a respecto de que el conocimiento preciso de aislados de MAC involucrados en infecciones puede suministrar importantes datos sobre la heterogeneidad clínica y la patogenicidad de estos miembros, así como sobre el establecimiento de terapia basada en la diversidad genotípica27,28,3.
CONCLUSIÓN
Aunque el gen rpoB haya sido propuesto como marcador molecular adicional para diferenciación de micobacterias, inclusive de aislados pertenecientes al MAC, los hallazgos de este estudio demostraron que los resultados del análisis del gen rpoB fueron discordantes de los encontrados para las secuencias del ARNr 16S y hsp65, sugiriendo que diferentes fuerzas evolutivas actúan sobre marcadores individuales. Delante de lo expuesto, alertamos sobre que, la selección de objetivos moleculares para fines taxonómicos debe estar basada en marcadores conservados, en donde se observe la manutención de la relación de las especies con sus grupos sistemáticos conocidos y que exhiban mayor poder discriminatorio que el gen del ARNr 16S. Adicionalmente, recomendamos que más de un marcador, incluyendo el ARNr 16S, sea evaluado para la identificación de las especies micobacterianas.
REFERENCIAS
1 Tortoli E. Impact of genotypic studies on mycobacterial taxonomy: the new mycobacteria of the 1990. Clin Microbiol Rev. 2003 Apr;16(2):319-54. [ Links ]
2 Smole SC, McAleese F, Ngampasutadol J, von Reyn CF, Arbeit RD. Clinical and epidemiological correlates of genotypes within the Mycobacterium avium complex defined by restriction and sequence analysis of hsp65. J Clin Microbiol. 2002 Sep;40(9):3374-80. DOI:10.1128/JCM.40.9.3374-3380.2002 [ Links ]
3 Tortoli E, Rindi L, Garcia MJ, Chiaradonna P, Dei R, Garzelli C, et al. Proposal to elevate the genetic variant MAC-A, included in the Mycobacterium avium complex, to species rank as Mycobacterium chimaera sp. nov. Int J Syst Evol Microbiol. 2004;54:1277-85. DOI:10.1099/ijs.0.02777-0 [ Links ]
4 von Reyn CF, Arbeit RD, Horsburgh CR, Ristola MA, Waddell RD, Tvaroha SM, et al. Sources of disseminated Mycobacterium avium infection in AIDS. J Infect. 2002 Apr;44(3):166-70. [ Links ]
5 Cappelluti E, Fraire AE, Schaefer OP. A case of 'hot tub lung' due to Mycobacterium avium complex in an immunocompetent host. Arch Intern Med. 2003 Apr;163(7):845-8. [ Links ]
6 Lumb R, Stapledon R, Scroop A, Bond P, Cunliffe D, Goodwin A, et al. Investigation of spa pools associated with lung disorders caused by Mycobacterium avium complex in immunocompetent adults. Appl Environ Microbiol. 2004;70(8):4906-10.
7 Myojo M, Fujiuchi S, Matsumoto H, Yamazaki Y, Takahashi M, Satoh K, et al. Disseminated Mycobacterium avium complex (DMAC) in an immunocompetent adult. Int J Tuberc Lung Dis. 2003 May;7(5):498-501. [ Links ]
8 Tsang AY, Denner JC, Brennan PJ, McClatchy JK. Clinical and epidemiological importance of typing of Mycobacterium avium complex isolates. J Clin Microbiol. 1992 Feb;30(2)479-84. [ Links ]
9 Telenti A, Marchesi F, Balz M, Bally F, Bottger EC, Bodmer T. Rapid identification of mycobacteria to the species level by polymerase chain reaction, restriction enzyme analysis. J Clin Microbiol. 1993 Feb;31:175-8. [ Links ]
10 Frothingham R, Wilson KH. Sequence-based differentiation of strains in the Mycobacterium avium complex. J Bacteriol. 1993 May;175(10):2818-25. [ Links ]
11 Turenne CY, Tschetter L, Wolfe J, Kabani A. Necessity of quality-controlled 16S rRNA gene sequence databases: identifying nontuberculous Mycobacterium species. J Clin Microbiol. 2001;39(10):3637-48. DOI:10.1128/JCM.39.10.3638-3648.2001 [ Links ]
12 McNabb A, Eisler D, Adie K, Amos M, Rodrigues M, Stephens G, et al. Assessment of partial sequencing of the 65-kilodalton heat shock protein gene (hsp65) for routine identification of Mycobacterium species isolated from clinical sources. J Clin Microbiol. 2004 Jul;42(7):3000-11. DOI:10.1128/JCM.42.7.3000-3011.2004 [ Links ]
13 Adékambi T, Colson P, Drancourt M. rpoB-based identification of nonpigmented and late-pigmenting rapidly growing mycobacteria. J Clin Microbiol. 2003 Dec;41(12):5699-708. DOI: 10.1128/JCM.41.12.5699-5708.2003 [ Links ]
14 Ben Salah I, Adékambi T, Raoult D, Drancourt M. rpoB sequence-based identification of Mycobacterium avium complex species. Microbiology. 2008 Dec;154(12):3715-23. DOI:10.1099/mic.0.2008/020164-0 [ Links ]
15 Griffith DE, Aksamit T, Brown-Elliott BA, Catanzaro A, Daley C, Gordin F, et al. An official ATS/IDSA statement: diagnosis, treatment, and prevention of nontuberculous mycobactecterial diseases. Am J Respir Crit Care Med. 2007;175:367-416. DOI:10.1164/RCCM.200604-571ST [ Links ]
16 Secretaria de Vigilância em Saúde. Manual Nacional de Vigilância Laboratorial da Tuberculose e outras Micobactérias. Brasília: Ministério da Saúde; 2008.
17 Kim H, Kim SH, Shim TS, Kim MN, Bai GH, Park YG, et al. PCR restriction fragment length polymorphism analysis (PRA)-algorithm targeting 644 bp Heat Shock Protein 65 (hsp65) gene for differentiation of Mycobacterium spp. J Microbiol Methods. 2005 Aug;62(2):199-209. DOI:10.1016/J.MIMET.2005.02.010 [ Links ]
18 Shin S, Kim EC, Yoon JH. Identification of nontuberculous mycobacteria by sequence analysis of the 16S ribosomal RNA, the heat-shock protein 65 and the RNA polymerase b-subunit genes. Korean J Lab Med. 2006 Jun;26(3):153-60. [ Links ]
19 Brunello F, Ligozzi M, Cristelli E, Bonora S, Tortoli E, Fontana R. Identification of 54 mycobacterial species by PCR-restriction fragment length polymorphism analysis of the hsp65 gene. J Clin Microbiol. 2001 Aug;39(8):2799-806. DOI:10.1128/JCM.39.8.2799-2806.2001 [ Links ]
20 Devallois A, Goh KS, Rastogi N. Rapid identification of mycobacteria to species level by PCR-restriction fragment length polymorphism analysis of the hsp65 gene and proposition of an algorithm to differentiate 34 mycobacterial species. J Clin Microbiol. 1997 Nov;35(11):2969-73. [ Links ]
21 Leão SC, Bernadelli A, Cataldi A, Zumarraga M, Robledo J, Realpe T, et al. Multicenter evaluation of mycobacteria identification by PCR restriction enzyme analysis in laboratories from Latin America and the Caribbean. J Microbiol Methods. 2005 May;61(2):193-9. DOI:10.1016/J.MIMET.2004.11.015 [ Links ]
22 Ritacco V, Kremer K, Van der Laan T, Pijnenburg JE, Haas PE, van Soolingen D. Use of IS901 and IS1245 in RFLP typing of Mycobacterium avium complex: relatedness among serovar reference strains, human and animal isolates. Int J Tuberc Lung Dis. 1998 Mar;2(3):242-51. [ Links ]
23 Yakrus MA, Good RC. Geographic distribution, frequency, and specimen source of Mycobacterium avium complex serotypes isolated from patients with acquired immunodeficiency syndrome. J Clin Microbiol. 1990 May;28(5):926-9.
24 Chimara E, Ferrazoli L, Ueky SY, Martins MC, Durham AM, Arbeit RD, et al. Reliable identification of mycobacterial species by PCR-restriction enzyme analysis (PRA)-hsp65 in a reference laboratory and elaboration of a sequence-based extended algorithm of PRA-hsp65 patterns. BMC Microbiology. 2008;8(48):1-12. DOI:10.1186/1471-2180-8-48 [ Links ]
25 Murcia MI, Tortoli E, Menendez MC, Palenque E, Garcia MJ. Mycobacterium colombiense sp. nov., a novel member of the Mycobacterium avium complex and description of MAC-X as a new ITS genetic variant. Int J Syst Evol Microbiol. 2006;56(9):2049-54. DOI:10.1099/ijs.0.64190-0 [ Links ]
26 Turenne CY, Wallace R Jr, Behr MA. Mycobacterium avium in the postgenomic era. Clin Microbiol Rev. 2007 Apr;20(2):205-29. DOI:10.1128/CMR.00036-06 [ Links ]
27 Frothingham R, Wilson KH. Molecular phylogeny of the Mycobacterium avium complex demonstrates clinically meaningful divisions. J Infect Dis. 1994 Feb;169(2):305-12. [ Links ]
28 Maekura R, Okuda Y, Hirotani A, Kitada S, Hiraga T, Yoshimura K, et al. Clinical and prognostic importance of serotyping Mycobacterium avium-Mycobacterium intracellulare complex isolates in human immunodeficiency virus-negative patients. J Clin Microbiol. 2005 Jul;43(7):3150-8. DOI:10.1128/JCM.43.7.3150-3158.2005 [ Links ]
 Correspondência
/ Correspondence/ Correspondencia:
Correspondência
/ Correspondence/ Correspondencia:
Ana Roberta Fusco da Costa
Instituto Evandro Chagas,
Seção de Bacteriologia e Micologia,
Laboratório de Biologia Molecular
Rodovia BR 316, km 07, s/no. Bairro: Levilândia
CEP:67030-000 Ananindeua-Pará-Brasil
E-mail:anacosta@iec.pa.gov.br/robertafusco@gmail.com
Tel./Fax: +55 (91) 3214-2116 / +55 (91) 3214-2129
Recebido em / Received / Recibido en: 31/7/2010
Aceito em / Accepted / Aceito en: 24/8/2010










 text in
text in