Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Pan-Amazônica de Saúde
Print version ISSN 2176-6215On-line version ISSN 2176-6223
Rev Pan-Amaz Saude vol.2 no.2 Ananindeua June 2011
http://dx.doi.org/10.5123/S2176-62232011000200009
COMUNICAÇÃO
Suscetibilidade de uma linhagem celular murina contínua (GRX) à infecção viral
Maria Liz GamarraI; Maria Carolina Maciel AlbuquerqueI; Anderson Junger TeodoroII; Renata Brum MartucciIII; Radovan BorojevicII; Fernando Portela CâmaraI; Maria Teresa Villela RomanosI; Norma SantosI
IDepartamento de Virologia, Instituto de Microbiologia Paulo de Góes, Universidade Federal do Rio de Janeiro, Rio de Janeiro, Rio de Janeiro, Brasil
IIDepartamento de Histologia, Instituto de Ciências Biológicas, Universidade Federal do Rio de Janeiro, Rio de Janeiro, Rio de Janeiro, Brasil
IIIDepartamento de Nutrição Aplicada, Instituto de Nutrição, Universidade Estadual do Rio de Janeiro, Rio de Janeiro, Brasil
Endereço para correspondência
Correspondence
Dirección para correspondencia
Original Title: Susceptibility of a continuous murine cell line (GRX) to viral infection. Translated by: André Monteiro Diniz
RESUMO
Este estudo avaliou a capacidade de uma linhagem celular murina (GRX) de realizar a replicação viral. Culturas de células GRX foram infectadas com diferentes vírus DNA e RNA. Foi observado que a linhagem celular GRX é suscetível à replicação dos vírus Herpes simplex tipos 1 e 2 (HSV-1 e HSV-2), Mayaro (MAY), Sindbis (SIN) e vírus da encefalite equina do oeste (WEE) e pode ser utilizada como suporte para estudos sobre replicação viral. A replicação viral induziu o efeito citopático 24 a 48 h pós-infecção. As células GRX produziram titulações de vírus infecciosos entre 102.4 TCID50 (dose infecciosa de cultura de tecido50)/25 µL e 105.4 TCID50/25 µL na primeira passagem viral. Esses resultados demonstram que as células GRX sustentam, de forma eficiente, a replicação viral e, portanto, podem ser utilizadas como uma ferramenta valiosa para estudos laboratoriais sobre virologia.
Palavras chave: Técnica de cultura de células; Células Estreladas do Fígado (GRX); Replicação Viral.
Durante as duas últimas décadas, a aplicação de metodologias moleculares e sorológicas tem tido um grande impacto na detecção de novos vírus. Ainda assim, o isolamento viral permanece sendo o método considerado padrão ouro para a identificação e caracterização de suas propriedades biológicas e bioquímicas. A cultura de células ainda é o método mais comum de propagação de vírus. Os métodos baseados em cultura celular também são utilizados na produção de vacinas e em estudos bioquímicos e biomoleculares sobre replicação viral1. Em 1985, a linhagem celular contínua GRX, representante das células estreladas hepáticas, foi estabelecida a partir de lesões fibrogranulomatosas induzidas no fígado de camundongos por meio de infecção esquistossômica2 e suas características biológicas e bioquímicas foram determinadas3,4,5. Ela se caracteriza por ser uma linhagem celular altamente prolífica que apresenta uma morfologia fibroblástica estrelada, poligonal ou alongada. Quando suas monocamadas são confluentes, as células agrupam-se em um padrão de crescimento bem delineado na forma de picos e vales2. A capacidade desta linhagem celular de servir de base para a replicação viral nunca foi demonstrada; portanto, o objetivo principal deste trabalho foi demonstrar a utilidade da linhagem celular GRX na propagação de certos vírus.
As células GRX foram obtidas do Banco de Células do Estado do Rio de Janeiro, Brasil. As linhagens celulares do rim do macaco verde africano (Vero), do rim do macaco rhesus (MA-104) e do epitélio humano (HEp-2) foram obtidas de soluções stock utilizadas para isolamento viral de rotina no Departamento de Virologia do Instituto de Microbiologia da Universidade Federal do Rio de Janeiro, Brasil. Todas as linhagens celulares foram cultivadas em meio mínimo essencial (MEM) complementado com 5% (células de HEp-2) ou 10% (células Vero, MA-104 e GRX) de soro fetal bovino (SFB) inativado pelo calor, 2 mM de L-glutamina, 50 µg/mL de gentamicina, 2,5 µg/mL de fungizona, bicarbonato de sódio a 0,25% e 10 mM de HEPES. As culturas foram incubadas a uma temperatura de 37o C com CO2 a 5%.
As cepas virais utilizadas nesta pesquisa foram obtidas do acervo de referência do Departamento de Virologia do Instituto de Microbiologia da Universidade Federal do Rio de Janeiro. Os vírus da encefalite equina do oeste (WEE), Herpes simplex tipos 1 (HSV-1) e 2 (HSV-2), Sindbis (SIN) e Mayaro (MAY) foram mantidos em células Vero; os sorotipos 2, 19, 40 e 41 do Adenovirus (AdV) foram mantidos em células HEp-2; e a cepa SA-11 de Rotavirus (RV) foi mantida em células MA-104.
Para a propagação dos vírus, as monocamadas celulares foram preparadas em placas de 48 poços; cada suspensão viral foi inoculada em triplicata. As placas inoculadas foram incubadas por 1 h a uma temperatura de 37o C. Posteriormente, os inóculos foram substituídos por MEM sem SFB (meio de manutenção). Para os rotavírus, foi adicionada ao inoculo tripsina a uma concentração final de 10 µg/mL, seguida de incubação a 37o C por 30 min. O inóculo foi então adicionado à monocamada com meio de manutenção a uma concentração final de tripsina de 5 µg/mL. Todas as culturas de células infectadas foram incubadas a 37o C com CO2 a 5%. As células foram monitoradas diariamente em microscópio ótico invertido visando observar o desenvolvimento do efeito citopático (CPE). Quando aproximadamente 75% das células apresentaram CPE, ou após sete dias de incubação sem que se observasse o CPE, as culturas infectadas foram submetidas a três ciclos de congelamento-descongelamento. Foram então coletadas as misturas de lisados celulares e sobrenadantes e armazenadas a -70o C para análises futuras. Cada cepa viral foi submetida a três passagens seriadas em células GRX. As misturas de sobrenadante e lisado celular (suspensão viral) de cada uma das três passagens foram submetidas à titulação pelo método da diluição limite (endpoint dilution method) para quantificar a infectividade viral. Diluições logarítmicas de 25 µL da suspensão viral foram inoculadas em monocamadas confluentes em placas de 96 poços (seis poços/diluição). As titulações foram calculadas de acordo com o método de Reed e Muench e expressas em log10 TCID50 (dose infecciosa de cultura celular50)/25 µL6.
Em placas de 24 poços, as células GRX, a uma densidade de 4,2 x 105 células/mL e células Vero a uma densidade de 1,2 x 106 células/mL foram semeadas em MEM complementado com SFB a 10% e infectadas com 100 µL de suspensão viral (MAY, SIN ou WEE) a uma multiplicidade de infecção de 1,0, 0,5 e 0,1. As culturas de células infectadas foram incubadas a 37o C em ambiente com CO2 a 5% por 48 h. Após esse período, a suspensão viral foi coletada e titulada como descrito acima. Todos os experimentos foram realizados em triplicata.
A análise estatística foi realizada utilizando-se a análise de variância (ANOVA) para determinar a diferença menos significante em p = 0.05. Essa análise foi efetuada com o programa Minitab® for Windows, versão 14.0 (Minitab Inc., State College, PA, EUA). O termo "significância" (estatisticamente significante) no texto equivale a p≤0.05.
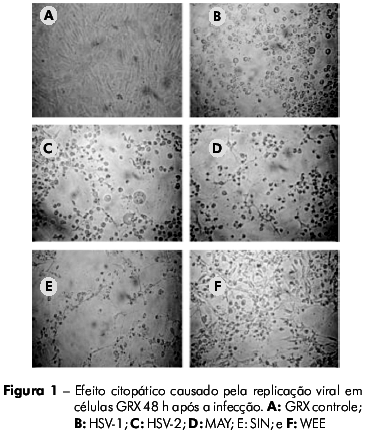
As células GRX foram suscetíveis à infecção por HSV-1, HSV-2, SIN e WEE, produzindo CPE visível em 48 h. O vírus MAY induziu o CPE em 24 h. Em contraste, as células GRX não auxiliaram replicação viral perceptível quando inoculadas com AdV ou RV (Tabela 1). O CPE observado nos vírus MAY, SIN e WEE em células GRX foi caracterizado por picnose celular, seguido de fragmentação citoplasmática e desprendimento da monocamada, semelhante ao CPE observado em células Vero. Já o CPE observado para os vírus HSV-1 e HSV-2 foi caracterizado por arredondamento e refringencia das células (Figuras 1 e 2).

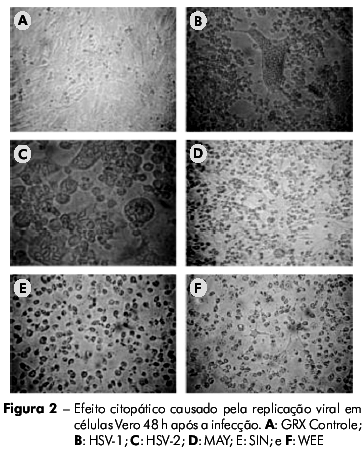
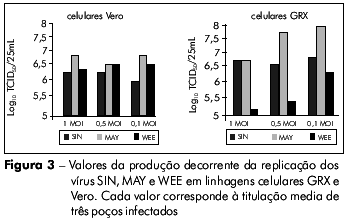
Para demonstrar a eficiência da replicação viral, foi medida a produção viral em células GRX comparada à de células Vero após a inoculação de vírus com diferentes multiplicidades de infecção. Não foi observada qualquer diferença estatisticamente significativa entre as duas linhagens celulares, o que indicou que a replicação viral ocorre de forma eficiente nos dois sistemas (Figura 3). No caso das células Vero, não houve nenhuma diferença significativa quando os vírus foram inoculados com diferentes multiplicidades de infecção. Já para as células GRX, apesar de não haver diferença significativa na produção viral para o os vírus SIN e MAY com diferentes multiplicidades de infecção, houve uma variância na replicação dos vírus MAY quando foram inoculados com baixa multiplicidade de infecção. Inversamente, houve uma diferença estatisticamente relevante (p = 0.016) na produção viral de WEE quando diferentes multiplicidades de infecção foram utilizadas (Figura 3).
Apesar da utilização de várias linhagens celulares para o isolamento viral, ainda há um número significativo de vírus que não podem ser propagados em laboratório. Portanto, o desenvolvimento de uma nova linhagem celular suscetível à replicação viral cria novas alternativas para o cultivo desses vírus. A busca por novas alternativas tem se intensificado principalmente por causa da emergência de novos vírus, como o metapneumovírus humano7 e os novos coronavírus humanos (SARS-CoV, HCoV-NL63, HCoV-HKU1)8,9,10, ou por causa da necessidade do isolamento viral para estudos moleculares e bioquímicos, como no caso do norovírus11, vírus das hepatites B12 e C13. Consequentemente, há o registro da publicação de pesquisas avaliando a utilidade de várias linhagens celulares para o isolamento viral14,15,16.
A linhagem GRX foi descrita pela primeira vez em 19852; no entanto, a utilidade dessas células para a replicação viral ainda não foi determinada. Neste estudo, foi avaliada a capacidade desta linhagem celular de servir de base à propagação de diversos vírus, bem como a sua utilidade como uma ferramenta em laboratórios para estudos sobre virologia. Geralmente, a determinação da suscetibilidade de uma linhagem celular para a replicação viral pode ser realizada por meio da observação do CPE característico, seguido de titulação viral, ou por meio de metodologias alternativas, como o teste de hemaglutinação, por imunofluorescência ou a detecção do genoma viral por métodos moleculares14,15,16. Neste estudo, foram utilizadas observações do CPE e titulação viral como os parâmetros para a demonstração da suscetibilidade celular. Todos os vírus neste estudo produziram CPE visível em outras linhagens celulares; por isso, seria vantajoso estabelecer uma nova linhagem celular para aqueles vírus que demandassem metodologias alternativas para demonstrar a propagação viral em culturas.
Os alfavírus WEE, MAY e SIN infectam uma grande variedade de hospedeiros na natureza, replicando-se em mamíferos, pássaros, artrópodes e anfíbios; esses vírus podem se propagar in vitro em várias linhagens celulares17,18,19. A inoculação de células GRX com esses vírus produziu CPE em 24 a 48 h. Altas titulações virais foram obtidas a partir da primeira passagem. Elas aumentaram progressivamente com as passagens consecutivas, demonstrando a produção de partículas virais infecciosas. As células GRX forneceram um substrato para a propagação viral tão eficiente quando as células Vero, que são regularmente utilizadas para a propagação de alfavírus.
Diferentes linhagens celulares podem ser utilizadas para o isolamento de HSVs20. Os vírus HSV-1 e HSV-2 foram propagados com sucesso em células GRX 48 h após a inoculação, o que demonstra que, apesar das titulações virais obtidas terem sido mais baixas do que aquelas das células Vero, as células GRX podem ser utilizadas de forma eficiente como sistema de cultura para esses vírus.
Curiosamente, neste estudo todos os vírus que se propagaram nas células GRX são envelopados (Alfavírus), contrariamente aos não envelopados, como os adenovírus e os rotavírus. Uma possível explicação para este fenômeno pode ser a especificidade do receptor viral. Outra explicação pode ser a presença de enzimas celulares necessárias para a replicação viral. Há a necessidade de novos estudos para elucidar esta questão.
Os resultados demonstraram que a linhagem celular GRX exibiu uma alta suscetibilidade a diferentes vírus, produzindo altas titulações. O CPE induzido por vírus foi observado no período entre 24 e 48 h, dependendo do vírus utilizado. A produção viral demonstrou que as titulações aumentaram progressivamente de acordo com as passagens, o que indicou que as células GRX podem sustentar, de forma eficiente, a replicação viral. A linhagem celular GRX mostrou-se altamente suscetível aos vírus HSV-1, HSV-2, MAY, SIN e WEE e pode ser utilizada como uma ferramenta para o isolamento viral e estudos bioquímicos.
Apesar de haver diversas linhagens celulares amplamente utilizadas no campo da virologia para propagar vírus, o estabelecimento de novos sistemas celulares sempre abre novas possibilidades de análises mais profundas da biologia e bioquímica das infecções virais. A linhagem celular GRX foi estabelecida no início da década de 1980 e suas propriedades biológicas e bioquímicas já foram determinadas. No entanto, sua capacidade de dar suporte á replicação viral ainda não foi demonstrada claramente. O principal objetivo deste trabalho foi demonstrar a utilidade desta linhagem celular na propagação viral. Para efeitos de comparação, inicialmente foram analisados vírus que já tenham um sistema celular eficiente. Após o estabelecimento da suscetibilidade da linhagem celular GRX a certos vírus, pode-se continuar a estudar sua capacidade de dar suporte ao crescimento de vírus que ainda não tenham sido propagados in vitro, como os da hepatite C e os bocavírus humanos.
Em conclusão, (i) foi possível alcançar o principal objetivo deste estudo, qual seja o de demonstrar a suscetibilidade da linhagem celular GRX à infecção viral; (ii) foi demonstrado que esta linhagem celular é uma ferramenta virológica valiosa; e (iii) os resultados aqui apresentados abrem a possibilidade de utilização da linhagem celular GRX para a avaliação de vírus não propagados anteriormente in vitro por estes pesquisadores ou por quaisquer outros laboratórios interessados nesses estudos.
AGRADECIMENTOS
Os autores gostariam de agradecer à Sra. Soluza dos Santos Gonçalves pelo apoio técnico.
APOIO FINANCEIRO
Esta pesquisa contou com o apoio financeiro do Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq), da Coordenação de Aperfeiçoamento de Pessoal de Nível Superior (CAPES), e da Fundação Carlos Chagas de Amparo à Pesquisa do Estado do Rio de Janeiro (FAPERJ), Brasil.
REFERÊNCIAS
1 Olivo PD. Transgenic cell lines for detection of animal viruses. Clin Microbiol Rev. 1996 Jul;9(3):321-34. [Link]
2 Borojevic R, Monteiro ANA, Vinhas SA, Domont GB, Mourão PAS, Emonard H, et al. Establishment of a continuous cell line from fibrotic schistosomal granulomas in mice livers. In Vitro Cell Dev Biol. 1985 Jul;21(7):382-90. [Link]
3 Fortuna VA, Trugo LC, Borojevic R. Acyl-CoA: retinol acyltransferase (ARAT) and lecithin: reti no l acyltransferase (LRAT) activation during the lipocyte phenotype induction in hepatic stellate cells. J Nutr Biochem. 2001 Nov;12(11):610-21. Doi:10.1016/S0955-2863(01)00179-6 [Link]
4 Guma FCR, Mello TG, Mermelstein CS, Fortuna V, Wofchuk ST, Gottfried C, et al. Intermediate filaments modulation in an in vitro model of the hepatic stellate cell activation or conversion into the lipocyte phenotype. Biochem Cell Biol. 2001;79(4):409-17. [Link]
5 Martucci RB, Ziulkoski AL, Fortuna VA, Guaragna RM, Guma FCR, Trugo LC, et al. β-carotene storage, convertion to retinoic acid, and induction of the lipocyte in hepatic stellate cells. J Cell Biochem. 2004 May;92(2):414-23. DOI:10.1002/jcb.20073 [Link]
6 Reed LJ, Muench H. A simple method of estimating fifty per cent endpoints. Am J Hyg. 1938 May;27(3):493-7.
7 Van den Hoogen BG, Jong JC, Groen J, Huiken T, Groot R, Fouchier RAM, et al. A newly discovered human pneumovirus isolated from young children with respiratory tract disease. Nat Med. 2001 Jun;7(6):719-24. Doi:10.1038/89098 [Link]
8 Ksiazek T, Erdman D, Goldsmith CS, Zaki SR, Peret T, Emery S, et al. A novel coronavirus associated with severe acute respiratory syndrome. N Engl J Med. 2003 May;348(20):1953-66. [Link]
9 Van der Hoek L, Pyrc K, Jebbink MF, Vermeulen-Oost W, Berkhout RJM, Wolthers KC, et al. Identification of a new coronavirus. Nat Med. 2004 Apr;10(4):368-73. [Link]
10 Woo PCY, Lau SKP, Chu CM, Chan KH, Tsoi HW, Huang Y, et al. Characterization and complete genome sequence of a novel coronavirus, coronavirus HKU1, from patients with pneumonia. J Virol. 2005 Jan;79(2):884-95. Doi:10.1128/JVI.79.2.884-895.2005 [Link]
11 Duizer E, Schwab KJ, Neil FH, Atmar RL, Koopmans MP, Estes MK. Laboratory effort to cultivate norovirus. J Gen Virol. 2004 Jan;85(Pt 1):79-87. [Link]
12 Guha C, Mohan S, Roy-Chowdhury N, Roy-Chowdhury J. Cell culture and animal models of viral hepatitis. Part I: hepatitis B. Lab Anim (NY). 2004 Jul-Aug;33(7):37-46. [Link]
13 Durantel D, Zoulim F. Going towards more relevant cell culture models to study the in vitro replication of serum-derived hepatitis C virus and virus/host cell interactions? J Hepatol. 2007 Jan;46(1):1-5.
14 El-Awady MK, Tabll AA, El-Abd YS, Bahgat MM, Shoeb HA, Youssef SS, et al. HepG2 cells support viral replication and gene expression of hepatitis C virus genotype 4 in vitro. World J Gastroenterol. 2006 Aug;12(30):4836-42. [Link]
15 Ingram RE, Fenwick F, McGuckin R, Tefari A, Taylor C, Toms GL. Detection of human metapneumovirus in respiratory secretions by reverse-transcriptase polymerase chain reaction, indirect immunofluorescence, and virus isolation in human bronchial epithelial cells. J Med Virol. 2006 Sep;78(9):1223-31. DOI:10.1002/jmv.20685 [Link]
16 Qin QW, Wu TH, Jia TL, Hegde A, Zhang RQ. Development and characterization of a new tropical marine fish cell line from grouper, Epinephelus coioides susceptible to iridovirus and nodavirus. J Virol Meth. 2006 Jan;131(1):58-64. Doi:10.1016/J.Jviromet.2005.07.009 [Link]
17 Bello FJ, Rodríguez JA, Escovar J, Olano VA, Morales A, González M, et al. A new continuous cell line from the mosquito Psorophora confinnis (Diptera: Culicidae) and its susceptibility to infections with some arboviruses. Mem Inst Oswaldo Cruz. 2001 Aug;96(6):865-73. http://dx.doi.org/10.1590/S0074-02762001000600022 [Link]
18 Ferreira DF, Santo MPE, Rebello MA, Rebello MCS. Weak bases affect late stages of Mayaro virus replication cycle in vertebrate cell. J Med Microbiol. 2000 Apr;49(4):313-8. [Link]
19 Weingartl HM, Sabara M, Pasick J, Van Moorlehem E, Babiuk L. Continuous porcine cell line developed from alveolar macrophages: partial characterization and virus susceptibility. J Virol Meth. 2002 Jul;104(2):203-16. Doi:10.1016/S0166-0934(02)00085-X [Link]
20 Ustaçelebi S. Diagnosis of herpes simplex virus infections. J Clin Virol. 2001 Jun;21(3):255-9.
 Correspondence / Correspondência / Correspondencia:
Correspondence / Correspondência / Correspondencia:
Norma Santos
Departamento de Virologia,
Instituto de Microbiologia Paulo de Góes
Universidade Federal do Rio de Janeiro
Cidade Universitária, CCS - Bl. I, Ilha do Fundão
CEP: 21.941-590
Rio de Janeiro-Rio de Janeiro-Brasil
Tel: + 55 (21) 2560-8344 Fax + 55 (21) 2560-88028
E-mail:nsantos@micro.ufrj.br
Received / Recebido em / Recibido en: 3/5/2011
Accepted / Aceito em / Aceito en: 16/9/2011










 text in
text in