Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista Pan-Amazônica de Saúde
versão impressa ISSN 2176-6215versão On-line ISSN 2176-6223
Rev Pan-Amaz Saude v.2 n.2 Ananindeua jun. 2011
http://dx.doi.org/10.5123/S2176-62232011000200009
COMUNICACIÓN
Susceptibilidad de un linaje celular murino continuo (GRX) a la infección viral
Maria Liz GamarraI; Maria Carolina Maciel AlbuquerqueI; Anderson Junger TeodoroII; Renata Brum MartucciIII; Radovan BorojevicII; Fernando Portela CâmaraI; Maria Teresa Villela RomanosI; Norma SantosI
IDepartamento de Virologia, Instituto de Microbiologia Paulo de Góes, Universidade Federal do Rio de Janeiro, Rio de Janeiro, Rio de Janeiro, Brasil
IIDepartamento de Histologia, Instituto de Ciências Biológicas, Universidade Federal do Rio de Janeiro, Rio de Janeiro, Rio de Janeiro, Brasil
IIIDepartamento de Nutrição Aplicada, Instituto de Nutrição, Universidade Estadual do Rio de Janeiro, Rio de Janeiro, Brasil
Endereço para correspondência
Correspondence
Dirección para correspondencia
Título original: Susceptibility of a continuous murine cell line (GRX) to viral infection. Traducido por: Lota Moncada
RESUMEN
Este estudio evaluó la capacidad de un linaje celular murino (GRX) de realizar la replicación viral. Culturas de células GRX fueron infectadas con diferentes virus ADN y ARN. Se observó que el linaje celular GRX es susceptible a la replicación de los virus Herpes simplex tipos 1 y 2 (HSV-1 y HSV-2), Mayaro (MAY), Sindbis (SIN) y al virus de la encefalitis equina del oeste (WEE) y puede utilizarse como soporte para estudios sobre replicación viral. La replicación viral indujo el efecto citopático 24 a 48 h pos-infección. Las células GRX produjeron titulaciones de virus infecciosos entre 102.4 TCID50 (dosis infecciosa de cultura de te¡¡do50)/25 µL y 105.4 TCID50/25 µL en el primer pasaje viral. Estos resultados demuestran que las células GRX sostienen, de forma eficiente, la replicación viral y, por lo tanto, pueden ser utilizadas como una valiosa herramienta para estudios de laboratorio sobre virología.
Palabras clave: Técnicas de Cultivo de Célula; Células Estrelladas Hepáticas (GRX); Replicación Viral.
Durante las dos últimas décadas, la aplicación de metodologías moleculares y serológicas han tenido un gran impacto en la detección de nuevos virus. Aún así, el asilado viral permanece siendo el método considerado estándar oro para la identificación y caracterización de sus propiedades biológicas y bioquímicas. El cultivo de células todavía es el método más común de propagación de virus. Los métodos basados en cultivo celular también se utilizan en la producción de vacunas y en estudios bioquímicos y biomoleculares sobre la replicación viral1. En 1985, el linaje celular continuo GRX, representante de las células estrelladas hepáticas, fue establecido a partir de lesiones fibrogranulomatosas inducidas en el hígado de ratones por medio de infección esquistosómica2 y sus características biológicas y bioquímicas fueron determinadas3,4,5. Se caracteriza por ser un linaje celular altamente prolífico que presenta una morfología fibroblástica estrellada, poligonal o alargada. Cuando sus monocapas son confluentes, las células se agrupan en un estándar de crecimiento bien delineado en forma de picos y valles2. La capacidad de este linaje celular de servir de base a la replicación viral nunca fue demostrada; por lo tanto, el objetivo principal de este trabajo fue el de demostrar la utilidad del linaje celular GRX en la propagación de ciertos virus.
Las células GRX se obtuvieron en el Banco de Células del Estado de Rio de Janeiro, Brasil. Los linajes celulares del riñón del mono verde africano (Vero), del riñón del mono rhesus (MA-104) y del epitelio humano (HEp-2) fueron obtenidos de soluciones stock utilizadas para aislado viral de rutina en el Departamento de Virología del Instituto de Microbiología de la Universidad Federal de Rio de Janeiro, Brasil. Todas los linajes celulares fueron cultivados en medio mínimo esencial (MEM) complementado con 5% (células de HEp-2) o 10% (células Vero, MA-104 y GRX) de suero fetal bovino (SFB) inactivado por calor, 2 mM de L-glutamina, 50 µg/mL de gentamicina, 2,5 µg/mL de fungizona, bicarbonato de sodio a 0,25% y 10 mM de HEPES. Los cultivos fueron incubados a una temperatura de 37o C con CO2 a 5%.
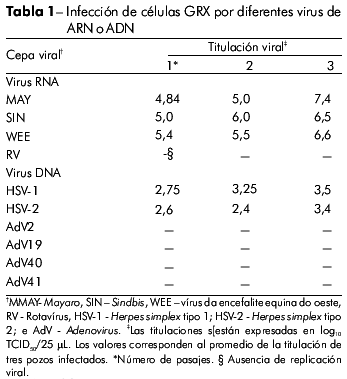
Las cepas virales utilizadas en esta investigación se obtuvieron del acervo de referencia del Departamento de Virología del Instituto de Microbiología de la Universidad Federal de Rio de Janeiro. Los virus de encefalitis equina del oeste (WEE), Herpes simplex tipos 1 (HSV-1) y 2 (HSV-2), Sindbis (SIN) y Mayaro (MAY) fueron mantenidos en células Vero; los serotipos 2, 19, 40 y 41 del Adenovirus (AdV) fueron mantenidos en células HEp-2; y la cepa SA-11 de Rotavirus (RV) se mantuvo en células MA-104.
Para la propagación de los virus, las monocapas celulares fueron preparadas en placas de 48 pozos; cada suspensión viral fue inoculada por triplicado. Las placas inoculadas fueron incubadas por 1h a una temperatura de 37o C. Posteriormente, los inóculos fueron sustituidos por MEM sin SFB (medio de manutención). Para los rotavirus, se adicionó el inóculo tripsina en una concentración final de 10 µg/mL, seguida de incubado a 37o C por 30 min. El inoculo fue entones agregado a la monocapa con medio de manutención a una concentración final de tripsina de 5 µg/mL. Todos los cultivos de células infectadas fueron incubados a 37o C con CO2 a 5%. Las células fueron monitoreadas diariamente en microscopio óptico invertido para observar el efecto citopático (CPE). Cuando aproximadamente 75% de las células presentaron CPE, o después de siete días de incubado sin que se observase el CPE, los cultivos infectados fueron sometidos a tres ciclos de congelado-descongelado. Fueron entonces recolectadas las mezclas de lisados celulares y sobrenadantes y almacenadas a -70° C para análisis futuros. Cada cepa viral fue sometida a tres pasajes seriados en células GRX. Las mezclas de sobrenadante y lisado celular (suspensión viral) de cada uno de los tres pasajes fueron sometidas a titulación por el método de dilución límite (endpoint dilution method) para cuantificar la infectividad viral. Diluciones logarítmicas de 25 µL de la suspensión viral fueron inoculadas en monocapas confluentes en placas de 96 pozos (seis pozos/dilución). Las titulaciones se calcularon de acuerdo al método de Reed y Muench y fueron expresadas en log10 TCID50 (dosis infecciosa de cultivo celular50)/25 µL6.
En placas de 24 pozos, las células GRX, a una densidad de 4,2 x 105 células/mL y células Vero a una densidad de 1,2 x 106 células/mL fueron sembradas en MEM complementado con SFB a 10% e infectadas con 100 µL de suspensión viral (MAY, SIN o WEE) a una multiplicidad de infección de 1,0, 0,5 y 0,1. Los cultivos de células infectadas fueron incubados a 37o C en ambiente con CO2 a 5% por 48 h. Luego de ese período, la suspensión viral fue colectada y titulada como antes descrito. Todos los experimentos se realizaron en triplicado.
El análisis estadístico se realizó utilizándose el análisis de varianza (ANOVA) para determinar la diferencia menos significante en p = 0.05. Ese análisis se efectuó con el programa Minitab® for Windows, versión 14.0 (Minitab Inc., State College, PA, EUA). El término "significante" (estadísticamente significante) en el texto equivale a p < 0.05.
Las células GRX fueron susceptibles a la infección por HSV-1, HSV-2, SIN y WEE, produciendo CPE visible en 48 h. El virus MAY indujo el CPE en 24 h. En contraste, las células GRX no auxiliaron replicación viral perceptible cuando inoculadas con AdV o RV (Tabla 1). El CPE observado en los virus MAY, SIN y WEE en células GRX se caracterizó por picnosis celular, seguida de fragmentación citoplasmática y desprendimiento de la monocapa, semejante al CPE observado en células Vero. El CPE observado para los vírus HSV-1 y HSV-2 fue caracterizado por redondeo y refringencia de las células (Figuras 1 y 2).
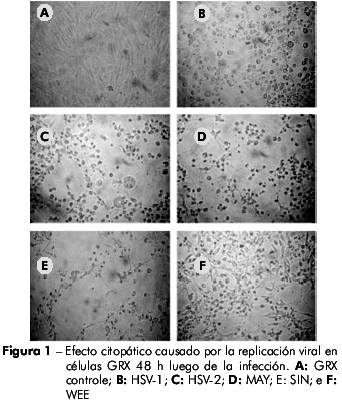
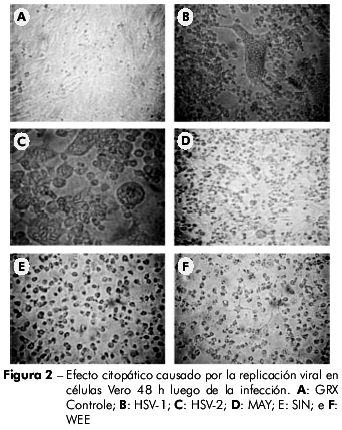
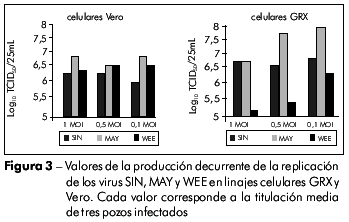
Para demostrar la eficiencia de la replicación viral, fue medida la producción viral en células GRX comparada a la de células Vero luego de la inoculación de virus con diferentes multiplicidades de infección. No se observó ninguna diferencia estadísticamente significativa entre los dos linajes celulares, lo que indicó que la replicación viral ocurre de forma eficiente en los dos sistemas (Figura 3). En el caso de las células Vero, no hubo diferencia significativa cuando los virus fueron inoculados con diferentes multiplicidades de infección. Ya para la células GRX, a pesar de no haber diferencia significativa en la producción viral para los virus SIN y MAY con diferentes multiplicidades de infección, hubo una varianza en la replicación de los virus MAY cuando fueron inoculados con baja multiplicidad de infección. Inversamente, hubo una diferencia estadísticamente relevante (p=0.016) en la producción viral de WEE cuando se utilizaron diferentes multiplicidades de infección (Figura 3).
A pesar de la utilización de varios linajes celulares para el aislado viral, todavía hay un número significativo de virus que no pueden ser propagados en laboratorio. Por lo tanto, el desarrollo de un nuevo linaje celular susceptible a la replicación viral crea nuevas alternativas para el cultivo de esos virus. La búsqueda de nuevas alternativas se ha intensificado principalmente por causa de la emergencia de nuevos virus, como el metapneumovirus humano7 y los nuevos coronavirus humanos (SARS-CoV, HCoV-NL63, HCoV-HKU1)8,9,10, o por causa de la necesidad del aislado viral para estudios moleculares y bioquímicos, como en el caso del norovirus11, virus de las hepatitis B12 y C13. Consecuentemente, existe registro de la publicación de investigaciones evaluando la utilidad de varios linajes celulares para el asilamiento viral14,15,16.
El linaje GRX fue descrito por primera vez en 19852; sin embargo, la utilidad de esas células para la replicación viral aún no ha sido determinada. En este estudio fue evaluada la capacidad de este linaje celular de servir de base a la propagación de diversos virus, bien como su utilidad como una herramienta en laboratorios para estudios sobre virología. Generalmente, para determinar la susceptibilidad de un linaje celular para la replicación viral se realiza por medio de la observación del CPE característico, seguido de titulación viral, o por medio de metodologías alternativas, como la prueba de hemoaglutinación, por inmunofluorescencia o la detección del genoma viral por métodos moleculares14,15,16. En este estudio, se utilizaron observaciones del CPE y titulación viral como los parámetros para la demostración de la susceptibilidad celular. Todos los virus en este estudio, produjeron CPE visible en otros linajes celulares; por eso, sería interesante establecer un nuevo linaje celular para los virus que demandaran metodologías alternativas para demostrar la propagación viral en cultivos.
Los alfavirus WEE, MAY y SIN infectan a una gran variedad de huéspedes en la naturaleza, replicándose en mamíferos, pájaros, artrópodos y anfibios; esos virus pueden propagarse in vitro en varios linajes celulares17,18,19. La inoculación de células GRX con esos virus produjo CPE en 24 a 48 h. Se obtuvieron altas titulaciones virales a partir del primer pasaje, las que aumentaron progresivamente con los pasajes consecutivos, demostrando la producción de partículas virales infecciosas. Las células GRX suministraron un substrato para la propagación viral tan eficiente como las células Vero, que son regularmente utilizadas para la propagación de alfavirus.
Pueden utilizarse diferentes linajes celulares para el aislado de HSVs20. Los virus HSV-1 y HSV-2 fueron propagados con éxito en células GRX 48 h después de la inoculación, lo que demuestra que, a pesar de que las titulaciones virales obtenidas fueron más bajas que las de las células Vero, las células GRX pueden utilizarse de forma eficiente como sistema de cultivo para esos virus.
Curiosamente, en este estudio todos los virus que se propagaron en las células GRX son encapsulados (Alfavirus), contrariamente a los no encapsulados, como los adenovirus y los rotavirus. Una posible explicación para este fenómeno puede ser la especificidad del receptor viral. Otra explicación puede ser la presencia de enzimas celulares necesarias para la replicación viral. Hay necesidad de nuevos estudios para elucidar este tema.
Los resultados demostraron que el linaje celular GRX exhibió alta susceptibilidad a diferentes virus, produciendo también altas titulaciones. El CPE inducido por virus se observó en el período entre 24 y 48 h, dependiendo del virus utilizado. La producción viral demostró que las titulaciones aumentaron progresivamente de acuerdo a los pasajes, lo que indicó que las células GRX pueden sustentar, de forma eficiente, la replicación viral. El linaje celular GRX se mostró grandemente susceptible a lo virus HSV-1, HSV-2, MAY, SIN y WEE y puede ser utilizado como una herramienta para el aislado viral y estudios bioquímicos.
A pesar de que hay diversos linajes celulares ampliamente utilizados en el campo de la virología para propagar virus, establecer nuevos sistemas celulares siempre abre nuevas posibilidades de análisis más profundas de la biología y la bioquímica de las infecciones virales. El linaje celular GRX fue establecido al inicio de la década de 1980 y sus propiedades biológicas y bioquímicas ya fueron determinadas. No obstante, su capacidad de dar soporte a la replicación viral aún no ha sido demostrada claramente. El principal objetivo de este trabajo fue de demostrar la utilidad de este linaje celular en la propagación viral. Para efectos de comparación, inicialmente se analizaron virus que ya tenían un sistema celular eficiente. Luego de establecer la susceptibilidad del linaje celular GRX a ciertos virus, se puede continuar a estudiar su capacidad de dar soporte al crecimiento de virus que todavía no hayan sido propagados in vitro, como los de la hepatitis C y los bocavirus humanos.
En conclusión, (i) fue posible alcanzarse el principal objetivo de este estudio, el de demostrar la susceptibilidad del linaje celular GRX a la infección viral; (ii) se demostró que este linaje celular es una herramienta virológica valiosa; y (iii) los resultados aquí presentados abren la posibilidad de utilizar el linaje celular GRX para la evaluación de virus no propagados anteriormente in vitro por estos investigadores o por cualesquier otros laboratorios interesados en estos estudios.
AGRADECIMIENTOS
Los autores desean agradecer a la Sra. Soluza dos Santos Goncalves por el apoyo técnico.
APOYO FINANCIERO
Esta investigación contó con el apoyo financiero del Consejo Nacional de Desarrollo Científico y Tecnológico (CNPq), de la Coordinación de Perfeccionamiento de Personal de Nivel Superior (CAPES), y de la Fundación Carlos Chagas de Amparo a la Investigación del Estado de Rio de Janeiro (FAPERJ), Brasil.
REFERENCIAS
1 Olivo PD. Transgenic cell lines for detection of animal viruses. Clin Microbiol Rev. 1996 Jul;9(3):321-34. [Link]
2 Borojevic R, Monteiro ANA, Vinhas SA, Domont GB, Mourão PAS, Emonard H, et al. Establishment of a continuous cell line from fibrotic schistosomal granulomas in mice livers. In Vitro Cell Dev Biol. 1985 Jul;21(7):382-90. [Link]
3 Fortuna VA, Trugo LC, Borojevic R. Acyl-CoA: retinol acyltransferase (ARAT) and lecithin: reti no l acyltransferase (LRAT) activation during the lipocyte phenotype induction in hepatic stellate cells. J Nutr Biochem. 2001 Nov;12(11):610-21. Doi:10.1016/S0955-2863(01)00179-6 [Link]
4 Guma FCR, Mello TG, Mermelstein CS, Fortuna V, Wofchuk ST, Gottfried C, et al. Intermediate filaments modulation in an in vitro model of the hepatic stellate cell activation or conversion into the lipocyte phenotype. Biochem Cell Biol. 2001;79(4):409-17. [Link]
5 Martucci RB, Ziulkoski AL, Fortuna VA, Guaragna RM, Guma FCR, Trugo LC, et al. β-carotene storage, convertion to retinoic acid, and induction of the lipocyte in hepatic stellate cells. J Cell Biochem. 2004 May;92(2):414-23. DOI:10.1002/jcb.20073 [Link]
6 Reed LJ, Muench H. A simple method of estimating fifty per cent endpoints. Am J Hyg. 1938 May;27(3):493-7.
7 Van den Hoogen BG, Jong JC, Groen J, Huiken T, Groot R, Fouchier RAM, et al. A newly discovered human pneumovirus isolated from young children with respiratory tract disease. Nat Med. 2001 Jun;7(6):719-24. Doi:10.1038/89098 [Link]
8 Ksiazek T, Erdman D, Goldsmith CS, Zaki SR, Peret T, Emery S, et al. A novel coronavirus associated with severe acute respiratory syndrome. N Engl J Med. 2003 May;348(20):1953-66. [Link]
9 Van der Hoek L, Pyrc K, Jebbink MF, Vermeulen-Oost W, Berkhout RJM, Wolthers KC, et al. Identification of a new coronavirus. Nat Med. 2004 Apr;10(4):368-73. [Link]
10 Woo PCY, Lau SKP, Chu CM, Chan KH, Tsoi HW, Huang Y, et al. Characterization and complete genome sequence of a novel coronavirus, coronavirus HKU1, from patients with pneumonia. J Virol. 2005 Jan;79(2):884-95. Doi:10.1128/JVI.79.2.884-895.2005 [Link]
11 Duizer E, Schwab KJ, Neil FH, Atmar RL, Koopmans MP, Estes MK. Laboratory effort to cultivate norovirus. J Gen Virol. 2004 Jan;85(Pt 1):79-87. [Link]
12 Guha C, Mohan S, Roy-Chowdhury N, Roy-Chowdhury J. Cell culture and animal models of viral hepatitis. Part I: hepatitis B. Lab Anim (NY). 2004 Jul-Aug;33(7):37-46. [Link]
13 Durantel D, Zoulim F. Going towards more relevant cell culture models to study the in vitro replication of serum-derived hepatitis C virus and virus/host cell interactions? J Hepatol. 2007 Jan;46(1):1-5.
14 El-Awady MK, Tabll AA, El-Abd YS, Bahgat MM, Shoeb HA, Youssef SS, et al. HepG2 cells support viral replication and gene expression of hepatitis C virus genotype 4 in vitro. World J Gastroenterol. 2006 Aug;12(30):4836-42. [Link]
15 Ingram RE, Fenwick F, McGuckin R, Tefari A, Taylor C, Toms GL. Detection of human metapneumovirus in respiratory secretions by reverse-transcriptase polymerase chain reaction, indirect immunofluorescence, and virus isolation in human bronchial epithelial cells. J Med Virol. 2006 Sep;78(9):1223-31. DOI:10.1002/jmv.20685 [Link]
16 Qin QW, Wu TH, Jia TL, Hegde A, Zhang RQ. Development and characterization of a new tropical marine fish cell line from grouper, Epinephelus coioides susceptible to iridovirus and nodavirus. J Virol Meth. 2006 Jan;131(1):58-64. Doi:10.1016/J.Jviromet.2005.07.009 [Link]
17 Bello FJ, Rodríguez JA, Escovar J, Olano VA, Morales A, González M, et al. A new continuous cell line from the mosquito Psorophora confinnis (Diptera: Culicidae) and its susceptibility to infections with some arboviruses. Mem Inst Oswaldo Cruz. 2001 Aug;96(6):865-73. http://dx.doi.org/10.1590/S0074-02762001000600022 [Link]
18 Ferreira DF, Santo MPE, Rebello MA, Rebello MCS. Weak bases affect late stages of Mayaro virus replication cycle in vertebrate cell. J Med Microbiol. 2000 Apr;49(4):313-8. [Link]
19 Weingartl HM, Sabara M, Pasick J, Van Moorlehem E, Babiuk L. Continuous porcine cell line developed from alveolar macrophages: partial characterization and virus susceptibility. J Virol Meth. 2002 Jul;104(2):203-16. Doi:10.1016/S0166-0934(02)00085-X [Link]
20 Ustaçelebi S. Diagnosis of herpes simplex virus infections. J Clin Virol. 2001 Jun;21(3):255-9.
 Correspondence / Correspondência / Correspondencia:
Correspondence / Correspondência / Correspondencia:
Norma Santos
Departamento de Virologia,
Instituto de Microbiologia Paulo de Góes
Universidade Federal do Rio de Janeiro
Cidade Universitária, CCS - Bl. I, Ilha do Fundão
CEP: 21.941-590
Rio de Janeiro-Rio de Janeiro-Brasil
Tel: + 55 (21) 2560-8344 Fax + 55 (21) 2560-88028
E-mail:nsantos@micro.ufrj.br
Received / Recebido em / Recibido en: 3/5/2011
Accepted / Aceito em / Aceito en: 16/9/2011










 texto em
texto em