Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Pan-Amazônica de Saúde
versión impresa ISSN 2176-6215versión On-line ISSN 2176-6223
Rev Pan-Amaz Saude v.2 n.3 Ananindeua sep. 2011
http://dx.doi.org/10.5123/S2176-62232011000300004
ARTIGO ORIGINAL | ORIGINAL ARTICLE | ARTÍCULO ORIGINAL
Genotipagem da resistência genotípica secundária aos antirretrovirais em pacientes com aids nos Estados do Pará e Amazonas, Brasil: 2002 a 2006*
Profile of secondary genotypic resistance to antiretroviral drugs in aids patients in the States of Pará and Amazonas, Brazil: 2002 to 2006
Genotipado de la resistencia genotípica secundaria a los antirretrovirales en pacientes con aids en los Estados de Pará y Amazonas, Brasil: 2002 a 2006
Olinda MacêdoI; Luciana Macedo FerreiraI; Pedro Fernando da Costa VasconcelosII; Rita Catarina Medeiros de SousaIII; Carmen Andrea FreitasIV; José Ricardo Mourão AraújoV
ISeção de Virologia, Instituto Evandro Chagas/SVS/MS, Ananindeua, Pará, Brasil
IISeção de Arbovirologia e Febres Hemorrágicas, Instituto Evandro Chagas/SVS/MS, Ananindeua, Pará, Brasil
IIINúcleo de Medicina Tropical, Universidade Federal do Pará, Belém, Pará, Brasil
IVHospital Universitário João de Barros Barreto, Universidade Federal do Pará, Belém, Pará, Brasil
VCasa Dia, Secretária Municipal de Saúde, Belém, Pará, Brasil
Endereço para correspondência
Correspondence
Dirección para correspondencia
RESUMO
A resistência às drogas antirretrovirais resulta da incompleta supressão da replicação do HIV-1. O presente estudo caracterizou o perfil de resistência genotípica aos antirretrovirais (ARV) em amostras sorológicas de 127 pacientes HIV positivos, originárias dos Estados do Amazonas e Pará, Região Norte do Brasil, no período de 2002 a 2006. As amostras foram submetidas ao teste de resistência pelo kit ViroSeqTM Genotyping System. Considerando as informações genéticas obtidas das regiões da protease e/ou transcriptase reversa do HIV-1, a mutação M184V (81,1%) foi a mais associada aos inibidores nucleosídicos da transcriptase reversa (ITRN), em indivíduos usando ARV no Estado do Pará, e a mutação T215F/Y (56,3%) em indivíduos do Estado do Amazonas. A mutação K103N foi a mais prevalente (33,5%) para os inibidores não nucleosídicos da transcriptase reversa (ITRNN) em ambos os Estados. Para o gene da protease a mutação minor L63P (65,3%) apresentou-se como a mais frequente em ambos os Estados. O estudo revelou a importância da identificação de mutações associadas à resistência às drogas antirretrovirais para o uso racional em esquemas terapêuticos e seus resultados apresentaram-se semelhantes aos obtidos em outras regiões do Brasil.
Palavras-chave: Terapia Antirretroviral; HIV-1; Mutação; Genotipagem.
ABSTRACT
Resistance to antiretroviral drugs results from the incomplete suppression of HIV-1 replication. The present study characterized the profile of genotypic resistance to antiretroviral drugs (ARVs) in serum samples from 127 HIV-positive patients from the States of Amazonas and Pará, in Northern Brazil, from 2002 to 2006. The samples were tested for resistance using the ViroSeqTM Genotyping System kit. Based on the genetic information obtained from the HIV-1 protease (PR) and/or reverse transcriptase (RT) genes, the M184V mutation (81.1%) was the most frequently associated with resistance to nucleoside reverse transcriptase inhibitors (NRTIs) in individuals using ARVs in Pará, while the T215F/Y mutation (56.3%) was the most frequently associated with resistance in individuals from Amazonas. The K103N mutation was the most prevalent (33.5%) resistance mutation to non-nucleoside reverse transcriptase inhibitors (NNRTIs) in both states. For the PR gene, the minor mutation L63P (65.3%) was the most frequent in both states. The present study showed the importance of identifying mutations associated with resistance to ARVs to the rational selection of therapeutic schemes. Additionally, the results found in Pará and Amazonas were found to be similar to those of other areas in Brazil.
Keywords: Terapia Anti-Retroviral; VIH-1; Mutation; Genotyping.
RESUMEN
La resistencia a las drogas antirretrovirales resulta de la supresión incompleta de la replicación del VIH-1. El presente estudio caracterizó el perfil de resistencia genotípica a los antirretrovirales (ARV) en muestras serológicas de 127 pacientes VIH positivos, originarias de los Estados de Amazonas y Pará, Región Norte de Brasil, en el período de 2002 a 2006. Las muestras fueron sometidas a la prueba de resistencia por el kit ViroSeqTM Genotyping System. Considerando las informaciones genéticas obtenidas de las regiones de la proteasa y/o transcriptasa inversa del VIH-1, la mutación M184V (81,1%) fue la más asociada a los inhibidores nucleósidos de la transcriptasa inversa (INTR), en individuos usando ARV en el Estado de Pará, y la mutación T215F/Y (56,3%) en individuos del Estado de Amazonas. La mutación K103N fue la más prevalente (33,5%) para los inhibidores no nucleósidos de la transcriptasa inversa (INTR) en ambos Estados. Para el gen de la proteasa la mutación minor L63P (65,3%) se presentó como la más frecuente en ambos Estados. El estudio reveló la importancia de la identificación de mutaciones asociadas a la resistencia a las drogas antirretrovirales para el uso racional en esquemas terapéuticos y sus resultados se mostraron semejantes a los obtenidos en otras regiones de Brasil.
Palabras clave: Terapia Antirretroviral; VIH-1; Mutación; Genotipado.
INTRODUÇÃO
A resistência do vírus da imunodeficiência humana tipo 1 (HIV-1) aos fármacos antirretrovirais é consequência da alta taxa de replicação e mutação deste vírus combinada com a sua capacidade de integração com o genoma do hospedeiro. O HIV-1 apresenta uma notável diversidade genética com implicações na patogênese, desenvolvimento de vacina, diagnóstico e suscetibilidade aos antirretrovirais (ARV)1,2,3.
O vírus possui distintos mecanismos que lhe permitem escapar tanto da pressão do sistema imunológico como da pressão farmacológica. O surgimento das quasispecies é favorecido pelos seguintes fatores: escassa fidelidade da transcriptase reversa (TR) em seu trabalho de replicação do RNA viral o que gera uma elevada população de cepas de vírus diversificadas - cerca de 1012 e extraordinária cinética de replicação viral com uma vida média plasmática inferior a 6 h4,5. A contínua produção dessas variantes proporciona ao vírus uma grande capacidade de adaptação no hospedeiro. A resistência antirretroviral é uma das razões primárias para que a terapia antirretroviral (TARV) falhe com o uso prolongado do esquema6,7.
A finalidade da TARV para indivíduos com infecção pelo HIV inclui impedir ou retardar a progressão da imunodeficiência; melhorar a sobrevida dos pacientes, diminuindo a ocorrência de infecções oportunistas; e melhorar a qualidade de vida dos infectados8,9,10. A redução da carga viral no sangue periférico e a reversão da imunodeficiência característica trazem benefícios ao paciente, aumentando a sobrevida do indivíduo em pelo menos 13 a 14 anos11. O esquema terapêutico é composto por uma associação de drogas para obter a diminuição da carga viral plasmática e, assim, atingir níveis virais indetectáveis na circulação periférica12.
As cepas resistentes do vírus HIV-1 podem ser transmitidas entre os indivíduos. A transmissão de variantes do HIV-1, com resistência aos inibidores da TR e da protease (PR), foi amplamente caracterizada em países desenvolvidos. Com efeito, os níveis de transmissão do HIV-1 estudados em pacientes com infecção recente documentada e com outros cronicamente infectados mostraram que de 10 a 20% dos novos pacientes diagnosticados na Europa e EUA correspondem a infecções com cepas de HIV-1 resistentes a pelo menos uma droga13,14,15,16.
Alguns autores17,18 estimaram que em torno de 70% dos indivíduos, embora corretamente tratados, apresentavam carga viral detectável e carregavam consigo resistência no mínimo a um medicamento. A transmissão do HIV-1 resistente a drogas também foi documentada entre todos os grupos com comportamento de risco19.
Sabe-se que a supressão da infecção pelo HIV-1 pelas drogas antirretrovirais é notável. No entanto, se a infecção é ocasionada por um vírus resistente, pode ocorrer a redução da eficácia dos medicamentos empregados no regime de primeira linha20.
A transmissão do HIV resistente aos ARV impacta negativamente a resposta terapêutica de indivíduos virgens de tratamento. Como a estratégia da terapia e utilização de drogas ARV evoluiu, os padrões de mutação transmitida podem mudar. Em investigações envolvendo 40 cidades dos EUA foi demonstrado que a alta prevalência de resistência aos ARV ocorreu amplamente, sugerindo que genotipagem e/ou fenotipagem deveria ser considerada na investigação desses indivíduos, especialmente se a terapia incluir um inibidor não nucleosídico da TR (IRTNN)21.
No Brasil, a distribuição gratuita e universal dos medicamentos ARV pelo Ministério da Saúde (MS), iniciou em 1996, com a introdução do esquema TARV, o qual modificou radicalmente a sobrevida dos portadores de aids22,23. Entretanto Clavel e Hance24, descreveram que, com o uso em massa desses medicamentos, ocorreu aumento do risco de resistência aos ARV, havendo assim a necessidade de ser adotado um monitoramento intenso e contínuo dessa resistência.
O presente estudo avaliou o perfil de resistência em amostras de sangue de indivíduos com aids para melhorar as estratégias de terapia em pacientes que falharam nos regimes terapêuticos prévios nos Estados do Pará e Amazonas, Região Norte do Brasil, no período de 2002 a 2006.
MATERIAL E MÉTODOS
A detecção da resistência primária e secundária aos ARV foi avaliada utilizando-se a lista proposta por Johnson e colaboradores25 para a classificação das mutações. No presente estudo, um largo espectro de códons mutacionais associados à maior ou menor resistência aos ARV foi observado durante o processo de análise conduzida em amostras de pacientes usuários de ARV
CRITÉRIO DE INCLUSÃO
Foram empregados protocolos próprios da Rede Nacional de Laboratórios de Genotipagem (Renageno) os quais continham informações pessoais, clínicas, laboratoriais e epidemiológicas, para, em seguida, fazer-se a escolha do grupo considerando os critérios da rede. Os pacientes concordaram e assinaram um documento contendo o termo de consentimento livre e esclarecido da Coordenação Nacional de DST e Aids do Ministério da Saúde (CN de DST/Aids do MS), concordando em tomar parte do estudo da Renageno.
DESCRIÇÃO DAS AMOSTRAS
Daqueles indivíduos que preencheram os critérios de inclusão foram coletadas amostras de plasma (1 mL) para poder ser efetuado o estudo.
POPULAÇÃO ESTUDADA
Considerando tratar-se de um estudo retrospectivo realizado no período de 2002 a 2006, o material foi processado adotando os procedimentos da Renageno, utilizando-se amostras de 127 pacientes, sendo 95 do Estado do Pará e 32 do Estado do Amazonas.
As medidas de frequência foram analisadas usando o Programa BioEstat 5.026. A análise das variáveis pelo teste do qui-quadrado que apresentaram valores de p menores que 5% (p < 0,05) proporcionaram aderência ao nível de significância estatística.
RESULTADOS
CARACTERÍSTICAS DA POPULAÇÃO ESTUDADA
O estudo incluiu 32 (25,2%) pacientes procedentes do Amazonas, 19 (59,4%) dos quais pertenciam ao sexo masculino e 95 (74,8%) do Pará, 75 (78,9%) dos quais do sexo masculino.
A idade variou de 19 a 72 anos (mediana de 39 anos) sendo que a maioria, 84,3% encontrava-se na faixa etária entre 20 e 49 anos.
Na ocasião da genotipagem, 84 (66,1%) indivíduos de ambos os Estados não apresentavam qualquer sintoma ou sinal clínico de aids. O diagnóstico da infecção pelo HIV-1 foi realizado em 63,8% no período entre 1991 e 1999.
A detecção de carga viral entre 10 mil e 100 mil cópias de RNA/mL foi positiva em 58,3% dos pacientes, com mediana de contagem em 39 cópias de RNA/mL. A contagem de linfócitos TCD4+ em 57,5% dos pacientes no momento de inclusão no estudo encontrava-se abaixo de 200 células/mm3 (mediana 97 células/mm3).
ANÁLISE MOLECULAR
A extração do RNA foi realizada a partir de 1.000 (µL) de plasma pelo método ViroSeqTM HIV-1 Genotyping System, seguindo as instruções do fabricante (Celera Dignostic, 2004). O sequenciamento das amostras foi realizado empregando-se um sequenciador automatizado modelo ABI PRISM® 3100 DNA Analyzer, (Applied Biosystems, EUA) no qual estava acoplado o Software HIV-1 Genotyping System, versão 2.6, para a detecção automática e análise de todas as sequências.
ANÁLISE ESTATÍSTICA
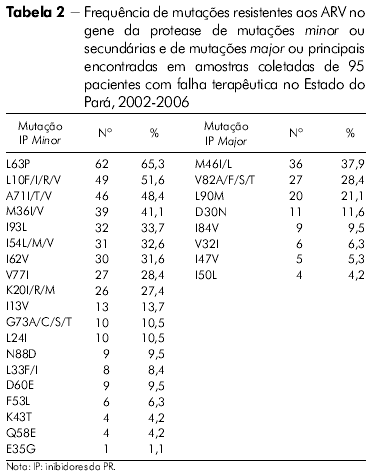
O perfil das mutações de resistência associado ao gene da TR de todos os indivíduos com falha terapêutica no Estado do Pará nas classes dos inibidores nucleosídicos da TR (ITRN), não nucleosídicos da TR (ITRNN) e dos inibidores da protease (IP) pode ser visto nas tabelas 1 e 2.
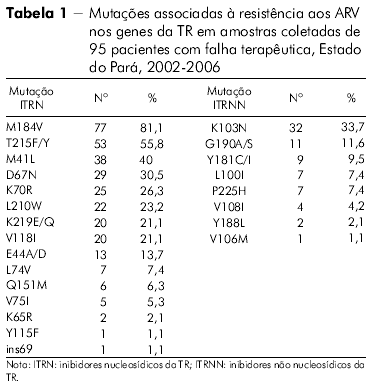
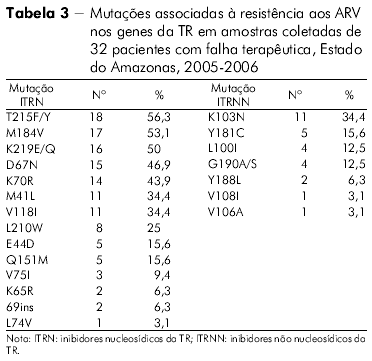
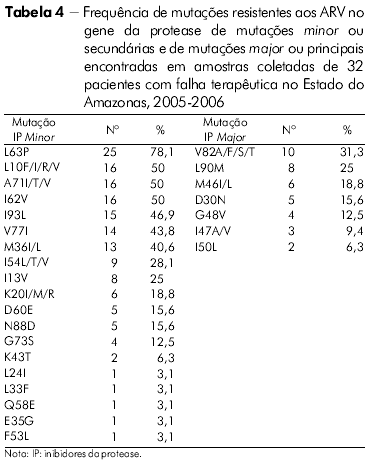
O perfil das mutações de resistência associado ao gene da TR de todos os indivíduos com falha terapêutica no Estado do Amazonas na classe dos ITRN, ITRNN e IP pode ser visto nas tabelas 3 e 4.
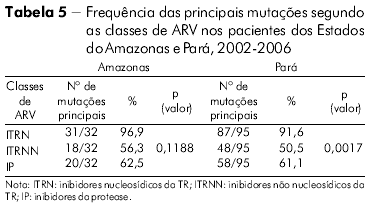
Considerando a presença de mutações principais relacionadas às três classes de ARV (Tabela 5), observou-se maior ocorrência na classe dos ITRN (96,9 %), seguida de IP (62,5%) e ITRNN (56,3%) no Amazonas. No Pará, os percentuais observados nas mesmas classes foram: ITRN (91,6%), IP (61,1%) e ITRNN (50,5%). Não foi observada significância estatística para os ARV com (p = 0,1188) no Amazonas, enquanto no Pará houve significância (p = 0,0017).
Foi inferido o nível de resistência e de sensibilidade do HIV-1 das amostras do Pará aos medicamentos com alvo no gene da TR (Tabela 6). Em ordem decrescente, relacionamos os fármacos mais comprometidos para utilização num próximo esquema pelos pacientes desta análise: Lamivudina, Zidovudina, Zidovudina + Lamivudina, Didanosina (na classe dos ITRN) e Efavirenz e Nevirapina (na classe dos ITRNN). Foi observada significância estatística para a resistência (*R), resistência intermediária (**RI) e (***S) sensível S com p (valor) 0,0001 no Pará pelo teste do qui-quadrado de aderência ao nível de significância 5% (0,05).
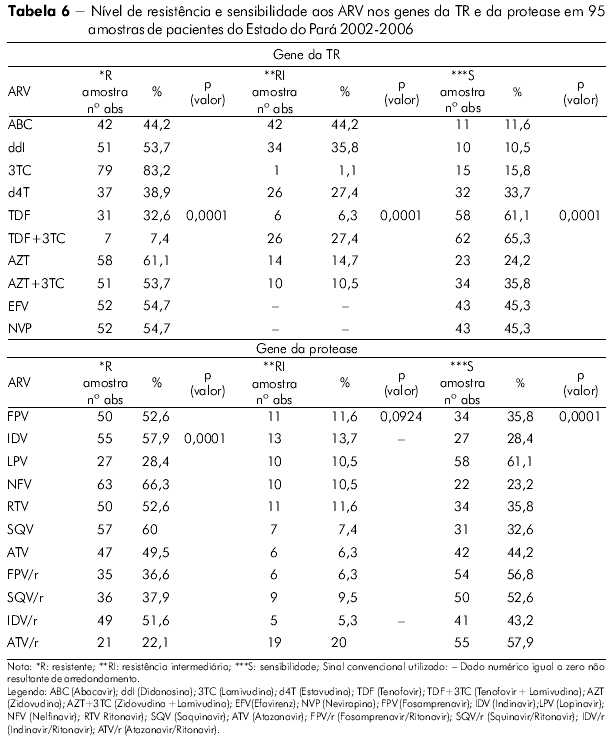
A mesma avaliação foi feita (Tabela 6) com os medicamentos utilizados no gene da PR e observamos os seguintes fármacos mais comprometidos: Nelfinavir, Saquinavir, Indinavir, Ritonavir, Fosamprenavir e Indinavir/Ritonavir. Foi observada elevada significância estatística para a *R e ***S com p (valor) 0,0001 no Estado do Pará pelo teste qui-quadrado de aderência, mas não foi significante para a variável **RI (p = 0,092).
Foi inferido o nível de resistência e sensibilidade do HIV-1 das amostras do Estado do Amazonas aos medicamentos com alvo no gene da TR (Tabela 7). Em ordem decrescente, relacionamos aqueles fármacos mais comprometidos para utilização num próximo esquema pelos pacientes desta análise: Zidovudina, Zidovudina + Lamivudina, Lamivudina, Estavudina, Didanosina e Abacavir na classe dos ITRN e Nevirapina e Efavirenz na classe dos ITRNN. Foi observada significância estatística para a RI e S com p = 0,0042 e p = 0,0001 respectivamente no Amazonas pelo teste qui-quadrado de aderência ao nível de significância 5% (p < 0,05). Não houve significância para a variável R (p = 0,2029).
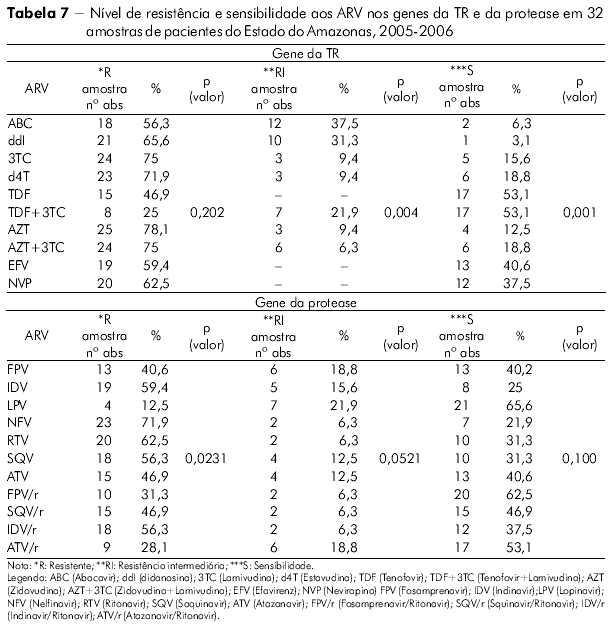
O mesmo acontece (Tabela 7) com os medicamentos utilizados no gene da protease. Observamos os seguintes fármacos mais comprometidos no Estado do Amazonas: Nelfinavir, Saquinavir, Indinavir, Ritonavir, Fosamprenavir e Indinavir/Ritonavir. Foi observada significância estatística para a R p = 0,0231 no Amazonas pelo teste qui-quadrado de aderência ao nível de significância 5% (0,05), não sendo significante para as variáveis RI e S p = 0,0521 e p = 0,100 respectivamente.
DISCUSSÃO
A mutação mais frequentemente encontrada no Estado do Pará, envolvendo os ITRN, foi a M184V, seguida dos códons 215, 41, 67, 70, 210 e 219, perfil este apresentado pelo HIV-1. Por outro lado, a mutação mais frequente nos pacientes do Estado do Amazonas foi a T215F/Y. Em ordem decrescente, os outros códons encontrados foram os seguintes: 184, 70, 219, 67, 41 e 210. O padrão de mutações observadas no Estado do Pará lembra o encontrado no Sudeste e no Nordeste brasileiros27,28, enquanto que o encontrado no Estado do Amazonas foi semelhante ao observado no Chile29, o que sugere que a origem das cepas entre esses dois Estados amazônicos seja provavelmente diferente.
O discreto aumento da prevalência no códon 215, observado nas amostras do Estado do Amazonas, comparado ao códon 184, anteriormente observado, possivelmente deveu-se ao fato de o padrão da mutação M184V sozinha ser encontrado em amostras de apenas dois dos 32 pacientes do Estado do Amazonas. Medeiros e colaboradores30 observaram em pacientes da Região Nordeste do Brasil, que apresentaram um aumento significativo de mutações TAM, quando comparados a grupos de pacientes enfrentando falha terapêutica primária e falha terapêutica secundária com aqueles multifalhados e, associaram esse fato ao maior número de terapias empregadas pelos pacientes multifalhados ocasionando, como consequência, o aumento das referidas mutações.
Ainda na classe dos ITRN, em outro estudo identificou-se que mais de um quinto das amostras de pacientes do Estado do Pará apresentavam a frequência de quatro mutações nas posições 184, 41, 210 e 215, enquanto que no Estado do Amazonas a frequência desses códons foi menor. O padrão de mutação desses códons nos dois Estados da Região Norte brasileira lembra o que foi anteriormente observado em pacientes do Estado do Rio de Janeiro27.
Além das mutações citadas para a classe dos ITRN, outras mutações importantes também foram observadas, porém em menor proporção, nos códons 74, 151, 75, 115, 65, e ins69, no Estado do Pará. As amostras do Estado do Amazonas apresentaram resistência para os mesmos códons com exceção do 115, presente somente nas amostras do Estado do Pará, o que está de acordo com a literatura para outras regiões do Brasil28.
Neste estudo foi detectada a presença de mutação principal para os ITRN em 96,9% das amostras virais do Estado do Amazonas e em 91,6% dos HIV-1 procedentes de amostras do Estado do Pará. Embora menos potente contra o HIV do que os ITRNN e os IP, os ITRN apresentam um papel central no TARV e permanecem como parte do tratamento padrão atual31,32.
Por outro lado, o HIV-1 das amostras do Estado do Amazonas mostrou nível de resistência acima de 50% aos seguintes ARV: Zidovudina, Zidovudina + Lamivudina, Lamivudina, Didanosina e Abacavir, e o HIV-1 das amostras do Estado do Pará apresentava o mesmo nível de resistência aos fármacos Lamivudina, Zidovudina, Zidovudina + Lamivudina e Didanosina. A maior sensibilidade foi observada ao medicamento Tenofovir-acima de 50%, em ambos os Estados. Esses dados são também semelhantes aos descritos para HIV-1 em pacientes procedentes do Nordeste do Brasil30.
Neste estudo foi detectada a mutação K103N na classe ITRNN como a mais prevalente nas amostras dos pacientes dos dois Estados e os valores encontrados são similares aos de outros relatos no Brasil27,28. Ressalte-se que essa mutação ocorreu em mais de 50% dos pacientes que desenvolveram falha virológica sob uso do Efavirenz, reduzindo sua suscetibilidade em aproximadamente 25 vezes33.
No presente estudo detectamos a presença de mutação principal para os ITRNN em 56,3% das amostras do Estado do Amazonas e em 50,5% das amostras do Pará, refletindo no nível de resistência do HIV-1 em 59,4% amostras do Amazonas e 57,4% do Estado do Pará, tanto ao Efavirenz quanto à Nevirapina, as únicas drogas pertencentes a essa classe de ARV avaliadas. Qualquer tipo de mutação detectada nesse grupo de drogas, ocasiona significativa resistência cruzada a todas as drogas, por se tratar de medicamentos que apresentam uma baixa barreira genética24. Esse fato foi também anteriormente observado em diversos outros estudos brasileiros27,28.
Entre os IP, a barreira genética para resistência é geralmente maior. Podemos citar, como exemplo, o regime ARV contendo Ritonavir, que requer múltiplas mutações que variam entre os IP, e seu grau de resistência depende tanto do número quanto do tipo de mutação presente34,35.
As mutações secundárias (minor) normalmente emergem antes das principais (major), e elas por si só não apresentam um efeito significativo no fenótipo25. Em alguns casos, seu efeito pode ser para compensar o fitness replicativo do vírus contendo mutações major e em outros que contribuem para a resistência36,37,25.
No presente estudo, mutações específicas associadas com resistência aos IP foram encontradas nas amostras de pacientes com falha para esses inibidores. A mutação minor L63P foi mais frequentemente encontrada nas amostras do Estado do Amazonas - 78,1%, para 65,1% no Estado do Pará. Em seguida, os códons minor mais encontrados nas amostras dos dois Estados foram 10 e 71, o que está de acordo com resultados obtidos no estudo realizado por Couto-Fernandez e colaboradores27.
CONCLUSÃO
A mutação mais frequente na classe dos ITRN foi M184V para as amostras do Estado do Pará enquanto para as do Estado do Amazonas foi a T21 5F/Y. A mutação mais frequente para as amostras dos Estados do Amazonas e Pará foi K103N, na classe dos ITRNN, e, entre os IP, a mutação L63P para ambos os Estados. Na classe dos ITRN, foi encontrado um percentual significativo de amostras de pacientes do Estado do Pará apresentando quatro mutações nas posições 184, 41, 210 e 215, sendo essa frequência um pouco menor nas amostras do Estado do Amazonas. O HIV-1 se mostrou mais resistente aos ARV da classe dos ITRN, seguido da classe dos IP e, posteriormente, aos ITRNN para as amostras de ambos os Estados. Inferiu-se o nível de resistência do HIV-1 aos ARV no gene da TR e as amostras analisadas do Estado do Amazonas apresentaram resistência acima de 50% para oito drogas, sendo sensíveis a somente duas. No gene da protease, apresentaram-se resistentes a cinco e sensíveis a três das drogas avaliadas. Inferiu-se o nível de resistência do HIV-1 aos ARV no gene da TR e as amostras analisadas do Estado do Pará apresentaram resistência acima de 50% para seis drogas e sensíveis para duas. No gene da protease, mostraram-se resistentes a seis e sensíveis a quatro. As amostras do Estado do Amazonas sugerem maior resistência a todas as classes de ARV analisadas em comparação com as amostras do Estado do Pará.
AGRADECIMENTOS
Aos profissionais do Instituto Evandro Chagas: Raimundo Macêdo dos Reis, Celina Serra de Freitas por todo apoio técnico durante a realização dos testes.
REFERÊNCIAS
1 Kanki PJ, Hamel DJ, Sankalé JL, Hsieh C, Thior I, Barin F, et al. Human immunodeficiency virus type 1 subtypes differ in disease progression. J Infect Dis. 1999 Jan;179(1):68-73. [Link]
2 Caride E, Brindeiro R, Hertogs K, Larder B, Dehertogh P, Machado E, et al. Drug-resistant reverse transcriptase genotyping and phenotyping of B and non-B subtypes (F and A) of human immunodeficiency virus type I found in Brazilian patients failing HAART. Virology. 2000 Sep;275(1):107-15. [Link]
3 Kaleebu P, French N, Mahe C, Yirrell D, Watera C, Lyagoba F, et al. Effect of human immunodeficiency virus (HIV) type 1 envelope subtypes A and D on disease progression in a large cohort of HIV-1-positive persons in Uganda. J Infect Dis. 2002 May;185(9):1244-50. [Link]
4 Eigen M. Viral quasispecies. Sci Am. 1993 Jul;269(1):42-9.
5 Hellerstein MK, McCune JM. T cell turnover in HIV-1 disease. Immunity. 1997 Nov;7(5):583-9.
6 Shafer RW, Winters MA, Palmer S, Merigan TC. Multiple concurrent reverse transcriptase and protease mutations and multidrug resistance of HIV-1 isolates from heavily treated patients. Ann Intern Med. 1998 Jun;128(11):906-11. [Link]
7 Vella S, Palmisano L. Antiretroviral therapy: state of the HAART. Antiviral Res. 2000 Jan;45(1):1-7.
8 Coffin JM. HIV population dynamics in vivo: implications for genetic variation, pathogenesis, and therapy. Science. 1996 Jan;267(5197):483-9.
9 Little SJ, Daar ES, D'Aquila RT, Keiser PH, Connick E, Whitcomb JM, et al. Reduced antiretroviral drug susceptibility among patients with primary HIV infection. JAMA. 1999 Sep;282(12):1142-9. Doi:10.1001/jama.282.12.1142 [Link]
10 Brasil. Ministério da Saúde. Secretaria de Vigilância em Saúde. Recomendações para a terapia anti-retroviral em adultos e adolescentes infectados pelo HIV. Brasília: Ministério da Saúde; 2006.
11 Vermund SH. Millions of life-years saved with potent antiretroviral drugs in the United States: a celebration, with challenges. J Infect Dis. 2006 Jul;194(1):1-5. Doi:10.1086/505154 [Link]
12 Perno CF, Cozzi-Lepri A, Balotta C, Forbici F, Violin M, Bertoli A, et al. Impact of mutations conferring reduced susceptibility to lamivudine on the response to anti retroviral th erapy. Antivir Ther. 2001 Sep;6(3):195-8.
13 Balotta C, Berlusconi A, Pan A, Violin M, Riva C, Colombo MC, et al. Prevalence of transmitted nucleoside analogue-resistant HIV-1 strains and preexisting mutations in pol reverse transcriptase and protease region: outcome after treatment in recently infected individuals. Antivir Ther. 2000 Mar;5(1):7-14. [Link]
14 Little SJ. Transmission and prevalence of HIV resistance among treatment-naive subjects. Antivir Ther. 2000 Mar;5(1):33-40. [Link]
15 Briones C, Pérez-Olmeda M, Rodríguez C, del Romero J, Hertogs K, Soriano V. Primary genotypic and phenotypic HIV-1 drug resistance in recent seroconverters in Madrid. J Acquir Immune Defic Syndr. 2001 Feb;26(2):145-50. [Link]
16 Duwe S, Brunn M, Altmann D, Hamouda O, Schmidt B, Walter H, et al. Frequency of genotypic and phenotypic drug-resistant HIV-1 among therapy-naive patients of the German Seroconverter Study. J Acquir Immune Defic Syndr. 2001 Mar;26(3):266-73. [Link]
17 Tamalet C, Fantini J, Tourres C, Yahi N. Resistance of HIV-1 to multiple antiretroviral drugs in France: a 6-year survey (1997-2002) based on an analysis of over 7000 genotypes. AIDS. 2003 Nov;17(16):2383-8. [Link]
18 Scott P, Arnold E, Evans B, Pozniak A, Moyle G, Shahmenesh M, et al. Surveillance of HIV antiretroviral drug resistance in treated individuals in England: 1998-2000. J Antimicrob Chemother. 2004 Mar;53(3):469-73. Doi:10.1093/jac/dkh102 [Link]
19 Tang JW, Pillay D. Transmission of HIV-1 drug resistance. J Clin Virol. 2004 May;30(1):1-10. [Link]
20 Johnson JA, Li JF, Wei X, Lipscomb J, Irlbeck D, Craig C, et al. Minority HIV-1 drug resistance mutations are present in antiretroviral treatment-naïve populations and associate with reduced treatment efficacy. PLoS Med. 2008 Jul;5(7):e158. Doi:10.1371/journal.pmed.0050158.g002 [Link]
21 Ross L, Lim ML, Liao Q, Wine B, Rodriguez AE, Weinberg W, et al. Prevalence of antiretroviral drug resistance and resistance-associated mutations in antiretroviral therapy-naïve HIV-infected individuals from 40 United States cities. HIV Clin Trials. 2007 Jan-Feb;8(1):1-8. Doi:10.1310/hct0801-1 [Link]
22 Brasil. Decreto no 9.313, de 13 nov. 1996. Dispõe sobre a distribuição gratuita de medicamentos aos portadores do HIV e Doentes de AIDS. Diário Oficial da União, Brasília, p. 1, 14 nov. 1996.
23 Marins JR, Jamal LF, Chen SY, Barros MB, Hudes ES, Barbosa AA, et al. Dramatic improvement in survival among adult Brazilian AIDS patients. AIDS. 2003 Jul;17(11):1675-82. [Link]
24 Clavel F, Hance AJ. HIV drug resistance. N Engl J Med. 2004 Mar;350(10):1023-35.
25 Johnson VA, Brun-Vezinet F, Clotet B, Conway B, Kuritzkes DR, Pillay D, et al. Update of the drug resistance mutations in HIV-1: Fall 2005. Top HIV Med. 2005 Oct-Nov;13(4):125-31. [Link]
26 Ayres M, Ayres Jr M, Ayres DL, Santos AS. BioEstat 5.0: aplicações estatísticas nas áreas das ciências biológicas e médicas. Belém: Sociedade Civil Mamirauá. 2007. 364 p.
27 Couto-Fernandez JC, Silva-de-Jesus C, Veloso VG, Rachid M, Gracie RS, Chequer-Fernandez SL, et al. Human immunodeficiency virus type 1 (HIV-1) genotyping in Rio de Janeiro, Brazil: assessing subtype and drug-resistance associated mutations in HIV-1 infected individuals failing highly active antiretroviral th era py. Mem Inst Oswaldo Cruz. 2005 Feb;100(1):73-8. [Link]
28 Cavalcanti AM, Lacerda HR, Brito AM, Pereira S, Medeiros D, Oliveira S. Antiretroviral resistance in individuals presenting therapeutic failure and subtypes of the human immunodeficiency virus type 1 in the Northeast Region of Brazil. Mem Inst Oswaldo Cruz. 2007 Nov;102(7):785-92. [Link]
29 Afani SA, Orellana RL, Duarte JP, Acevedo MW, Morales BO, Wolff RM, et al. Resistance to antiretroviral therapy in Chilean patients with HIV-1 from 2002 to 200 5. Rev Med Chil. 2007 Oct;135(10):1237-44. Doi:10.4067/S0034-98872007001000002 [Link]
30 Medeiros MS, Arruda EA, Guerrant RL, Brown C, Hammarskjold ML, Rekosh D, Lima AA, et al. Genotype testing and antiretroviral resistance profiles from HIV-1 patients experiencing therapeutic failure in northeast Brazil. Braz J Infect Dis. 2007 Aug;11(4):390-4.
31 Shen L, Peterson S, Sedaghat AR, McMahon MA, Callender M, Zhang H, et al. Dose-response curve slope sets class-specific limits on inhibitory potential of anti-HIV drugs. Nat Med. 2008 Jul;14(7):762-6.
32 Department of Health and Human Services Panel on antiretroviral guidelines for adults and adolescents: guidelines for the use of antiretroviral agents in HIV-1-infected adults and adolescents. [Internet]. USA, 2009. [Cited 2010 Apr 30]. Available from: http://www.antimicrobe.org/h04c.files/history/Guideline-NIH-AdultandAdolescentGL-2009.pdf.
33 Ceccherini-Silberstein F, Svicher V, Sing T, Artese A, Santoro MM, Forbici F, et al. Characterization and structural analysis of novel mutations in human immunodeficiency virus type 1 reverse transcriptase involved in the regulation of resistance to nonnucleoside inhibitors. J Virol. 2007 Oct;81(20):11507-19. Doi:10.1128/JVI.00303-07 [Link]
34 Shafer RW, Schapiro JM. Drug resistance and antiretroviral drug development. J Antimicrob Chemother. 2005 Jun;55(6):817-20. Doi:10.1093/jac/dki127 [Link]
35 Von Wyl V, Yerly S, Böni J, Bürgisser P, Klimkait T, Battegay M, et al. Emergence of HIV-1 drug resistance in previously untreated patients initiating combination antiretroviral treatment: a comparison of different regimen types. Arch Intern Med. 2007 Sep;167(16):1782-90. [Link]
36 Condra JH, Schleif WA, Blahy OM, Gabryelski LJ, Graham DJ, Quintero JC, et al. In vivo emergence of HIV-1 variants resistant to multiple protease inhibitors. Nature. 1995 Apr;374(6522):569-71. Doi:10.1038/374569a0 [Link]
37 Mammano F, Trouplin V, Zennou V, Clavel F. Retracing the evolutionary pathways of human immunodeficiency virus type 1 resistance to protease inhibitors: virus fitness in the absence and in the presence of drug. J Virol. 2000 Sep;74(18):8524-31. [Link]
 Correspondência / Correspondence / Correspondencia:
Correspondência / Correspondence / Correspondencia:
Olinda Macêdo
Instituto Evandro Chagas, Seção de Virologia
Rodovia BR 316, km 7, s/no Levilândia
CEP: 67030-000 Ananindeua-Pará-Brasil
Tel:. +55 (91) 3214 2009
E-mail: olindamacedo@iec.pa.gov.br
Recebido em / Received / Recibido en: 18/04/2011
Aceito em / Accepted / Aceito en: 19/8/2011
*Artigo resultado de dissertação apresentada ao Programa de Pós-Graduação em Biologia dos Agentes Infecciosos e Parasitários do Instituto de Ciências Biológicas da Universidade Federal do Pará, como requisito para a obtenção do grau de Mestre em Biologia de Agentes Infecciosos e Parasitários










 texto en
texto en 
 Curriculum ScienTI
Curriculum ScienTI