INTRODUÇÃO
A Escherichia coli é uma bactéria pertencente à família Enterobacteriaceae, sendo amplamente distribuída na natureza, tendo como principal habitat o trato intestinal humano e animal1,2,3,4,5. A E. coli comensal, que faz parte da microbiota intestinal, não é patogênica e apresenta um importante papel fisiológico para o funcionamento do organismo. Existem seis categorias patogênicas de E. coli que causam infecção intestinal em homens e animais, sendo denominadas de E. coli diarreiogênicas6 que são diferenciadas pela presença de fatores de virulência como adesinas fimbriais e afimbriais, toxinas e invasinas, e classificadas em: E. coli enteropatogênica (EPEC), E. coli enterotoxigênica (ETEC), E. coli enteroinvasora (EIEC), E. coli enterohemorrágica (EHEC) ou E. coli produtora da toxina de Shiga (STEC), E. coli enteroagregativa (EAEC) e E. coli aderente difusa (DAEC)7,8,9.
A EPEC foi a primeira E. coli descoberta em 1940 e ainda hoje é considerada a mais versátil entre as categorias diarreiogênicas e uma das principais causas de diarreia em crianças menores de 5 anos de idade10,11,12,13. Em 1995, a EPEC foi classificada em duas subcategorias: EPEC típica (EPEC-t) e atípica (EPEC-a). As EPEC-t são identificadas pela presença do gene eae (EPEC attaching and effacing) e plasmídio EAF (EPEC adherence factor). As EPEC-a apresentam o gene eae, porém são desprovidas do plasmídio EAF. Tanto EPEC-t como EPEC-a devem ser desprovidas do gene stx (Shiga toxina) que caracteriza a STEC/EHEC14. Por muito tempo, as EPEC-t estiveram associadas à diarreia infantil, mas atualmente observa-se uma redução desta subcategoria e o aumento de isolamento de EPEC-a15,16,17. Cada subcategoria é representada por diferentes sorotipos e as EPEC-t pertencem aos sorotipos: O55:H[6], O86:H34, O111:H[2], O114:H2, O119:H[6], O127:H6, O142:H6 e O142:H34, enquanto que as EPEC-a pertencem aos sorotipos: O26:H[11], O55:H[7], O55:H[34], O86:H[8], O111ac:H[8], O111:H[9], O111:H25, O119:H2, O125:H6 e O128:H2, sendo identificados mais de 200 sorotipos de EPEC-a18.
A EPEC-t representa a categoria original de EPEC com comportamento epidemiológico restrito aos seres humanos e patogenia bem definida. A EPEC-a está associada à diarreia humana tanto em países desenvolvidos quanto em desenvolvimento, pode ser encontrada em diversos animais, alimentos e estar distribuída em diferentes ambientes tanto aquáticos como terrestres2,3,5,19. Apesar de sua ampla distribuição e da sua comprovada participação como causa de diarreia humana, sua patogênese e via de transmissão não estão claramente estabelecidas20.
O mecanismo da patogênese de EPEC é a lesão A/E (attaching and effacing) que envolve genes localizados na região LEE (locus of enterocyte effacement), considerada uma ilha de patogenicidade, onde são encontrados os genes ler (regulador transcricional), esp (proteínas do sistema de secreção do tipo III), tir (translocated intimin receptor - receptor de intimina) e o gene eae/E. coli attachment-effacement (adesina intimina)21,22. Três estágios de interação entre EPEC e a célula podem ser observados: 1) aderência localizada (AL) mediada pela fímbria BFP (bundle-forming pilus) e codificada pelo gene plasmidial EAF; 2) sinais de transdução; e 3) aderência íntima promovida pela intimina (gene eae)7.
Para o diagnóstico da EPEC, inicialmente realiza-se a coprocultura para o isolamento da bactéria que depois será submetida a diferentes testes complementares como: sorotipagem, ensaio de aderência com células HEp-2, prova de FAS (fluorescent actin-staining - coloração fluorescente à actina) e técnicas de biologia molecular (reação em cadeia mediada pela polimerase - PCR e suas variações) que amplificam genes que codificam os fatores de virulência que permitem a identificação e subclassificação de EPEC. Outros fatores de virulência que não estão envolvidos com a classificação desta categoria podem estar presentes e distribuídos entre EPEC-t e EPEC-a19,22,23.
Diante das constantes pesquisas e da diversidade de questionamentos em relação à EPEC, este artigo apresenta uma consolidação de material bibliográfico publicado nas últimas duas décadas, com objetivo de proporcionar aos alunos, profissionais microbiologistas, epidemiologistas, médicos e outros da área biomédica, esclarecimentos sobre a epidemiologia, os principais fatores de virulência e a identificação laboratorial desta importante e versátil categoria de E. coli diarreiogênica e suassubcategorias.
METODOLOGIA
Esta revisão consiste em uma pesquisa bibliográfica exploratória realizada em duas bases de dados científicas: Medical Literature Analysis and Retrieval System Online (Medline/PubMed) e Scientific Electronic Library Online (SciELO). Além dos artigos, teses, dissertações e livros didáticos referentes à área microbiológica foram utilizados como fontes bibliográficas. A estratégia de busca nas bases de dados foi realizada por meio da combinação dos termos "EPEC", "EPEC típica", "EPEC atípica" com os seguintes preditores em português, inglês e espanhol, "fatores de virulência", "patogenicidade", "humanos", "animais", "ambiente", "diagnóstico" e "epidemiologia". O período considerado na busca bibliográfica foi de 1992 a 2013. O critério de busca permitiu a identificação de aproximadamente 400 publicações, que foram inicialmente avaliadas quanto ao título e resumo, sendo selecionadas as que abordavam a combinação dos termos "EPEC", "EPEC típica", "EPEC atípica" com os preditores citados acima. Posteriormente, uma leitura completa das publicações pré-selecionadas foi realizada e o fator relevante para a seleção final dos artigos foi quando o conjunto composto por título, resumo, resultado e discussão forneciam informações para o esclarecimento da epidemiologia, dos principais fatores de virulência e da identificação laboratorial de E. coli enteropatogênica e suas subcategorias. Assim, 98 publicações foram utilizadas nesta revisão, sendo 81 artigos, oito teses, quatro dissertações e cinco livros ou capítulos de livro. Para síntese desta revisão, os resultados foram apresentados de maneira condensada na formade quadros.
RESULTADOS
As EPEC podem ser isoladas de diferentes fontes como em: alimentos crus e processados; animais domésticos e silvestres; ambientes naturais como água, solo e areia de praia; fezes e solo de abatedouro bovino e fazendas. Observa-se, com os resultados deste levantamento, que a maioria dos artigos sobre epidemiologia não difere as EPEC em subcategoria típica ou atípica. Entre os trabalhos que fizeram esta diferenciação, pode-se observar que as EPEC-a são, constantemente, identificadas na água e com frequência em animais domésticos e silvestres, enquanto que as EPEC-t foram identificadas em raras ocorrências entre animais silvestres (como macaco de cativeiro, coiote e cães) e, no ambiente aquático natural, como rios, lagoas e enseadas. Com relação à frequência em humanos, tanto a EPEC-a quanto a EPEC-t foram identificadas em crianças e adultos com diarreia aguda ou sem diarreia. Também foram identificadas poucas ocorrências de infecção concomitante com outros micro-organismos (Quadro 1).
Entre os principais fatos epidemiológicos envolvendo as EPEC, observa-se: a) o aumento da ocorrência em EPEC-a e diminuição de EPEC-t entre amostras de fezes humanas, sendo relatados surtos de diarreia causada por EPEC-a em diversos países; b) a presença de EPEC em animais domésticos evidenciando esses animais como possíveis reservatórios; e c) a classificação de EPEC-a em dois subgrupos com base na classificação sorológica(Quadro 2).
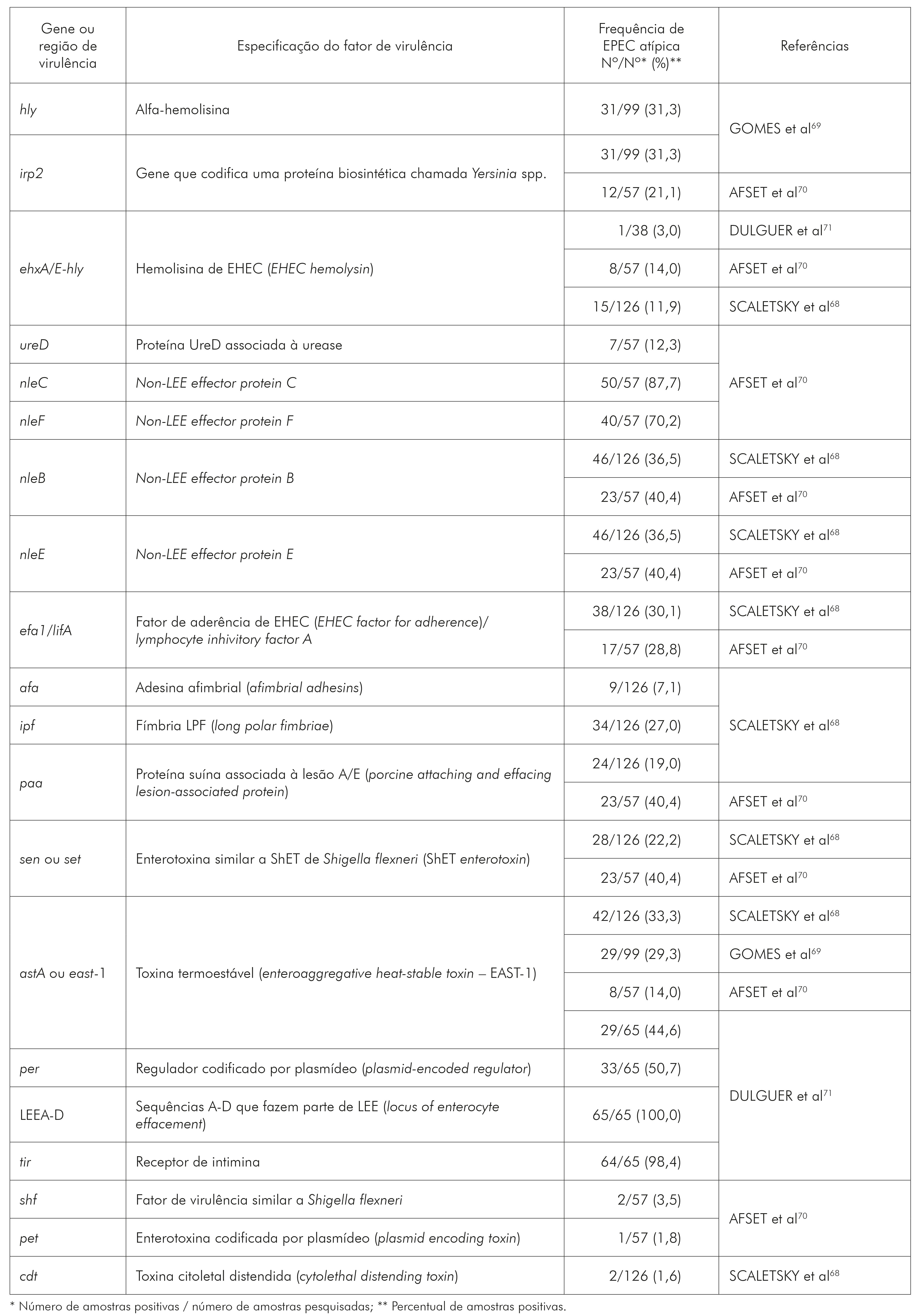
De acordo com o quadro 3, observa-se que os genes da região LEEA-D que codificam o sistema de secreção do tipo III, as proteínas secretadas por este sistema (Esp) e a intimina (eae-EPEC attaching and effacing), e o gene tir que codifica o receptor de intimina, ambos participantes da lesão A/E, foram os genes identificados em EPEC-a com as maiores frequências, 100% e 98,4%, respectivamente. Os demais fatores de virulência observados em EPEC-a são as proteínas efetoras não codificadas pelo LEE- (non LEE-nle), nle C (87,7%) e F (70,2%), seguido de nle-B e E (ambas com variação de frequência entre 36,5% a 40,4%) e o gene per (regulador de LEE localizado em um plasmídeo) com 50%. As frequências aproximadas de 40% a 20% foram observadas para os fatores hly, irp2, efa/lifA, ipf, paa e sen. Frequências abaixo de 20% foram observadas para os demais fatores de virulência (ehxA, afa, shf, pet e cdt). Especificamente para enterotoxina EAST-1, codificada pelo gene astA e pesquisada por vários autores, os percentuais variaram de 14% a 44,6%. Todos esses fatores não relacionados à lesão A/E de EPEC (hly, irp2, efa/lifA, ipf, paa, sen, ehxA, afa, shf, pet e cdt) são frequentemente encontrados em outras categorias patogênicas como E. coli uropatogênica (UPEC), EHEC, EAEC, DAEC, além das EPEC.
De acordo com a revisão bibliográfica, podem-se identificar quatro técnicas laboratoriais utilizadas para o diagnóstico e caracterização de EPEC: a sorotipagem, o teste de adesão em cultura de células, o FAS e as técnicas de biologia molecular. Entre estas técnicas, as únicas utilizadas para diferenciação entre EPEC-t e EPEC-a, são as técnicas de biologia molecular como a PCR convencional, PCR multiplex e PCR em tempo real (Quadro 4). Estas técnicas laboratoriais abrangem metodologias voltadas para aspectos específicos da EPEC como a identificação de antígenos (sorotipagem) e genes codificantes de fatores de virulência (PCR e suas variações), bem como, aspectos voltados à investigação da interação da bactéria com as células, como a formação da lesão A/E e condensamento de actina constatados no teste de FAS e a observação e classificação do tipo de adesão que as EPEC promovem em cultura de células.
A identificação e a diferenciação entre EPEC-t e EPEC-a compreendem, principalmente, critérios moleculares como a pesquisa dos genes eae, stx, bfp e a sequência EAF, sendo o perfil eae+stx−EAF+bfp+ identificado para EPEC-t e o perfil eae+stx−EAF−bfp−/+ para EPEC-a. Aspectos importantes que contribuem para melhor caracterização de EPEC-t e EPEC-a são: a observação da formação da lesão A/E que é encontrada nestas duas subcategorias; a expressão de BFP, o tipo de aderência promovido em cultura de células, sendo o padrão único de AL encontrado nas EPEC-t; e padrões variados encontrados entre as EPEC-a (Quadro 5).
DISCUSSÃO
A EPEC é uma das principais causadoras de enteroinfecções em crianças e adultos e, nas últimas décadas, estudos sobre esta categoria apontam que esse patógeno está cada vez mais presente na vida de diversas populações, interagindo e se adaptando ao ambiente, ao ponto de serem classificadas em duas subcategorias distintas: EPEC-t eEPEC-a43,48,53,68.
As EPEC-t estiveram por longo tempo associadas à diarreia infantil, no entanto, o atual cenário etiológico demonstra redução na prevalência desta subcategoria em detrimento do aumento dos isolamentos de EPEC-a em casos de diarreia15,16,17, inclusive como única categoria de E. coli diarreiogênica associada à diarreia90. Além deste fato, constatou-se que recentes surtos ocorridos em humanos em diferentes localidades foram ocasionados porEPEC-a51,53,58,62,63.
Além dos seres humanos, as EPEC podem ser identificadas em uma diversidade de animais e ambientes naturais, porém este comportamento epidemiológico pode ser diferenciado dependendo da subcategoria de EPEC. Segundo Oh et al41,42, Tóth et al35, Ibenyassine et al46 e Chandran e Mazumder43, as EPEC-a são identificadas com frequência em águas de rios, enseadas, lagoas e regiões portuárias, além de estarem presentes em animais domésticos e silvestres. Os mesmos estudos apontam a presença das EPEC-t nos mesmos ambientes relatados acima, porém, sua ocorrência entre os animais é rara, tendo sido identificadas apenas em animais silvestres criados em cativeiro, como o macaco37, coiotes e cães de uma área de bacia hidrográfica no Canadá43. Estes dados apontam para uma diferença comportamental entre as duas subcategorias que, provavelmente, influenciam o modo de transmissão para oser humano.
Os artigos de revisão de Hernandes et al20 e Sousa91, ressaltam o alto índice de isolados de EPEC-a em animais domésticos e silvestres, embora não haja evidência de transmissão direta de animais para seres humanos, algumas estirpes de EPEC-a isoladas de animais pertencem aos sorogrupos implicados em doenças humanas, por exemplo O26, O103, O119, O128 e O142.
Ao longo de muitos anos, o homem tem mantido um estreito contato com os animais, porém esta convivência tem se intensificado, principalmente em relação aos animais domésticos e, apesar de cães e gatos terem sido animais de companhia por milhares de anos, nos dias atuais desempenham um importante papel para a sociedade humana32. O problema dessa convivência é que tanto os animais domésticos quanto os seus donos tornam-se possíveis reservatórios de doenças como gastroenterite humana33. Um importante achado epidemiológico sobre as EPEC-a é a presença delas em animais domésticos saudáveis ou diarreicos2,31, aumentando as chances de transmissão para humanos, mas mesmo com sua ampla distribuição e sua comprovada participação como causa de diarreia humana, sua via de transmissão não está claramente estabelecida20.
Em contraste com a epidemiologia das EPEC-a, Hernandes et al20 e Sousa91 descrevem o ser humano como principal reservatório de EPEC-t, sendo raramente identificada entre os animais. Esta observação está de acordo com outros estudos que consideram os seres humanos como único reservatório de EPEC-t, com exceção de alguns relatos de amostras isoladas de cães44 e macacos37.
Estudos epidemiológicos que demonstram a frequência destes patógenos em diferentes animais e no ambiente são importantes para revelar que as EPEC-a, assim como a EHEC (categoria zoonótica de E. coli), podem ser observadas em diferentes hospedeiros animais e indicam que eles podem ser um reservatório destes patógenos com potencial para contaminar as águas superficiais e impactar na saúde humana43. No caso específico de EPEC-a, esse impacto vai depender do perfil de virulência apresentado pela bactéria. Como também demonstrado por Chandran e Mazumder43, a maioria das amostras de EPEC isoladas de animais foram de EPEC-a (60/593: 10,11%), comparado ao número inferior de EPEC-t (8/593: 1,3%) isoladas apenas de coiotes (sete isolados) e cães (um isolado).
Gomes et al69, ao avaliarem 17 sequências gênicas relacionadas a fatores de virulência em EPEC-a (eae+EAF-), identificaram 34 diferentes perfis de virulência sendo os mais frequentes: eae (31,3% - 31/99), eae hly astA pet irp2 (8,1% - 8/99), eae hly (6,1% - 6/99), eae shf (5,1% - 5/99), eae irp2 (5,1% - 5/99), eae perA bfpA astA (4% - 4/99), eae perA bfpA (4% - 4/99). Além destes fatores de virulência citados por Gomes et al69, Dulguer et al71, incluindo em sua pesquisa os fatores de virulência comuns em EPEC (lesão A/E), como a região LEE (A-D), o receptor de intimina (Tir) e o gene regulador plasmidial (per), identificaram os perfis de virulência: LEEA-D tir (43% - 28/65), perA LEEA-D tir astA (33,8% - 22/65), perA LEEA-D tir (13,8% - 9/65) como os mais frequentes, observando que, com exceção da toxina AST, os demais fatores de virulência pesquisados estiveram ausentes (hly, pet, afa, ehxA, agg-A). Estes dados indicam, com exceção dos fatores de virulência comuns à lesão A/E, que ocorre variação e flexibilidade dos fatores de virulência acessórios presentesem EPEC-a.
Apesar do gene astA (enterotoxina EAST-1) ser detectado com frequência em EPEC-a (Quadro 3) quando comparada à EPEC-t71 e dos surtos de diarreia relacionados à EPEC-a produtora de EAST-1 (gene astA), Yatsuyanagi et al92 e Afset et al70 observaram associação negativa deste gene (astA) e de outros fatores de virulência (yjaA, ibeA e b1121) com EPEC-a isoladas de casos diarreicos. De acordo com Scaletsky et al68, foram identificados dois marcadores de virulência, os genes para toxina ehxA e a adesina paa, que poderão ser úteis na detecção de EPEC-a e que foram associados positivamente com EPEC-a isoladas de crianças com diarreia70.
Além de ehxA e paa, os genes localizados na ilha de patogenicidade OI-122 (efa1/lifA, nleB, nleE, set/ent) quando tiveram suas frequências comparadas entre EPEC-a proveniente de casos com e sem diarreia, foram associados significativamente às amostras de EPEC-a isoladas de casos diarreicos70. De acordo com Vieira et al93, a presença de ilha de patogenicidade (PAI) OI122 e a ocorrência simultânea dos genes efa1/lifA, sen, nleB e nleE foram estatisticamente associadas à EPEC-a isoladas de casos diarreicos, sugerindo que a detecção completa da PAI OI122 poderia auxiliar na identificação de potenciais estirpes patogênicas de EPEC-a. Enquanto que a presença de PAI OI122 é complementar à patogênese de EPEC-a, as EPEC-a que apresentam o locus LEE completo (A-D) possuem potencial patogênico71, o que foi confirmado por Trabulsi et al14 ao observarem que 75% das EPEC-a LEEA-D+ apresentam potencial para promover a lesão A/E observado pelo teste deFAS positivo.
Entre as EPEC com perfil de virulência (eae+EAF−stx−) classificadas como EPEC-a, Trabulsi et al14 identificaram que a maioria (80% - 72/90) apresentou AL ou suas variações como: a aderência localizada like (LAL) e a aderência localizada após 6 h de ensaio (LA6). No entanto, outros tipos de aderência como a aderência agregativa (AA) e aderência difusa (AD) e a combinação de AL com AA ou AD também foram observadas. Isto indica que o teste de adesão em cultura de células não tem poder discriminatório para diferenciação entre EPEC-t e EPEC-a. O mesmo deve ser utilizado para complementar os estudos de patogenicidade para estasubcategoria.
De acordo com Elias et al94, ao avaliarem a detecção de marcadores de virulência entre 34 E. coli pertencentes aos sorogrupos de EPEC apresentando o fenótipo AA, observaram que apenas duas amostras foram positivas para o gene eae, nas quais os demais marcadores pesquisados foram negativos (bfp, aggR, aaf, shf, astA e irp). Os marcadores mais frequentes entre as amostras eae negativas foram: aggR (35,3%), shf (55,9%), astA (64,7%) e irp (70,6%), característicos de EAEC, sugerindo que apesar das amostras de EPEC-a compartilharem diversos genes de virulência entre as outras categorias patogênicas, incluindo estes fatores frequentes entre EAEC, quando as E. coli apresentaram adesão AA, existiu baixa propabilidade das mesmas serem EPEC-a, contudo a adesão AA não excluiu uma E. coli de ser EPEC-a.
Um estudo com EPEC-a (eae+EAF−stx−) isoladas de pacientes com diarreia sanguinolenta, utilizando o MLST (multilocus sequence typing), os perfis de virulência e a sorotipagem, identificou estas amostras como EHEC que perderam o gene stx durante a infecção95. Este evento é conhecido como interconversão de E. coli A/E, ocasionado pelo ganho ou perda de genes por transferência horizontal ou por meio de elementos genéticos móveis, tornando mais complicada a caracterização das diferentes categorias de E. coli diarreiogênica, principalmente entre as produtoras de lesão A/E que apresentam o gene eae+, como a EPEC e a STEC/EHEC96. Este evento pode influenciar os aspectos epidemiológicos de EPEC-a e justificar a capacidade de adquirir, manter e expressar genes de virulência codificados por outras categorias de E. coli diarreiogênicas97.
Nesta revisão foram identificadas quatro técnicas capazes de identificar E. coli enteropatogênica: a sorotipagem, o teste de adesão em cultura de células, o FAS (fluorescent actin staining) e as técnicas de biologia molecular55,72,73,74,75,80. De acordo com Lluque et al72, são utilizados dois métodos principais: a sorotipagem e a PCR, sendo apenas esta última com potencial para identificação de EPEC. Atualmente a sorotipagem é uma técnica utilizada especificamente para detecção apenas de antígenos somáticos e flagelares, sendo inapropriada para identificação de EPEC e diferenciação das suas subcategorias, pois um mesmo sorogrupo ou sorotipo pode ser EPEC-t ou EPEC-a. Entretanto, essa técnica tem uma importância epidemiológica para descrição dos sorotipos por reservatórios ou regiões geográficas, além de permitir a classificação da EPEC-a em dois subgrupos: sorotipos clássicos, associados com diarreia e as que não são tipáveis pertencentes aos sorotipos nãoclássicos23,24,68,73.
O diagnóstico molecular é capaz de identificar EPEC e diferenciar EPEC-t de EPEC-a e os genes alvos utilizados neste diagnóstico são: eae, que está presente nas duas subcategorias de EPEC; o gene bfp, presente apenas na EPEC-t; e os genes stx, que são ausentes entre as EPEC, pois são específicos de EHEC/STEC (E. coli enterohemorrágica)20. Para identificação de EPEC-a é mais comum o perfil EAF−e bfp−, porém, algumas EPEC-a podem apresentar o gene bfp (EAF− e bfp+), isso acontece devido à existência de um plasmídeo EAF defectivo não detectado pela PCR, o que leva, na maioria das vezes, à ausência de expressão da fímbria BFP detectada molecularmente81.
De acordo com Trabulsi et al14, das 99 amostras EPEC consideradas atípicas (eae+EAF−stx−) e testadas para o gene bfp, 14,1% (14/99) foram positivas, no entanto apenas duas destas expressaram a fímbria BFP e carreavam o gene perA, significando que as EPEC-a podem expressar gene bfp mesmo com ausência do plasmídeo EAF, porém não apresentam AL em 3 h de ensaio, como observado em EPEC típicas. Baseando-se nesta informação, as EPEC-a podem expressar fímbria BFP, porém sem apresentar AL, ou podem apresentar AL que independe da presença da fímbria BFP. As observações de Trabulsi et al14, de que alguns sorotipos como O142:H6, O119:H2 e O128:H2 apresentam bfp com EAF defectivo (EAF−), sugerem que a melhor característica para distinção de EPEC-t e EPEC-a seria a produção ou não da fímbria BFP, respectivamente, todavia a expressão desta fímbria pode não estar associada ao padrão de adesão localizada característico de EPEC-t.
Após extensas análises genotípicas e fenotípicas, Vieira et al98 concluíram que amostras de EPEC-a (eae+EAF−stx−) de sorogrupos não clássicos compreendem um grupo heterogêneo composto por EPEC-a, EAEC, DAEC e UPEC que adquiriram o locus LEE por transferência horizontal, ou representam EPEC-t que perderam o plasmídeo EAF (ou parte dele), EHEC/STEC que perderam o stx, ou E. coli da microbiota normal que adquiriram o locus LEE. Estes achados demonstram os questionamentos para esta subcategoria e evidenciam a versatilidade deste patotipo, que apesar das inúmeras pesquisas ainda necessita de esclarecimentos.
CONCLUSÃO
A EPEC é reconhecida como uma das principais causas da diarreia desde 1940 e, até o momento, continua associada a casos esporádicos e surtos de diarreia infantil, porém apresentando distintos perfis de virulência que permitem classificá-las em duas subcategorias (EPEC-t e EPEC-a) que apresentam comportamento epidemiológico distinto.
As EPEC-t e EPEC-a podem ser encontradas entre humanos, animais e no ambiente, entretanto com algumas diferenças. As EPEC-a são frequentemente encontradas entre humanos e em uma variedade de hospedeiros animais, indicando que os mesmos podem servir de reservatórios e de fontes de contaminação para o homem e o ambiente. As EPEC-t têm como principal reservatório os seres humanos, no entanto já foram registradas raras ocorrências em alguns animais, como macaco, coiote e cachorro.
Os fatores de virulência mais frequentes em EPEC-a são os codificados por genes que participam da lesão A/E localizados na região LEE (A-D) que codificam o sistema de secreção do tipo III, as proteínas secretadas por este sistema (Esp) e a intimina (eae) e o repector de intimina (tir), além das proteínas efetoras não codificadas por LEE (nle). A presença da região LEE completa (LEEA-D) e da ilha de patogenicidade OI-122 (efa1/lifA, nleB, nleE, set/ent), juntamente com os genes da hemolisina (ehxA) e adesina (paa), podem auxiliar na identificação de potenciais estirpes patogênicas de EPEC-a.
As EPEC-a apresentam inúmeros fatores de virulência comuns e específicos de outras categorias patogênicas como UPEC, EHEC, EAEC e DAEC. Esse fenômeno conhecido como interconversão pode ser responsável pelo aumento da frequência epidemiológica de EPEC-a e justificar a habilidade das EPEC-a em expressar diferentes perfis de virulência.
A identificação conclusiva de EPEC é realizada pelo diagnóstico molecular, onde se pesquisam os genes eae, EAF e stx, sendo o perfil eae+EAF+stx− de EPEC-t e o eae+EAF−stx− de EPEC-a. A investigação da presença do gene bfp e a expressão desta fímbria são importantes para esclarecer a patogênese de EPEC-a e melhor definir estes critérios moleculares utilizados para classificação das duas subcategorias de EPEC.