Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista Pan-Amazônica de Saúde
versão impressa ISSN 2176-6215versão On-line ISSN 2176-6223
Rev Pan-Amaz Saude v.7 n.3 Ananindeua set. 2016
http://dx.doi.org/10.5123/s2176-62232016000300004
ARTIGO ORIGINAL
Virus de la varicela zóster: identificación de los genotipos en casos de varicela y herpes zóster en los Municipios de Ananindeua, Belém y Marituba, Estado de Pará, Brasil
1Núcleo de Medicina Tropical, Universidade Federal do Pará, Belém, Pará, Brasil
2Seção de Virologia, Instituto Evandro Chagas/SVS/MS, Ananindeua, Pará, Brasil
3Seção de Bacteriologia e Micologia, Instituto Evandro Chagas/SVS/MS, Ananindeua, Pará, Brasil
Este es un estudio de tipo transversal, en que el criterio clínico para inclusión de las muestras fue la sospecha clínica y/o aparición de síntomas de varicela o herpes zóster para identificación de los genotipos del virus de la varicela zóster (VVZ), por intermedio de las técnicas de reacción en cadena de la polimerasa (PCR) y secuenciación, con posterior estudio de polimorfismo de nucleótido único (SNP). De noviembre de 2008 a marzo de 2012, se recolectaron 93 muestras (85 casos de varicela y ocho de herpes-zóster) de hisopado nasal, oral y de secreción de vesículas rotas en varios sitios anatómicos (49 de individuos del sexo masculino y 44 del femenino). Las estirpes Dumas y Oka parental fueron incluidas como referencia estándar de los genotipos Europeo (clase 1 o 3) y Japonés (clase 2), respectivamente. Estirpes con variabilidad en el SNP se clasificaron dentro de un grupo de genotipo denominado Mosaico (clase 5). Las clases 1 o 3 fueron identificadas en 11 muestras del VVZ, nueve casos de varicela y dos casos de herpes-zóster, mientras que dos muestras presentaron el genotipo de clase 5, ambos casos de varicela. El genotipo clase 1 o 3 se encontró en los Municipios de Ananindeua, Belém y Marituba, Estado de Pará, Brasil, siendo que el genotipo clase 5 solamente en Ananindeua. La PCR fue sensible en 13 muestras de material directo de vesículas, mientras que en muestras saliva no se obtuvo suceso.
Palabras clave: Virus de la Varicela-Zóster; Varicela; Herpes Zóster; Genotipos; Reacción en Cadena de la Polimerasa; Polimorfismos de Nucleótido Simple
INTRODUCCIÓN
El herpesvirus humano-3 (HHV-3), también conocido como virus de la varicela-zóster (VVZ) de la subfamilia Alphaherpesvirinae, género Varicellovirus, presenta como principal característica la capacidad de establecer latencia. La partícula intacta del VVZ mide de 180-200 nm de diámetro, con genoma lineal de ADN doble cadena de 125 kb, contenido en un duramen de 75 nm dentro de una nucleocápside. Así como otros herpesvirus, el VVZ tiene regiones únicas largas y cortas, una región de repetición terminal (TR) y otra invertida (IR), dentro de las cuales se encuentran los genes duplicados, además de las regiones internas de repeticiones R1 a R51,2.
El VVZ puede causar varicela durante la infección primaria, estableciendo posteriormente una infección latente. En diversas situaciones, en las que ocurre una disminución de la inmunidad celular específica contra el VVZ puede haber una reactivación de la replicación viral, con posibilidad de manifestación clínica por medio del herpes zóster3. La vacuna con el VVZ atenuado estirpe Oka/Merck, licenciada en los Estados Unidos de América (EUA), cuando aplicada en dos dosis con un intervalo de tres meses, presenta 85% de eficacia en la prevención contra la varicela y 95% contra enfermedad grave que requiere terapia antiviral, siendo bastante segura y bien tolerada por niños saludables. Eventos adversos, tales como herpes zóster posvacunal, son raros y ocurren en la mayoría de los casos en inmunocomprometidos4.
Para distinguir la estirpe Oka (utilizada en vacunas) del tipo "salvaje" de VVZ, diversos estudios combinaron las técnicas de polimorfismo en el largo de fragmentos de restricción (RFLP) y reacción en cadena de la polimerasa (PCR) para producir métodos de laboratorio con relativo grado de éxito para uso en la rutina. Esos métodos muestran la divergencia genética en las ORF38 y ORF54 del genoma viral, las que tienen como producto, respectivamente, las siguientes glicoproteínas (gp): gp40, una proteína tegumentaria que interactúa con los microtúbulos de la célula en la formación de la partícula viral y gp55, una proteína tegumentaria estructural denominada de proteína portal de la cápside5,6,7,8,9.
A través de técnicas de PCR y RFLP, dos regiones clivadas por enzimas de restricción correspondientes a polimorfismos en polimorfismo de nucleótido único (single nucleotide polymorphisms - SNP) del VVZ han funcionado como poderosa referencia puntual para la caracterización epidemiológica y geográfica de las variaciones de las estirpes10. SNPs son regiones que contienen variaciones únicas de pares de bases10. En el caso del VVZ, a partir de 2000, sus SNPs se caracterizaron a partir de metodologías de genotipado publicadas por dos grupos, uno norteamericano y otro inglés11,12,13,14.
Las dos metodologías de SNP basadas en genotipado difieren en sus enfoques. El método de SNP disperso, desarrollado por el Grupo de Breuer, en el Reino Unido, se basa en ensayos de movilidad heterodúplex para identificar 92 polimorfismos en 37 ORFs distribuidos uniformemente por el genoma del VVZ. Ese grupo de investigación identificó tres genotipos filogenéticamente distintos circulando en el Reino Unido y un cuarto en Japón2,11,14.
El grupo norteamericano (Universidad de Iowa, Iowa, EUA), liderado por Grose, hizo la secuenciación de cinco genes de glicoproteínas, entre esos la ORF que codifica la mayor proteína de transactivación inmediata (IE62)15. Sesenta y un polimorfismos, 21 en las glicoproteínas y 40 en la ORF62, fueron identificados12. El análisis filogenético de esas regiones identificó cuatro clases16.
Un tercer grupo de investigación, liderado por Schmid en el Centers for Disease Control and Prevention (CDC), localizó un fragmento de 447 pb en la ORF22, región denominada de "up stream" C-terminal, el cual fue suficientemente polimórfico para permitir la identificación de cuatro subtipos virales13. Loparev et al13 analizaron variaciones genotípicas utilizando un fragmento de 447 pb de la región ORF22 (específicamente las posiciones nucleótidas 37902, 38055, 38081, 38177 y 38229), que tienen como producto la gp24, una gran proteína estructural que hace parte del tegumento viral, con localización subcelular en el virión1,13. La diversidad nucleótida de esta secuencia se usó para definir el genotipo de 326 aislados de VVZ de muestras de cinco continentes13.
El uso de las endonucleasas de restricción PstI y Bgll, permitió identificar los sitios de clivaje para estas enzimas en la ORF38 y ORF54, respectivamente. Diversas estirpes aisladas en los EUA contienen un sitio de restricción de la enzima PstI en la región ORF38, en donde ocurre una sustitución del nucleótido guanina (G) por adenina (A) en la posición 69349 que la estirpe Oka vacunal no posee5. Además, una sustitución del nucleótido citosina (C) por timina (T) en la posición 95241 resulta en la eliminación del sitio de restricción de la enzima BglI, localizada en ORF54, la cual se encuentra en todas las estirpes japonesas y que está ausente en muchos aislados de los EUA6,7,8,9. De esa manera, las estirpes japonesas pueden presentar los genotipos PstI+ BglI+ o PstI- BglI+, mientras que muchos VVZ aislados en los EUA son PstI+ BglI-6,7,8,9.
En el año 2007, el análisis de esas tres ORFs permitió la clasificación de los genotipos del VVZ por distribución geográfica en: europeo (E), japonés (J) y mosaico (M)17.
En total, esos tres grupos de investigación identificaron cinco subtipos virales distintos, aunque ningún grupo haya identificado a todos los cinco. De acuerdo con la conferencia VZV Nomenclature Meeting 20082, realizada el 25 de julio de 2008 en Whitechapel, Londres, Reino Unido, las cepas de VVZ detectadas y caracterizadas por secuenciación de los SNPs serían agrupadas en clases de 1 a 5, sustituyendo los genotipos preconizados en la clasificación del CDC. Las diferentes nomenclaturas utilizadas por cada grupo se presentan en el cuadro 1.
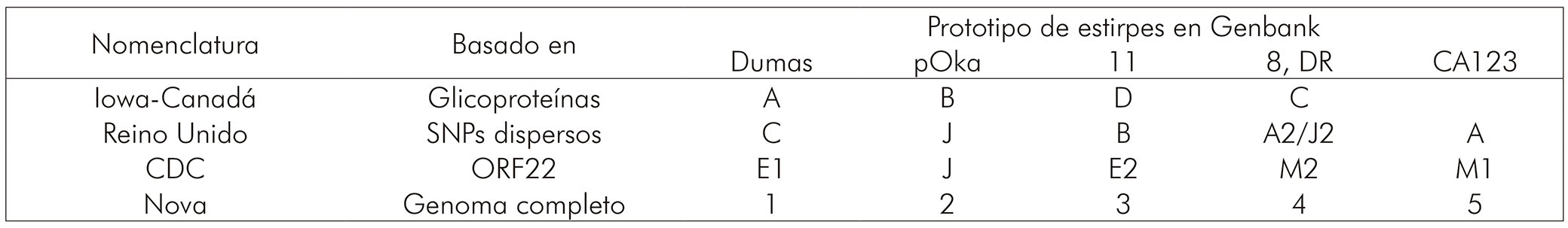
Fuente: Adaptado de Breuer et al2, 2010.
Cuadro 1 - Nomenclatura genotípica del VVZ utilizada actualmente
Loparev et al18 identificaron SNPs que discriminan los genotipos E1 y E2 en las ORF21, ORF22 [región 2], y ORF50. Un análisis de SNP en cualquiera de estas regiones es suficiente para discriminar cepas E1 de otras europeas. Dos posiciones adicionales en ORF38 (69349) y ORF54 (95241), originariamente usadas para confirmar los eventos adversos de la vacuna Oka, también ofrecen informaciones para genotipado. Esos dos marcadores no son útiles para la vacuna Oka específicamente, pero sirven para distinguir algunas cepas J, M1, M2 y M419 de otros genotipos. Un subconjunto de aislados de genotipo J (por ejemplo, pOka, que fue la fuente para la vacuna de virus Oka) contiene un sitio de restricción PstI en la ORF38 y ORF54 incluyendo la secuencia de un local de restricción Bgll común a genotipos J, M1, M2 y M42,13.
El genotipo E fue identificado en cepas obtenidas de pacientes de Alemania, Islandia, República Checa, Polonia, Rusia europea, Bielorrusia, Ucrania, Lituania, Letonia, Moldavia y Estonia13,20. Ese genotipo también fue el más prevalente en Asia, Rusia, Kazajistán, este de Australia, Canadá, Estados Unidos, Jordania, Chile y Nicaragua, mostrándose altamente cosmopolita. Con base en el análisis de los aislados obtenidos a partir de varios locales geográficos distintos, el genotipo E mostró un grupo de bases conservadoras en el perfil de los SNPs de la ORF22 y todas las cepas E también presentaron el estándar de marcador PstI Bgll. Una observación sorprendente fue el descubrimiento del dominio geográfico de cepas del genotipo M en áreas tropicales y subtropicales, como África (Congo, República de Chad, Marruecos y Guinea Bissau) y en Asia (India, Bangladesh, Nepal y China)13,21.
Además de las cepas J comunes en Japón, Australia y algunos países del este asiático, los estándares Bgl + también son comúnmente aislados en climas tropicales (por ejemplo, en África Ecuatorial, India, Bangladesh, en el sur de China, América Central y norte de Australia). En contraste, virus Bgl - son comunes en climas templados. Dos aislados Pst - Bgl - también fueron observados en Austrália13,18,19,22. De esta forma, para dominancia geográfica, se observaron los genotipos E1 y E2 (Europa), M1 (África) y J (Japón). El predominio de cepas genotipo E en América del Norte y Australia probablemente, se estableció durante la colonización europea en los siglos XVI y XVII. Cambios relativamente recientes en la inmigración y/o patrones de viajes internacionales para ciertas regiones geográficas (principalmente Europa, Australia y Estados Unidos) han ocasionado la cocirculación de una variedad de genotipos de VVZ13,17,19,20,23,24,25,26,27.
Un genotipo propuesto como recombinante de VVZ de muestras de Brasil fue clasificado como una estirpe del genotipo M19. De diez aislados obtenidos de forma independiente en Brasil, ocho estirpes tipo M y dos del genotipo E tenían estándares de SNP similares a los descritos por Barrett-Muir et al13,28.
En Brasil, la mayor parte de los estudios involucrando varicela y herpes-zóster considera apenas los aspectos sintomatológicos29,30,31,32,33. Siendo así, este estudio tuvo como objetivo permitir el conocimiento de estirpes circulantes del VVZ en la región metropolitana de Belém, Estado de Pará, Brasil y poner a disposición informaciones importantes de identificación y distribución de los genotipos del VVZ, en las diversas franjas etarias investigadas y/o asociadas a una determinada patología.
MATERIALES Y MÉTODOS
ÁREA DE ESTUDIO
Las muestras para este estudio se obtuvieron en los Municipios de Ananindeua, Belém y Marituba, localizados en la región metropolitana de Belém, Estado de Pará.
POBLACIÓN DEL ESTUDIO
Estudio de tipo transversal en el que el criterio clínico para inclusión de las muestras fue la sospecha clínica y/o aparecimiento de los síntomas de varicela o herpes-zóster. Durante el período de noviembre de 2008 a marzo de 2012, se recolectaron 93 muestras de hisopado nasal/oral (49 de individuos del género masculino y 44 del femenino). De esos 93 individuos, 26 (13 de individuos del género masculino y 13 del femenino) presentaron vesículas rotas, de las cuales se recolectaron los hisopos. Todas las muestras, oriundas de pacientes procedentes de las redes pública y privada de salud, fueron recolectadas en el Instituto Evandro Chagas (IEC), localizado en Ananindeua, Estado de Pará.
IDENTIFICACIÓN, PROCESO Y ALMACENADO DE LAS MUESTRAS
La recolección de las muestras se realizó en 93 individuos (85 casos de varicela y ocho de herpes zóster), siendo 93 muestras de hisopado nasal/oral y 26 muestras (22 casos de varicela y cuatro de herpes-zóster) de secreción de vesículas rotas en varios sitios anatómicos. Las muestras fueron identificadas y recolectadas el día de la atención y encaminadas a la Sección de Virología del IEC. El período de recolección de las muestras se definió entre el primero hasta el 12º día después del inicio de los síntomas.
En la etapa de proceso, sin la extracción de ácidos nucleicos y la utilización directa del material clínico para etapa de amplificación, las muestras de hisopado nasal, oral y vesículas fueron sumergidas, cada una, en un microtubo conteniendo 100 µL de agua destilada y esterilizada, y el hisopado lavado en 400 µL del mismo tipo de agua, presionándolo contra la pared del tubo para retirar el máximo de partículas virales y, luego de esta etapa, la suspensión se almacenó a -70º C34.
AMPLIFICACIÓN DEL ADN VIRAL POR PCR
Las estirpes Dumas y Oka parental fueron incluidas como referencia estándar de los genotipos E (clases 1 o 3) y J (clase 2), respectivamente. Las estirpes se clasificaron en las clases de 1, 2 o 3 cuando exhibieron los SNPs de uno de los dos genotipos. Algunas estirpes presentaron variabilidades en esas secuencias y fueron clasificadas dentro del grupo de genotipo M (clases 4 o 5)2,13.
Luego del proceso de las muestras de hisopado nasal, oral y/o de vesículas, el material fue utilizado directamente en la PCR. Muestras de células diploides humanas HEp-2 (carcinoma epidermoide humano) fueron usadas como control negativo, mientras que una muestra comercial de la vacuna Varicela Biken® (Sanofi Pasteur, Suita, Osaka, Japón), estirpe vOka, fue utilizada como control positivo para identificación de las ORFs 22, 38 y 54.
La reacción de amplificación de un fragmento de 447 pb de la región ORF22 se realizó en termociclador Gene Amp PCR System 9600 (Applied Biosystems, Foster City, California, EUA) con los oligonucleótidos p22R1f 5'-GGGTTTTGTATGAGCGTTGG-3' y p22R1r 5'-CCCCCGAGGTTCGTAATATC-3'; para amplificación de 350 pb de la región ORF38 fueron utilizados los oligonucleótidos PstA 5'-TTGAACAATCACGAACCGTT-3' y PstB 5'-CGGGTGAACCGTATTCTGAG-3'; y para amplificación de 222 pb de la ORF54 fueron utilizados los oligonucleótidos Nla 5'-GGAACCCCTGCACCATTAAA-3' y Fok 5'-TCCCTTCATGCCCGTTACAT-3'10.
Las mismas condiciones de reacción de PCR se adoptaron para esas dos regiones con volumen final de 50 µL, usando AmpliTaq Gold PCR Master Mix (0,025 U de la enzima GoldTaq ADN polimerasa, tampón II 1X para PCR, 2,5 mM de MgCl2, 200 µM de cada desoxinucleótido trifosfato [Invitrogen, Carlsbad, California, EUA]), 0,2 µM de cada oligonucleótido, y 5 µL de la muestra. Cada etapa incluyó un calentamiento inicial de 96º C por 5 min. para activar la enzima, seguida por 40 ciclos de: 94º C por 1 min., 58º C por 2 min. y 72º C por 2 min.; luego de esa etapa se realizó una extensión final a 72º C por 7 min.10.
ELECTROFORESIS EN GEL DE AGAROSA
La visualización de los productos de la PCR fue por electroforesis horizontal en gel de agarosa a 1,8% conteniendo 6 µL de Sybr Safe (Invitrogen, Carlsbad, California, EUA) e inmerso en tampón tris-borato-EDTA (TBE) 1X. Se mezclaron 10 µL de la reacción de PCR con 1 µL de azul de bromofenol y se aplicaron en el gel para una electroforesis a 100 V y 400 mA por 30 min. Para análisis del patrón de las bandas, se utilizó un marcador de peso molecular de 123 pb (Invitrogen) diluido en tampón TBE 1X10. Los amplicons se visualizaron con el sistema de fotodocumentación Gel Doc 1000 (Bio Rad, Hercules, California, EUA).
PURIFICACIÓN DEL PRODUCTO DE LA PCR
El producto de la PCR fue purificado con el objetivo de la secuenciación de nucleótidos (nt). Para tal, se utilizó el kit QIAquick PCR purification (QIAGEN, Hilden, Alemania) siguiendo el protocolo descrito por el fabricante.
SECUENCIACIÓN DE NUCLEÓTIDOS
Los productos de la PCR fueron secuenciados para determinación del genotipo de los VVZ por la identificación de mutación en el sitio de clivaje de la enzima de restricción PstI, la cual resulta en la sustitución G69349A (ORF38). La mutación C95241T (ORF54) causa la eliminación del sitio de restricción para la enzima BglI. El producto del SNP de la ORF22 se utilizó para análisis de mutaciones en las posiciones nucleotídicas 37902, 38055, 38081, 38177 y 3822910.
En la reacción de secuenciación se realizó una mezcla de 3,2 pmol de cada oligonucleótido correspondiente y 10 ng del producto como fuente. Se hicieron 25 ciclos de 96º C por 10 s, 50º C por 5 s y 60º C por 4 min. Los productos resultantes fueron purificados con columnas giratorias (Princeton Separations, Adelphia, Nova Jersey, EUA) y suspensos en tampón de elución (Applied Biosystems, Waltham, Massachusetts, EUA)10.
Los polimorfismos nucleotídicos específicos do VVZ se analizaron en secuenciador automático, 3130X/Genetic Analyzer (AB Applied Biosystems/Hitachi, Chiyoda, Tokio, Japón) empleando el método Big Dye Terminator Cycle Sequencing Ready Reaction Kit (Applied Biosystems, Waltham, Massachusetts, EUA)10.
EDICIÓN Y ALINEAMIENTO DE LAS SECUENCIAS NUCLEOTÍDICAS
Las secuencias nucleotídicas obtenidas en este estudio se alinearon y editaron usando el programa BioEdit Sequence Alignment Editor v.7.0.5.2 y comparadas con secuencias de otras estirpes del VVZ disponibles en el banco de datos (GenBank - National Center for Biotechnology Information, EUA [www.ncbi.nlm.nih.gov]), usando el aplicativo Basic Local Alignment Search Tool (BLAST). Las secuencias genómicas de cada ORF del VVZ fueron comparadas entre sí, bien como con secuencias de otros herpesvirus (virus herpes simples 1 y 2, virus de Epstein-Barr y citomegalovirus) disponibles en el GenBank10.
Las estirpes Dumas (X04370), HJ0 (AJ871403), CA 123 (número de acceso en el GenBank DQ457052), DR o Moroco 1 (DQ452050), Var160 o México_68 (KC112914.1), pOka (AB097933) y vOka (AB097932) se utilizaron para comparación de las sustituciones nucleotídicas en las muestras analizadas en este estudio10.
ANÁLISIS FILOGENÉTICO
Los dendrogramas se construyeron utilizando el programa MEGA5.1 (Molecular Evolutionary Genetics Analysis, v5.1.3.308). El análisis de la distancia genética entre las secuencias fue calculada por el algoritmo de neighbor-joining (NJ), por el método de Kimura 2-parámetros. El método de bootstrap con 2.000 réplicas fue utilizado para el cálculo de la robustez de los dendrogramas35.
ASPECTOS ÉTICOS Y DE BIOSEGURIDAD
El estudio no ocasionó cualquier riesgo o perjuicio para los participantes o sus responsables. El proyecto involucrado en el presente estudio fue evaluado por el Comité de Ética en Investigación (CEP) del IEC, bien como por la Comisión Nacional de Ética en Investigación, cuando necesario, bajo el n° 0028/2009, con parecer de aprobación de n° 0004/2009. Con relación a los aspectos de bioseguridad, en todos los procedimientos se utilizaron equipamientos de protección individual, siendo que la manipulación de todo material potencialmente infectante se realizó en laboratorio de seguridad NB2, en cabina de seguridad biológica clase 2.
RESULTADOS
SNP UTILIZADOS PARA GENOTIPADO DEL VVZ
Las muestras de hisopado nasal, oral y de vesículas fueron analizadas en la PCR. De 93 muestras, apenas 26 individuos presentaban vesículas y de esos fue recolectado el hisopado. Luego de la realización de la PCR, se amplificaron 50% de las muestras (13/26) e hisopado de vesículas. Las muestras amplificadas y secuenciadas se analizaron con relación a las sustituciones nucleotídicas en cinco posiciones en la ORF22, dos posiciones en la ORF38 y dos posiciones en la ORF54 (Figura 1).
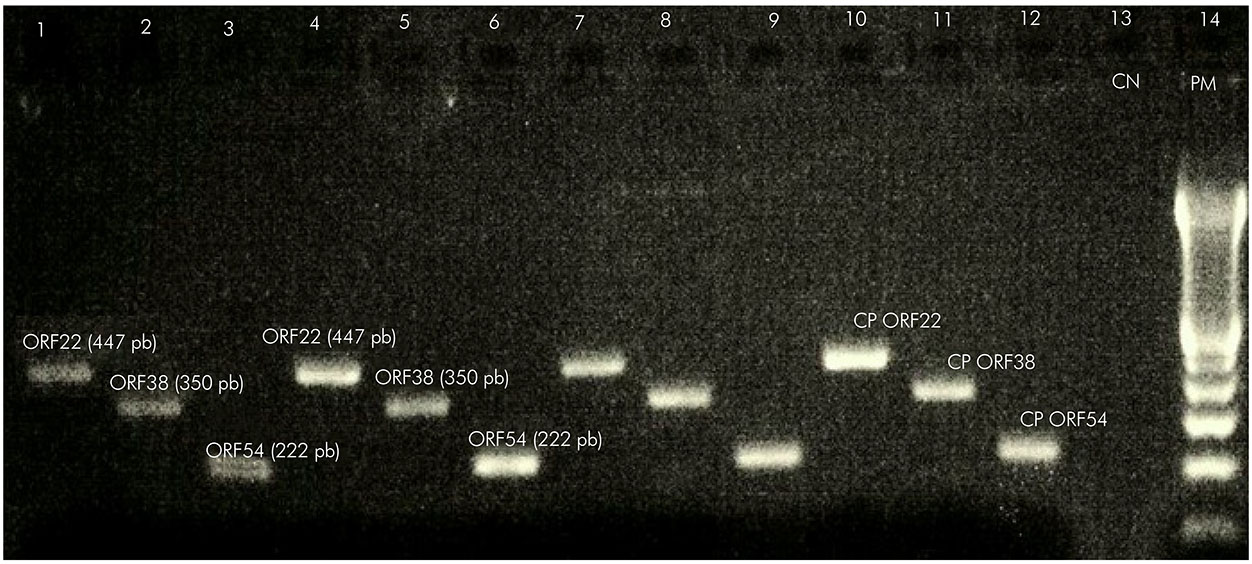
CP: control positivo; CN: control negativo; PM: peso molecular 123 pb.
Figura 1 - Visualización de los productos de las ORFs 22, 38 y 54 luego de PCR
Las muestras VVZ118686 y VVZ119611 presentaron sustituciones nucleotídicas en las posiciones A38081C de la ORF22 y T95241C, C95300T de la ORF54. Las muestras VVZ118683, VVZ118684, VVZ118685, VVZ118845, VVZ119609, VVZ119701 y VVZ119954 presentaron cambio de nucleótido en la posición G69424A de la ORF38 (Cuadro 2).
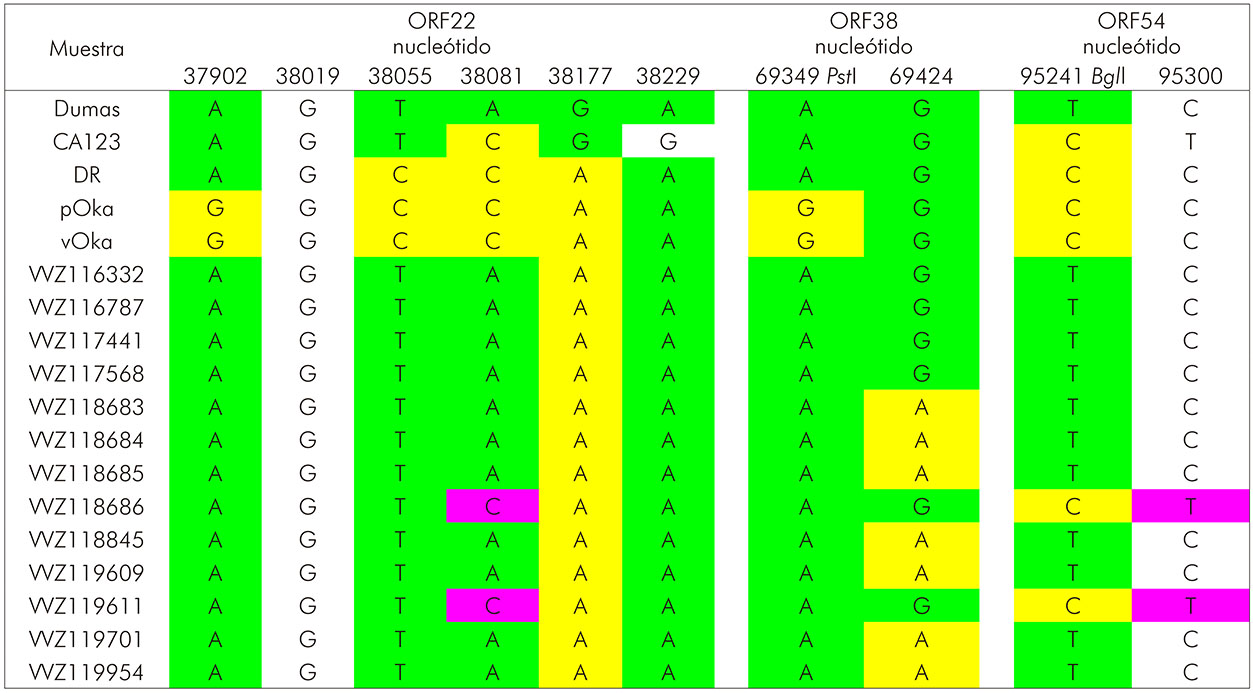
Números en las líneas superiores indican ORF y la posición del SNP en el genoma. Las estirpes a seguir fueron definidas como referencia: Dumas del genotipo E; CA123 y DR del genotipo M; pOka y vOka del genotipo J, para comparación de los pares de bases de posiciones donde hubo cambio de nucleótido. El SNP de la ORF54 es el sitio usado en algunos protocolos para discriminar la estirpe vacunal Oka de muchas estirpes desconocidas. El color lila indica mutación individual intraespecífica; verde indica mutaciones comunes con la estirpe de referencia del genotipo E; amarilla indica mutaciones en común con la estirpe Oka parental genotipo J; blanco indica bases en común para ambas estirpes de referencia E y J.
Cuadro 2 - Análisis de variación genotípica utilizando datos de tres ORFs del VVZ
Los genotipos 1 o 3 se encontraron en 11 muestras VVZ, mientras que dos muestras presentaron el genótipo 5 (Figura 2).

Las estirpes de referencia utilizadas fueron: Dumas, representando el genotipo E; pOka y vOka referencia del genotipo J; VZVi/Philadelphia.USA/73/V[3] referencia del genotipo E; DR y Moroco 1 referencia del genotipo M; y CA 123 referencia del genotipo M. Los valores de bootstrap (2.000 seudorréplicas) están indicados en los nudos del árbol.
Figura 2 - Análisis filogenético utilizando el método de unión de vecinos (NJ) basado en el polimorfismo nucleotídico de la ORF22 (447 pb), involucrando 13 muestras de los Municipios de Belém, Ananindeua y Marituba
El análisis filogenético muestra la distribución de las estirpes dentro de las clases 1 o 3 y clase 5. Estas muestras son oriundas de los Municipios de Ananindeua (siete casos de varicela y uno de herpes-zóster), Belém (tres casos de varicela y uno de herpes-zóster) y Marituba (un caso de varicela). La descripción de las estirpes y las secuencias de las muestras analizadas se registraron en el GenBank (Cuadro 3).
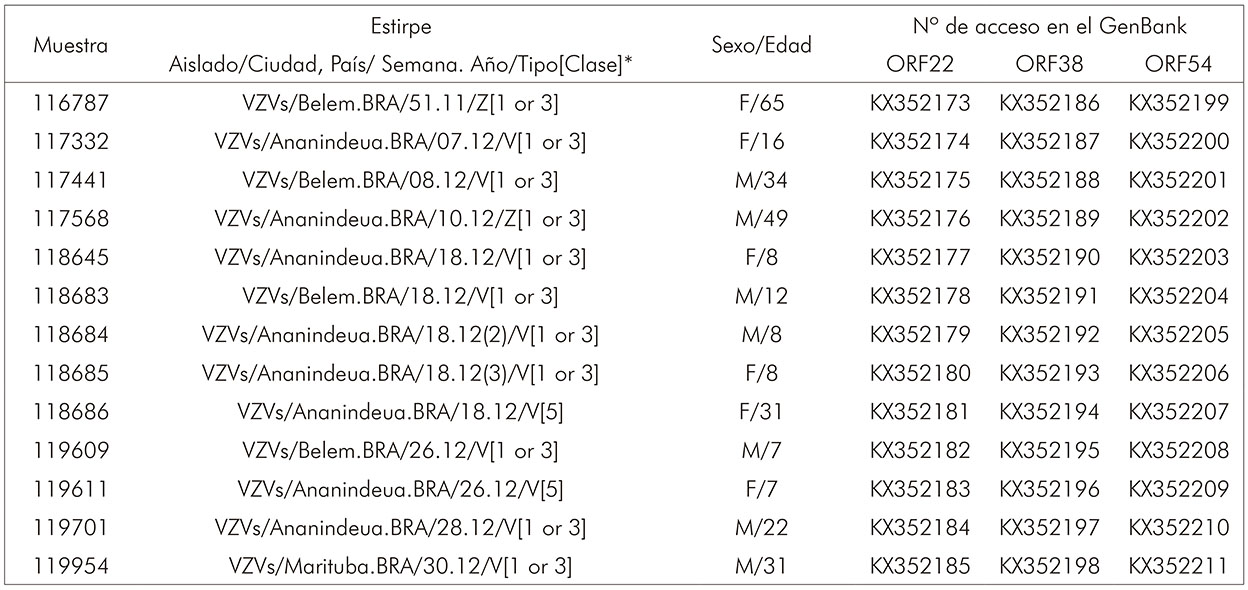
* Descripción de la estirpe: espécimen (representado por la sigla del virus "VZV" y la letra "s" sigla de la palabra specimen, ambas descritas en inglés), lugar de origen de la muestra (ciudad y país), período de la recolección (semana y año de la recolección), tipo (muestra de origen de casos de varicela "V" o zóster "Z") y la clase a que pertenece la estirpe.
Cuadro 3 - Descripción de las estirpes y números de acceso en el GenBank
DISCUSIÓN
Este es un estudio inédito de investigación genotípica del VVZ en la Región Norte de Brasil, adoptando SNP para analizar las diferencias entre las clases 1 o 3 y clase 52,13,15. La preferencia por ese método se basó en el hecho de que las técnicas de genotipado generalmente utilizadas en el laboratorio deben ser de ejecución simple y capaces de alcanzar resultados satisfactorios en muestras con cantidad limitada de ADN, como, por ejemplo, a partir de vesículas y costras.
Algunos estudios muestran que extracción y purificación de ácido nucleico no siempre son necesarias para realizar la PCR36,37. La amplificación del material se vuelve rápida, sensible y con un costo efectivo menor, principalmente cuando son analizadas muchas muestras34. Las muestras de este estudio fueron sumergidas en solución de agua destilada y almacenadas en congelador a -70º C por varios meses sin deterioro en su calidad, aún después de varios ciclos de congelamiento y descongelamiento.
La PCR de material directo de vesículas es sensible y específica para la detección del VVZ, y especímenes de saliva pueden funcionar como una herramienta de soporte para el diagnóstico en determinados casos en que, luego de la extracción con kit comercial, la PCR no funciona por baja cantidad de ADN viral38.
En este estudio, la PCR se realizó en todas las muestras de hisopado nasal, oral y de vesículas. De 26 muestras recolectadas de vesículas y procesadas en agua destilada sometidas a PCR, 13 (50%) fueron amplificadas. Las muestras se recolectaron del primer hasta el 12º día después del surgimiento de las vesículas en la piel, siendo que las muestras amplificadas se recolectaron entre el primero y hasta el noveno día luego del inicio de los síntomas. Lo ideal es que la recolección suceda hasta el séptimo día, cuando las vesículas todavía están productivas38,39,40.
Chun et al39, en estudio con niños que presentaron varicela postvacunal, recolectaron muestras de vesículas en hisopo, las almacenaron en frasco estéril y posteriormente realizaron extracción y purificación del ADN del VVZ con kit comercial, con una positividad de 58%. Esos resultados fueron superiores a los encontrados en otros dos estudios, que utilizaban la misma metodología de extracción y purificación, con presencia de ADN del VVZ en 46%40 y 35%41, respectivamente.
Resultados semejantes se encontraron en estudio realizado en Suiza en el cual el ADN del VVZ estaba presente en 21 (47%) de 45 muestras de suero recolectadas hasta los primeros ocho días de la fase aguda de la enfermedad42.
El análisis de las secuencias de las ORFs 22, 38 y 54 asoció la semejanza de las estirpes encontradas en esta investigación con las clasificadas dentro de las clases 1 o 3 y clase 5. Del total de 13 estirpes aisladas, apenas dos presentaron un genotipo clase 5, las demás fueron clasificadas en clase 1 o 3. Varios estudios demostraron una distribución geográfica distinta de los principales genotipos del VVZ cuando compararon las regiones de clima templado con las de clima tropical19,22,26,28,43,44,45. El dominio regional de genotipos específicos podría haber sido establecido por el clima y/u otros factores, como interacciones virus-huésped o introducción de estirpes en una población inocua al VVZ, por medio de la inmigración o viajes. Estrategias modernas de genotipado pueden ayudar a resolver esos temas. Las regiones en las que varios genotipos VVZ predominan, y esos datos, combinados con los patrones migratorios históricos humanos, contribuyen para identificar los probables puntos de origen para esos genotipos19. La presencia de dos tipos diferentes de genotipos en este estudio puede estar asociada a uno o más de esos factores.
En Argentina, un estudio utilizando la misma técnica de análisis de la ORF22 en muestras de hisopado de vesículas encontró solamente estirpes de genotipo E (clase 1 o 3) en muestras de 13 niños con varicela43.
Los resultados de este estudio, realizado en la región metropolitana de Belém, también mostraron un predominio de la clase 1 o 3, con 11 muestras de VVZ, y difieren, por lo tanto, del estudio en el cual, de diez muestras de VVZ recolectadas en Brasil, ocho estirpes fueron clasificadas como clase 5 y solamente dos como pertenecientes a la clase 1 o 32,13.
El genoma del VVZ es muy estable y la variación en la secuencia del ADN observada ocurre, generalmente, como mutaciones puntuales individuales. Esa justificativa es plausible e indica que un pequeño número de variantes favorecidas ocurre independientemente entre las estirpes de tipo salvaje, en más de una localización geográfica. La literatura destaca que no se puede descartar la posibilidad de que el VVZ de clase 5 haya surgido, primero, en las regiones tropicales, cuando sufrieron mutaciones después que ocurrieron las migraciones humanas para el clima templado. Estirpes clases 1, 2 y 3 pueden haber surgido independientemente de la 5 como resultado del aislado de las islas japonesas y las exploraciones y colonización de los europeos en diversos continentes, respectivamente2,13.
El análisis de las estirpes circulantes en Brasil permitiría, por ejemplo, la asociación de cuadros clínicos graves a la infección por determinada estirpe. Además, estudios comparando las estirpes vacunales con estirpes VVZ circulantes en el País posibilitarían una evaluación más detallada en relación al aclaramiento de síntomas postvacunales, pues permitiría evaluar si una infección está siendo causada por el virus vacunal o si el individuo adquirió, incidentalmente, el virus salvaje próximo al período de vacunación, asociando el aparecimiento de vesículas a la vacuna.
En India, investigadores observaron, en evaluaciones clínicas, que estudiantes con varicela presentaban vesículas largas con tamaño mayor a 8 mm en diámetro, similar a lo que ocurre con pacientes adultos del sexo masculino infectados por el genotipo clase 5 del VVZ36. Dos regiones del VVZ que han sido utilizadas para investigar las transmisiones hospitalarias son la R1 en la ORF11 y OriS2,37,38. Sin embargo, los parámetros precisos que informarán su utilización para la investigación de brotes todavía no están establecidos. Los genotipos más asociados con hospitalizaciones de pacientes con varicela continúan desconocidos, y la virulencia de un genotipo puede estar influenciada más por el nivel de respuesta del huésped y su inmunidad que por un genotipo específico46,47.
Es importante la vigilancia con relación a los tipos de genotipos del VVZ, especialmente para monitoreo de eventos adversos futuros causados por la varicela, y el análisis de polimorfismos en el genoma dl virus en momentos diferentes permitiría estimar su tasa evolutiva48.
En este estudio fue posible la evaluación del genotipo de tan sólo dos muestras de pacientes con herpes-zóster. El análisis genotípico en muestras de pacientes con herpes-zóster podría proporcionar una evaluación más detallada de cuanto tiempo determinada estirpe circula en el País (tasa evolutiva) además de evaluar las estirpes más asociadas a la reactivación viral y surgimiento del herpes-zóster años después de la infección primaria por el VVZ.
La infección primaria con VVZ ocurre, generalmente, durante la infancia, alrededor de los 10 años de edad49. El herpes-zóster se desarrolla a partir de la reactivación del VVZ y la estirpe aislada en casos de zóster es la misma encontrada en infecciones por este virus adquiridas en la infancia. Por lo tanto, una estirpe de VVZ aislado de una paciente de 70 años de edad con herpes-zóster, en Corea del Sur, probablemente circuló cerca de 60 años atrás, en la década de 194050. En este estudio, la muestra de VVZ a partir de un caso de herpes-zóster en adulto de 65 años de edad, oriundo de Belém, reveló estirpe del genotipo clase 1 o 3, sugiriendo que la misma ya circula por, mínimo, cerca de cinco décadas en Brasil. Otra muestra de paciente con herpes-zóster también fue identificada como clase 1 o 3, este con edad de 45 años, quien siempre vivió en Ananindeua, pero serán necesarios estudios de análisis de tasa evolutiva para estimar mejor el período en que as clases 1 o 3 fueron introducidas en la región.
El análisis de las ORFs 21 y 50 se hace necesario para definir las estirpes de genotipo europeo (E1 y E2) en clase 1 o 32,19. Otros estudios de genotipado son necesarios para acompañar el posible impacto de la vacunación en la circulación de genotipos de VVZ en Brasil, además de posibles asociaciones con diferentes cuadros clínicos y la posibilidad de análisis de la tasa evolutiva del virus.
Este es un estudio inédito en Brasil, ya que es la primera vez que el VVZ tiene su genotipo definido por pruebas realizadas en una institución de investigación nacional.
CONCLUSIÓN
Los genotipos del VVZ se clasificaron en clases 1 o 3 y clase 5. El genotipo clase 1 o 3 se encontró en los Municipios de Ananindeua, Belém y Marituba, y el clase 5 fue encontrado apenas en Ananindeua. La PCR fue sensible en 13 muestras de material directo de vesículas, mientras que en muestras de saliva no se obtuvo éxito. La investigación de las ORFs 21 y 50 es importante para definir las clases 1 y 3 en las muestras analizadas. Otros estudios son necesarios para identificar los genotipos del VVZ circulantes en Brasil.
REFERENCIAS
1 Davison AJ, Scott JE. The complete DNA sequence of varicella-zoster virus. J Gen Virol. 1986 Sep;67(9):1759-816. Doi: 10.1099/0022-1317-67-9-1759 [Link] [ Links ]
2 Breuer J, Grose C, Norberg P, Tipples G, Schmid DS. A proposal for a common nomenclature for viral clades that form the species varicella-zoster virus: summary of VZV Nomenclature Meeting 2008, Barts and the London School of Medicine and Dentistry, 24-25 July 2008. J Gen Virol. 2010 Apr;91(4):821-8. Doi: 10.1099/vir.0.017814-0 [Link] [ Links ]
3 Hope-Simpson RE. The nature of herpes zoster: along term study and a new hypothesis. Proc R Soc Med. 1965 Jan;58(1):9-20. [Link] [ Links ]
4 Hambleton S, Gershon AA. Preventing varicella-zoster disease. Clin Microbiol Rev. 2005 Jan;18(1):70-80. Doi: 10.1128/CMR.18.1.70-80.2005 [Link] [ Links ]
5 Hondo R, Yogo Y, Yoshida M, Fujima A, Itoh S. Distribution of varicella-zoster virus strains carrying a PstI-site-less mutation in Japan and DNA change responsible for the mutation. Jpn J Exp Med. 1989 Dec;59(6):233-7. [ Links ]
6 LaRussa P, Lungu O, Hardy I, Gershon A, Steinberg SP, Silverstein S. Restriction fragment length polymorphism of polymerase chain reaction products from vaccine and wild-type varicella-zoster virus isolates. J Virol. 1992 Feb;66(2):1016-20. [Link] [ Links ]
7 LaRussa P, Steinberg S, Arvin A, Dwyer D, Burgess M, Menegus M, et al. Polymerase chain reaction and restriction fragment length polymorphism analysis of varicella-zoster virus isolates from the United States and other parts of the world. J Infect Dis. 1998 Nov;178 Suppl 1:64-6. Doi: 10.1086/514267 [Link] [ Links ]
8 Loparev VN, Argaw T, Krause PR, Takayama M, Schmd DS. Improved identification and differentiation of varicella-zoster virus (VZV) wild-type strains and an attenuated varicella vaccine strain using a VZV open reading frame 62-based PCR. J Clin Microbiol. 2000 Sep;38(9): 3156-60. [Link] [ Links ]
9 Loparev VN, McCaustland K, Holloway BP, Krause PR, Takayama M, Schmid DS. Rapid genotyping of varicella-zoster virus vaccine and wild-type strains with fluorophore-labeled hybridization probes. J Clin Microbiol. 2000 Dec;38(12):4315-9. [Link] [ Links ]
10 Collins FS. Sequencing the human genome. Hosp Pract. 1997 Jan;32(1):35-43. Doi: 10.1080/21548331.1997.11443403 [Link] [ Links ]
11 Barret-Muir W, Hawrami K, Clarke J, Breuer J. Investigation of varicella-zoster virus variation by heteroduplex mobility assay. In: Gershon AA, Arvin AM, Calisher CH, editors. Immunity to and prevention of herpes Zoster. New York: Springer-Verlag Wien; 2011. p. 17-25. [ Links ]
12 Faga B, Maury W, Bruckner DA, Grose C. Identification and mapping of single nucleotide polymorphism in the varicella-zoster virus genome. Virology. 2001 Feb;280(1):1-6. Doi: 10.1006/viro.2000.0775 [Link] [ Links ]
13 Loparev VN, Gonzalez A, Carnes MD, Tipples G, Fickenscher H, Torfason EG, et al. Three major genotypes of varicella-zoster virus: geographical clustering and strategies for genotyping. J Virol. 2004 Aug;78(15):8349-58. [Link] [ Links ]
14 Muir WB, Nichols R, Breuer J. Phylogenetic analysis of varicella-zoster virus: evidence of intercontinental spread of genotypes and recombination. J Virol. 2002 Feb;76(4):1971-9. Doi: 10.1128/JVI.76.4.1971-1979.2002 [Link] [ Links ]
15 Kenyon TK, Lynch J, Hay J, Ruyechan W, Grose C. Varicella-zoster virus ORF47 protein serine kinase: characterization of a cloned, biologically active phosphotransferase and two viral substrates, ORF62 and ORF63. J Virol. 2001 Sep;75(18):8854-8. Doi: 10.1128/JVI.75.18.8854-8858.2001 [Link] [ Links ]
16 Peters GA, Tyler SD, Grose C, Severini A, Gray MJ, Upton C, et al. A full-genome phylogenetic analysis of Varicella-zoster virus reveals a novel origin of replication-based genotyping scheme and evidence recombination between major circulating clades. J Virol. 2006 Oct;80(19):9850-60. Doi: 10.1128/JVI.00715-06 [Link] [ Links ]
17 Koskiniemi M, Lappalainen M, Schmid DS, Rubtcova E, Loparev VN. Genotypic analysis of varicella-zoster virus and its seroprevalence in Finland. Clin Vaccine Immunol. 2007 Sep;14(9):1057-61. Doi: 10.1128/CVI.00348-06 [Link] [ Links ]
18 Loparev VN, Rubtcova EN, Bostik V, Govil D, Birch CJ, Druce JD, et al. Identification of five major and two minor genotypes of varicella-zoster virus strains: a practical two-amplicon approach used to genotype clinical isolates in Australia and New Zealand. J Virol. 2007 Dec;81(23):12758-65. Doi: 10.1128/JVI.01145-07 [Link] [ Links ]
19 Loparev V, Martro E, Rubtcova E, Rodrigo C, Piette JC, Caumes E, et al. Toward universal varicella-zoster virus (VZV) genotyping: diversity of VZV strains from France and Spain. J Clin Microbiol. 2007 Feb;45(2):559-63. Doi: 10.1128/JCM.01738-06 [Link] [ Links ]
20 Zell R, Taudien S, Pfaff F, Wutzler P, Platzer M, Sauerbrei A. Sequencing of 21 varicella-zoster virus genomes reveals two novel genotypes and evidence of recombination. J Virol. 2012 Feb;86(3): 1608-22. Doi: 10.1128/JVI.06233-11 [Link] [ Links ]
21 Depledge DP, Gray ER, Kundu S, Cooray S, Poulsen A, Aaby P, et al. Evolution of cocirculating varicella-zoster virus genotypes during a chickenpox outbreak in Guinea-Bissau. J Virol. 2014 Dec;88(24):13936-46. Doi: 10.1128/JVI.02337-14 [Link] [ Links ]
22 Jiang L, Gan L, Chen J, Wang M. Genetic analysis of clinical VZV isolates collected in China reveals a more homologous profile. Biomed Res Int. 2013;(681234):1-6. Doi: 10.1155/2013/681234 [Link] [ Links ]
23 Sauerbrei A, Rubtcova E, Wutzler P, Schmid DS, Loparev VN. Genetic profile of an Oka varicella vaccine virus variant isolated from an infant with zoster. J Clin Microbiol. 2004 Dec;42(12):5604-8. Doi: 10.1128/JCM.42.12.5604-5608.2004 [Link] [ Links ]
24 Sergeev N, Rubtcova E, Chizikov V, Schmid DS, Loparev VN. New mosaic subgenotype of varicella-zoster virus in the USA: VZV detection and genotyping by oligonucleotide-microarray. J Virol Methods. 2006 Sep;136(1-2):8-16. Doi: 10.1016/j.jviromet.2006.03.021 [Link] [ Links ]
25 Norberg P, Liljeqvist JA, Bergström T, Sammons S, Schmid DS, Loparev VN. Complete genome phylogenetic approach to varicella-zoster virus evolution: genetic divergence and evidence for recombination. J Virol. 2006 Oct;80(19): 9569-76. Doi: 10.1128/JVI.00835-06 [Link] [ Links ]
26 Quinlivan M, Hawrami K, Barret-Muir W, Aaby P, Arvin A, Chow VT, et al. The molecular epidemiology of varicella-zoster virus: evidence for geographic segregation. J Infect Dis. 2002 Oct;186(7):888-94. Doi: 10.1086/344228 [Link] [ Links ]
27 Sengupta N, Taha Y, Scott FT, Leedham-Green ME, Quinlivan M, Breuer J. Varicella-zoster virus genotypes in East London: a prospective study in patients with herpes zoster. J Infect Dis. 2007;196(7):1014-20. Doi: 10.1086/521365 [Link] [ Links ]
28 Barrett-Muir W, Scott FT, Aaby P, John J, Matondo P, Chaudhry QL, et al. Genetic variation of varicella-zoster virus: evidence for geographical separation of strains. J Med Virol. 2003;70(Suppl 1):S42-7. Doi: 10.1002/jmv.10319 [Link] [ Links ]
29 Carvalho ES, Martins RM. Varicela: aspectos clínicos e prevenção. J Pediatr. 1999 jan-fev;75(supl 1): S126-34. Doi: 10.2223/JPED.379 [Link] [ Links ]
30 Kupek E, Tritany EF. Impacto da vacinação contra varicela na redução da incidência da doença em crianças e adolescentes de Florianópolis (SC). J Pediatr. 2009 ago;85(4):365-8. Doi: 10.2223/JPED.1909 [Link] [ Links ]
31 Secretaria de Estado da Saúde (São Paulo). Centro de Vigilância Epidemiológica "Prof. Alexandre Vranjac". Divisão de Doenças de Transmissão Respiratória e Divisão de Imunização. Instruções sobre a utilização de imunobiológicos para controle de varicela em creches [Internet]. São Paulo: CVE; 2003 [citado 2005 set 30]. Disponível em: Disponível em: http://www.cve.saude.sp.gov.br/htm/resp/pdf/inst_varicela.pdf . [ Links ]
32 Hirose M, Gilio AE, Ferronato AE, Ragazzi SLB. The impact of varicella vaccination on varicella-related hospitalization rates: global data review. Rev Paul Pediatr. 2016;34(3):359-66. Doi: 10.1016/j.rpped.2015.12.006 [Link] [ Links ]
33 Dinelli MIS, Ono E, Viana PO, Spina FG, Weckx LY, Santos AMN, et al. Response to immunization in children born to renal transplant recipients using immunosuppressive drugs during gestation. Vaccine. 2016 Jan;34(4):404-7. Doi: 10.1016/j.vaccine.2015.12.017 [Link] [ Links ]
34 Sakai K, Wakasugi S, Muchemwa FC, Ihn H. Quick detection of herpes viruses from skin vesicles and exudates without nucleic acid extraction using multiplex PCR. Biosci Trends. 2008 Aug;2(4):164-8. [Link] [ Links ]
35 Saitou N, Nei M. The neighbor-joining method: a new method for reconstructing phylogenetic trees. Mol Biol Evol. 1987 Jul;4(4):406-25. [Link] [ Links ]
36 Pandori MW, Lei J, Wong EH, Klausner J, Liska S. Real-Time PCR for detection of herpes simplex virus without nucleic acid extraction. BMC Infect Dis. 2006;6(104):1-9. [ Links ]
37 Bezold G, Volkenandt M, Gottlöber P, Peter RU. Detection of herpes simplex virus and varicella-zoster virus in clinical swabs: frequent inhibition of PCR as determined by internal controls. Mol Diagn. 2000 Dec;5(4):279-84. Doi: 10.1054/modi.2000.19215 [Link] [ Links ]
38 Leung J, Harpaz R, Baughman AL, Heath K, Loparev V, Vázquez M, et al. Evaluation of laboratory methods for diagnosis of varicella. Clin Infect Dis. 2010 Jul;51(1):23-32. Doi: 10.1086/653113 [Link] [ Links ]
39 Chun C, Weinmann S, Riedlinger K, Mullooly JP, Houston H, Schmid DS, et al. Laboratory characteristics of suspected herpes zoster in vaccinated children. Pediatr Infect Dis J. 2011 Aug;30(8):719-21. [ Links ]
40 Jumaan AO, Seward J. The effectiveness of varicella vaccine [letter]. N Engl J Med. 2001 Aug;345(6):464-5. Doi: 10.1056/NEJM200108093450613 [Link] [ Links ]
41 Watson B, Perella D, Heath K. Laboratory confirmation of suspected breakthrough varicella infections (1927). In: Program and Abstracts of the Pediatric Academic Societies Annual Meeting. 2004 May 1-4; San Francisco, CA. Woodlands (TX): Pediatric Academic Societies; 2004. [ Links ]
42 Dobec M, Bossart W, Kaeppeli F, Mueller-Schoop J. Serology and serum DNA detection in shingles. Swiss Med Wkly. 2008 Jan;138(3-4):47-51. [Link] [ Links ]
43 Dayan GH, Panero MS, Debbag R, Urquiza A, Molina M, Prieto S, et al. Varicella seroprevalence and molecular epidemiology of varicella-zoster virus in Argentina. J Clin Microbiol. 2004 Dec;42(12):5698-704. Doi: 10.1128/JCM.42.12.5698-5704.2004 [Link] [ Links ]
44 Lolekha S, Tanthiphabha W, Sornchai P, Kosuwan P, Sutra S, Warachit B, et al. Effect of climatic factors and population density on varicella zoster virus epidemiology within a tropical country. Am J Trop Med Hyg. 2001 Mar-Apr;64(3-4):131-6. [Link] [ Links ]
45 Wagenaar TR, Chow VTK, Buranathai C, Thawatsupha P, Grose C. The out of Africa model of varicella-zoster virus evolution: single nucleotide polymorphisms and private alleles distinguish Asian clades from European/North American clades. Vaccine. 2003 Mar;21(11-12):1072-81. Doi: 10.1016/S0264-410X(02)00559-5 [Link] [ Links ]
46 Molyneaux PJ, Parker S, Khan IH, Millar CGM, Breuer J. Use of genomic analysis of varicella-zoster virus to investigate suspected varicella-zoster transmission within a renal unit. J Clin Virol. 2006 May;36(1):76-8. Doi: 10.1016/j.jcv.2005.12.012 [Link] [ Links ]
47 Tang JW, Eames I, Li Y, Taha YA, Wilson P, Bellingan G, et al. Door-opening motion can potentially lead to transient breakdown in negative-pressure isolation conditions: the importance of vorticity and buoyancy airflows. J Hosp Infect. 2005 Dec;61(4):283-6. Doi: 10.1016/j.jhin.2005.05.017 [Link] [ Links ]
48 Kolesnik M, Bonnekoh B, Tammer I, Gollnick H, Sauerbrei A. Varicella outbreak in an Indian couple living in Germany caused by VZV clade VI acquired during a trip to the Netherlands. Case Rep Med. 2012;2012(838241):1-3. Doi: 10.1155/2012/838241 [Link] [ Links ]
49 Gnann Jr JW, Whitley RJ. Herpes zoster. N Engl J Med. 2002 Aug;347(5):340-6. Doi: 10.1056/NEJMcp013211 [Link] [ Links ]
50 Kim KH, Choi YJ, Song KH, Park WB, Jeon JH, Park SW, et al. Genotype of varicella-zoster virus isolates in South Korea. J Clin Microbiol. 2011 May;49(5):1913-6. Doi: 10.1128/JCM.02356-10 [Link] [ Links ]
Cómo citar este artículo / How to cite this article: Costa MRM, Monteiro TAF, Linhares AC, Costa IB, Kaiano JHL, Oliveira DS, et al. Virus de la varicela zóster: identificación de los genotipos en casos de varicela y herpes zóster en los Municipios de Ananindeua, Belém y Marituba, Estado de Pará, Brasil. Rev Pan-Amaz Saude. 2016 jul-sept;7(3):1-11. Doi: http://dx.doi.org/10.5123/S2176-62232016000300004
Recibido: 10 de Noviembre de 2015; Aprobado: 06 de Julio de 2016










 texto em
texto em