INTRODUÇÃO
O herpesvírus humano-3 (HHV-3), também conhecido como vírus da varicela-zoster (VVZ) da subfamília Alphaherpesvirinae, gênero Varicellovirus, apresenta como principal característica a capacidade de estabelecer latência. A partícula intacta do VVZ mede de 180-200 nm de diâmetro, com genoma linear de DNA dupla fita de 125 kb, que está contido em um cerne de 75 nm dentro de um nucleocapsídio. Assim como outros herpesvírus, o VVZ tem regiões únicas longas e curtas, uma região de repetição terminal (TR) e outra invertida (IR), dentro das quais se encontram os genes duplicados, além das regiões internas de repetições R1 a R51,2.
O VVZ pode causar varicela (catapora) durante a infecção primária, estabelecendo posteriormente uma infecção latente. Em diversas situações em que ocorre um declínio da imunidade celular específica contra o VVZ pode ocorrer reativação da replicação viral, com possibilidade de manifestação clínica por meio do herpes-zoster3. A vacina com o VVZ atenuado estirpe Oka/Merck, licenciada nos Estados Unidos da América (EUA), quando aplicada em duas doses com diferença de três meses, apresenta 85% de eficácia na prevenção contra a varicela e 95% contra doença grave que requer terapia antiviral, sendo bastante segura e bem tolerada por crianças saudáveis. Eventos adversos, tais como herpes-zoster pós-vacinal, são raros e ocorrem na maioria dos casos em imunocomprometidos4.
Para distinguir a estirpe Oka (utilizada em vacinas) do tipo "selvagem" de VVZ, diversos estudos combinaram as técnicas de polimorfismo no comprimento de fragmentos de restrição (RFLP) e reação em cadeia da polimerase (PCR) para produzir métodos laboratoriais com relativo grau de sucesso para uso na rotina. Esses métodos evidenciam a divergência genética nas ORF38 e ORF54 do genoma viral, as quais têm como produto, respectivamente, as seguintes glicoproteínas (gp): gp40, uma proteína tegumentar que interage com os microtúbulos da célula na formação da partícula viral e gp55, uma proteína tegumentar estrutural denominada de proteína portal do capsídeo5,6,7,8,9.
Por meio de técnicas de PCR e RFLP, duas regiões clivadas por enzimas de restrição correspondentes a polimorfismos em polimorfismo de nucleotídeo único (single nucleotide polymorphisms - SNP) do VVZ têm funcionado como poderosa referência pontual para caracterização epidemiológica e geográfica das variações das estirpes10. SNPs são regiões que contêm variações únicas de pares de bases10. No caso do VVZ, a partir de 2000, seus SNPs foram caracterizados a partir de metodologias de genotipagem publicadas por dois grupos, um norte-americano e outro inglês11,12,13,14.
As duas metodologias de SNP baseadas em genotipagem diferem em suas abordagens. O método de SNP disperso, desenvolvido pelo Grupo de Breuer, no Reino Unido, baseia-se em ensaios de mobilidade heteroduplex para identificar 92 polimorfismos em 37 ORFs distribuídos uniformemente pelo genoma do VVZ. Esse grupo de pesquisa identificou três genótipos filogeneticamente distintos circulando no Reino Unido e um quarto no Japão2,11,14.
O grupo norte-americano (Universidade de Iowa, Iowa, EUA), liderada por Grose, sequenciou cinco genes de glicoproteínas, dentre esses a ORF que codifica a maior proteína de transativação imediata (IE62)15. Sessenta e um polimorfismos, 21 nas glicoproteínas e 40 na ORF62, foram identificados12. A análise filogenética dessas regiões identificou quatro classes16.
Um terceiro grupo de pesquisa, liderado por Schmid no Centers for Disease Control and Prevention (CDC), localizou um fragmento de 447 pb na ORF22, região denominada de "up stream" C-terminal, o qual foi suficientemente polimórfico para permitir a identificação de quatro subtipos virais13. Loparev et al13 analisaram variações genotípicas utilizando um fragmento de 447 pb da região ORF22 (especificamente as posições nucleotídicas 37902, 38055, 38081, 38177 e 38229), que tem como produto a gp24, uma grande proteína estrutural que faz parte do tegumento viral, com localização subcelular no virion1,13. A diversidade nucleotídica desta sequência foi usada para definir o genótipo de 326 isolados de VVZ de amostras de cinco continentes13.
O uso das endonucleases de restrição PstI e Bgll, permitiu identificar os sítios de clivagem para estas enzimas na ORF38 e ORF54, respectivamente. Diversas estirpes isoladas nos EUA contêm um sítio de restrição da enzima PstI na região ORF38, onde ocorre uma troca do nucleotídeo guanina (G) por adenina (A) na posição 69349 que a estirpe Oka vacinal não possui5. Além disso, uma troca do nucleotídeo citosina (C) por timina (T) na posição 95241 resulta na eliminação do sítio de restrição da enzima BglI, localizada na ORF54, a qual é encontrada em todas as estirpes japonesas e que está ausente em muitos isolados dos EUA6,7,8,9. Dessa maneira, as estirpes japonesas podem apresentar os genótipos PstI+ BglI+ ou PstI- BglI+, enquanto muitos VVZ isolados nos EUA são PstI+ BglI-6,7,8,9.
Em 2007, a análise dessas três ORFs permitiu a classificação dos genótipos do VVZ por distribuição geográfica em: europeu (E), japonês (J) e mosaico (M)17.
Ao todo, esses três grupos de pesquisa identificaram cinco subtipos virais distintos, embora nenhum grupo tenha identificado todos os cinco. De acordo com a conferência VZV Nomenclature Meeting 20082, realizada em 25 de julho de 2008 em Whitechapel, Londres, Reino Unido, as cepas de VVZ detectadas e caracterizadas por sequenciamento dos SNPs seriam agrupadas em classes de 1 a 5, em substituição aos genótipos preconizados na classificação do CDC. As diferentes nomenclaturas utilizadas por cada grupo são apresentadas no quadro 1.
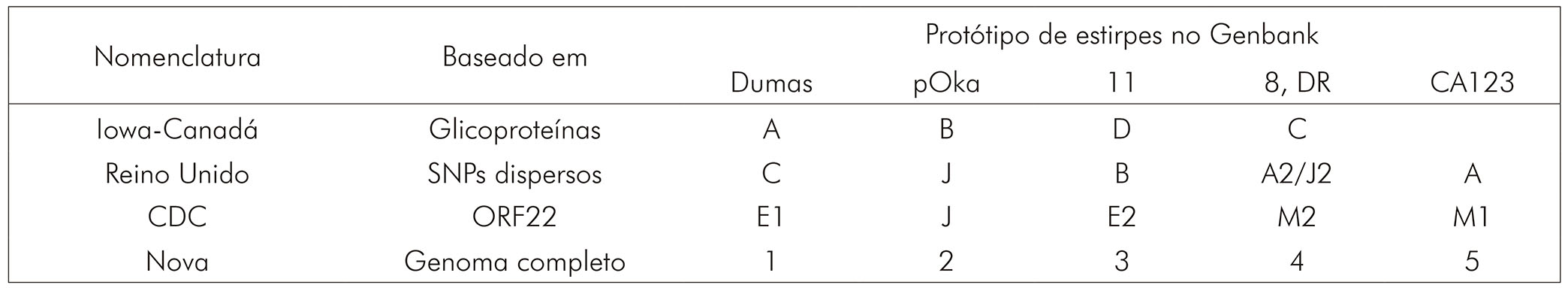
Fonte: Adaptado de Breuer et al2, 2010.
Quadro 1 - Nomenclatura genotípica do VVZ utilizada atualmente
Loparev et al18 identificaram SNPs que discriminam os genótipos E1 e E2 nas ORF21, ORF22 [região 2], e ORF50. Análise de SNP em qualquer destas regiões é suficiente para discriminar cepas E1 de outras europeias. Duas posições adicionais em ORF38 (69349) e ORF54 (95241), originalmente usadas para confirmar os eventos adversos da vacina Oka, também fornecem informações para genotipagem. Esses dois marcadores não são úteis para vacina Oka especificamente, mas servem para distinguir algumas cepas J, M1, M2 e M419 de outros genótipos. Um subconjunto de isolados de genótipo J (por exemplo, pOka, que foi a fonte para a vacina de vírus Oka) contém um sítio de restrição PstI na ORF38 e ORF54 incluindo a sequência de um local de restrição Bgll comum a genótipos J, M1, M2 e M42,13.
O genótipo E foi identificado em cepas obtidas de pacientes da Alemanha, Islândia, República Tcheca, Polônia, Rússia europeia, Bielorrússia, Ucrânia, Lituânia, Letônia, Moldávia e Estônia13,20. Esse genótipo também foi o mais prevalente na Ásia, Rússia, Cazaquistão, leste da Austrália, Canadá, Estados Unidos, Jordânia, Chile e Nicarágua, mostrando-se altamente cosmopolita. Com base na análise dos isolados obtidos a partir de vários locais geográficos distintos, o genótipo E mostrou um grupo de bases conservadoras no perfil dos SNPs da ORF22 e todas as cepas E também apresentaram o padrão de marcador PstI Bgll. Uma observação surpreendente foi a descoberta do domínio geográfico de cepas do genótipo M em áreas tropicais e subtropicais, como a África (Congo, República do Tchad, Marrocos e Guiné-Bissau) e na Ásia (Índia, Bangladesh, Nepal e China)13,21.
Além das cepas J comuns no Japão, Austrália e alguns países do leste asiático, os padrões Bgl + também são comumente isolados em climas tropicais (por exemplo, na África equatorial, Índia, Bangladesh, no sul da China, América Central e norte da Austrália). Em contraste, vírus Bgl - são comuns em climas temperados. Dois isolados Pst - Bgl - também foram observados na Austrália13,18,19,22. Assim, para dominância geográfica, foram observados os genótipos E1 e E2 (Europa), M1 (África) e J (Japão). O predomínio de cepas genótipo E na América do Norte e Austrália provavelmente foi estabelecido durante a colonização europeia nos séculos XVI e XVII. Mudanças relativamente recentes na imigração e/ou padrões de viagens internacionais para certas regiões geográficas (principalmente Europa, Austrália e Estados Unidos) têm ocasionado a cocirculação de uma variedade de genótipos de VVZ13,17,19,20,23,24,25,26,27.
Um genótipo proposto como recombinante do VVZ de amostras do Brasil foi classificado como uma estirpe do genótipo M19. De dez isolados obtidos de forma independente no Brasil, oito estirpes tipo M e dois do genótipo E tinham padrões de SNP semelhantes aos descritos por Barrett-Muir et al13,28.
No Brasil, a maior parte dos estudos envolvendo varicela e herpes-zoster considera apenas os aspectos sintomatológicos29,30,31,32,33. Assim sendo, este estudo teve por objetivo permitir o conhecimento de estirpes circulantes do VVZ na região metropolitana de Belém, Estado do Pará, Brasil e disponibilizar informações importantes de identificação e distribuição dos genótipos do VVZ, nas diversas faixas etárias investigadas e/ou associação desses à determinada patologia.
MATERIAIS E MÉTODOS
ÁREA DE ESTUDO
As amostras para este estudo foram obtidas nos Municípios de Ananindeua, Belém e Marituba, localizados na região metropolitana de Belém, Estado do Pará.
POPULAÇÃO DO ESTUDO
Estudo do tipo transversal em que o critério clínico para inclusão das amostras foi a suspeita clínica e/ou aparecimento dos sintomas de varicela ou herpes-zoster. Durante o período de novembro de 2008 a março de 2012, foram coletadas 93 amostras de swab nasal/oral (49 de indivíduos do gênero masculino e 44 do feminino). Desses 93 indivíduos, 26 (13 de indivíduos do gênero masculino e 13 do feminino) apresentavam vesículas rompidas, das quais foram coletados swab. Todas as amostras, oriundas de pacientes procedentes das redes pública e privada de saúde, foram coletadas no Instituto Evandro Chagas (IEC), localizado em Ananindeua, Estado do Pará.
IDENTIFICAÇÃO, PROCESSAMENTO E ARMAZENAMENTO DAS AMOSTRAS
A coleta das amostras foi realizada em 93 indivíduos (85 casos de varicela e oito de herpes-zoster), sendo 93 amostras de swab nasal/oral e 26 amostras (22 casos de varicela e quatro de herpes-zoster) de secreção de vesículas rompidas em vários sítios anatômicos. As amostras foram identificadas e coletadas no dia do atendimento e encaminhadas para a Seção de Virologia do IEC. O período de coleta das amostras foi definido entre o primeiro até 12º dia após o início dos sintomas.
Na etapa de processamento, sem extração de ácidos nucleicos e utilização direta do material clínico para etapa de amplificação, as amostras de swab nasal, oral e vesículas foram mergulhadas, cada uma, em um microtubo contendo 100 µL de água destilada e esterilizada, sendo o swab lavado em 400 µL do mesmo tipo de água, pressionando-o contra a parede do tubo para retirar o máximo de partículas virais e, após esta etapa, a suspensão foi armazenada a -70º C34.
AMPLIFICAÇÃO DO DNA VIRAL POR PCR
As estirpes Dumas e Oka parental foram incluídas como referência padrão dos genótipos E (classes 1 ou 3) e J (classe 2), respectivamente. As estirpes foram classificadas nas classes de 1, 2 ou 3 quando exibiram os SNPs de um dos dois genótipos. Algumas estirpes apresentaram variabilidades nessas sequências e foram classificadas dentro do grupo de genótipo M (classes 4 ou 5)2,13.
Após o processamento das amostras de swab nasal, oral e/ou de vesículas, o material foi utilizado diretamente na PCR. Amostras de células diploides humanas HEp-2 (carcinoma epidermoide humano) foram usadas como controle negativo, enquanto que uma amostra comercial da vacina Varicela Biken® (Sanofi Pasteur, Suita, Osaka, Japão), estirpe vOka, foi utilizada como controle positivo para identificação das ORFs 22, 38 e 54.
A reação de amplificação de um fragmento de 447 pb da região ORF22 foi realizada em termociclador Gene Amp PCR System 9600 (Applied Biosystems, Foster City, Califórnia, EUA) com os oligonucleotídeos p22R1f 5'-GGGTTTTGTATGAGCGTTGG-3' e p22R1r 5'-CCCCCGAGGTTCGTAATATC-3'; para amplificação de 350 pb da região ORF38 foram utilizados os oligonucleotídeos PstA 5'-TTGAACAATCACGAACCGTT-3' e PstB 5'-CGGGTGAACCGTATTCTGAG-3'; e para amplificação de 222 pb da ORF54 foram utilizados os oligonucleotídeos Nla 5'-GGAACCCCTGCACCATTAAA-3' e Fok 5'-TCCCTTCATGCCCGTTACAT-3'10.
As mesmas condições de reação de PCR foram adotadas para essas duas regiões com volume final de 50 µL, usando AmpliTaq Gold PCR Master Mix (0,025 U da enzima GoldTaq DNA polimerase, tampão II 1X para PCR, 2,5 mM de MgCl2, 200 µM de cada deoxinucleotídeo trifosfato [Invitrogen, Carlsbad, Califórnia, EUA]), 0,2 µM de cada oligonucleotídeo, e 5 µL da amostra. Cada etapa incluiu um aquecimento inicial de 96º C por 5 min para ativação da enzima, seguida por 40 ciclos de: 94º C por 1 min, 58º C por 2 min e 72º C por 2 min; após essa etapa foi realizada uma extensão final a 72º C por 7 min10.
ELETROFORESE EM GEL DE AGAROSE
A visualização dos produtos da PCR ocorreu por eletroforese horizontal em gel de agarose a 1,8% contendo 6 µL de Sybr Safe (Invitrogen, Carlsbad, Califórnia, EUA) e imerso em tampão tris-borato-EDTA (TBE) 1X. Foram misturados 10 µL da reação de PCR com 1 µL de azul de bromofenol e aplicados no gel para uma eletroforese a 100 V e 400 mA por 30 min. Para análise do padrão das bandas, foi utilizado um marcador de peso molecular de 123 pb (Invitrogen) diluído em tampão TBE 1X10. Os amplicons foram visualizados com o sistema de fotodocumentação Gel Doc 1000 (Bio Rad, Hercules, Califórnia, EUA).
PURIFICAÇÃO DO PRODUTO DA PCR
O produto da PCR foi purificado visando o sequenciamento de nucleotídeos (nt). Para tal, utilizou-se o kit QIAquick PCR purification (QIAGEN, Hilden, Alemanha) seguindo-se o protocolo descrito pelo fabricante.
SEQUENCIAMENTO DE NUCLEOTÍDEOS
Os produtos da PCR foram sequenciados para determinação do genótipo dos VVZ pela identificação de mutação no sítio de clivagem da enzima de restrição PstI, a qual resulta na mudança G69349A (ORF38). A mutação C95241T (ORF54) causa a eliminação do sítio de restrição para a enzima BglI. O produto do SNP da ORF22 foi utilizado para análise de mutações nas posições nucleotídicas 37902, 38055, 38081, 38177 e 3822910.
Na reação de sequenciamento foi realizada uma mistura de 3,2 pmol de cada oligonucleotídeo correspondente e 10 ng do produto como fonte. Foram realizados 25 ciclos de 96º C por 10 s, 50º C por 5 s e 60º C por 4 min. Os produtos resultantes foram purificados com colunas giratórias (Princeton Separations, Adelphia, Nova Jersey, EUA) e resuspendidos em tampão de eluição (Applied Biosystems, Waltham, Massachusetts, EUA)10.
Os polimorfismos nucleotídicos específicos do VVZ foram analisados em sequenciador automático, 3130X/Genetic Analyzer (AB Applied Biosystems/Hitachi, Chiyoda, Tóquio, Japão) empregando o método Big Dye Terminator Cycle Sequencing Ready Reaction Kit (Applied Biosystems, Waltham, Massachusetts, EUA)10.
EDIÇÃO E ALINHAMENTO DAS SEQUÊNCIAS NUCLEOTÍDICAS
As sequências nucleotídicas obtidas neste estudo foram alinhadas e editadas usando o programa BioEdit Sequence Alignment Editor v.7.0.5.2 e comparadas com sequências de outras estirpes do VVZ disponíveis no banco de dados (GenBank - National Center for Biotechnology Information, EUA [www.ncbi.nlm.nih.gov]), usando o aplicativo Basic Local Alignment Search Tool (BLAST). As sequências genômicas de cada ORF do VVZ foram comparadas entre si, bem como com sequências de outros herpesvírus (vírus herpes simples 1 e 2, vírus Epstein-Barr e citomegalovírus) disponíveis no GenBank10.
As estirpes Dumas (X04370), HJ0 (AJ871403), CA 123 (número de acesso no GenBank DQ457052), DR ou Morroco 1 (DQ452050), Var160 ou Mexico_68 (KC112914.1), pOka (AB097933) e vOka (AB097932) foram utilizadas para comparação das substituições nucleotídicas nas amostras analisadas neste estudo10.
ANÁLISE FILOGENÉTICA
Os dendrogramas foram construídos utilizando-se o programa MEGA5.1 (Molecular Evolutionary Genetics Analysis, v5.1.3.308). A análise da distância genética entre as sequências foi calculada pelo algoritmo de neighbor-joining (NJ), pelo método de Kimura 2-parâmetros. O método de bootstrap com 2.000 réplicas foi utilizado para o cálculo da robustez dos dendrogramas35.
ASPECTOS ÉTICOS E DE BIOSSEGURANÇA
O estudo não ocasionou qualquer risco ou prejuízo para os participantes ou seus responsáveis. O projeto envolvido no presente estudo foi avaliado pelo Comitê de Ética em Pesquisa (CEP) do IEC, bem como pela Comissão Nacional de Ética em Pesquisa, quando cabível, sob n° 0028/2009, com parecer de aprovação n° 0004/2009. Quanto aos aspectos de biossegurança, em todos os procedimentos foram utilizados equipamentos de proteção individual, sendo que a manipulação de todo material potencialmente infectante foi realizada em laboratório de segurança NB2, em cabine de segurança biológica classe 2.
RESULTADOS
SNP UTILIZADOS PARA GENOTIPAGEM DO VVZ
As amostras de swab nasal, oral e de vesículas foram analisadas na PCR. De 93 amostras, apenas 26 indivíduos apresentavam vesículas e desses foram coletados swab. Após a realização da PCR, foram amplificadas 50% das amostras (13/26) e swab de vesículas. As amostras amplificadas e sequenciadas foram analisadas quanto às substituições nucleotídicas em cinco posições na ORF22, duas posições na ORF38 e duas posições na ORF54 (Figura 1).
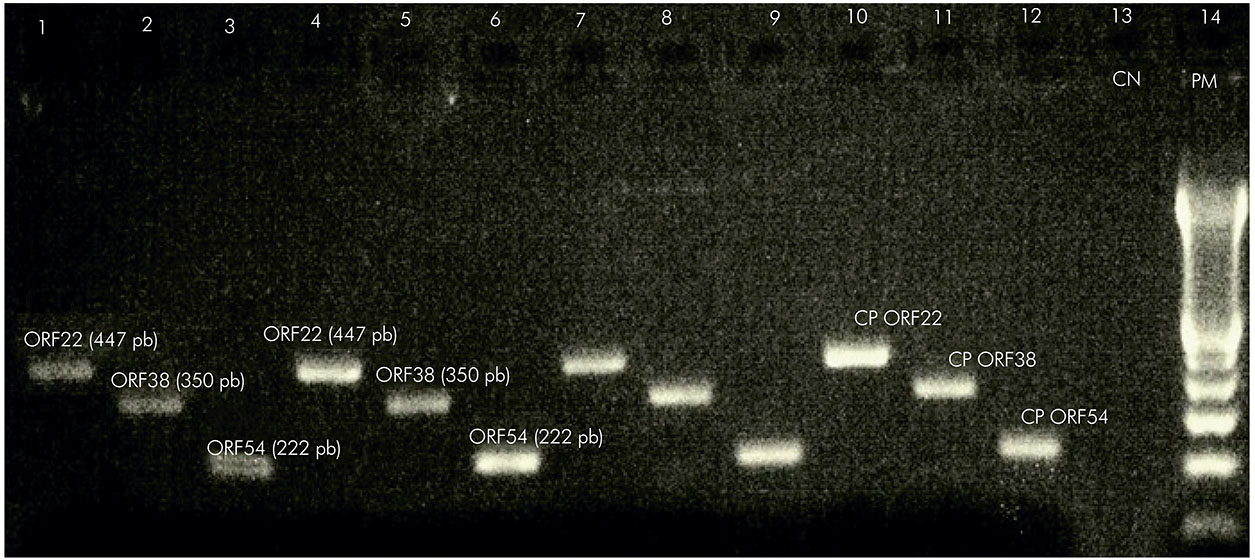
CP: controle positivo; CN: controle negativo; PM: peso molecular 123 pb.
Figura 1 - Visualização dos produtos das ORFs 22, 38 e 54 após PCR
As amostras VVZ118686 e VVZ119611 apresentaram substituições nucleotídicas nas posições A38081C da ORF22 e T95241C, C95300T da ORF54. As amostras VVZ118683, VVZ118684, VVZ118685, VVZ118845, VVZ119609, VVZ119701 e VVZ119954 apresentaram mudança de nucleotídeo na posição G69424A da ORF38 (Quadro 2).
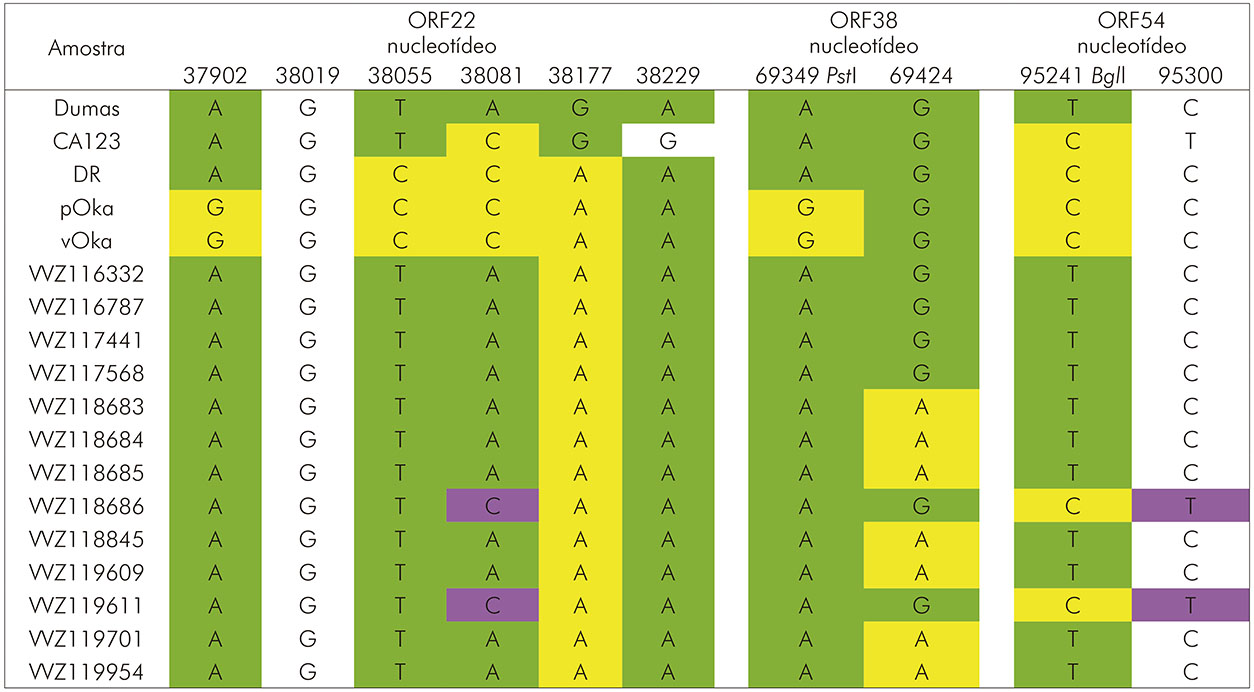
Números nas linhas superiores indicam ORF e a posição do SNP no genoma. As estirpes a seguir foram definidas como referência: Dumas do genótipo E; CA123 e DR do genótipo M; pOka e vOka do genótipo J, para comparação dos pares de bases de posições onde ocorreram mudança de nucleotídeo. O SNP da ORF54 é o sítio usado em alguns protocolos para discriminar a estirpe vacinal Oka de muitas estirpes desconhecidas. A cor lilás indica mutação individual intraespecífica; verde indica mutações comuns com a estirpe de referência do genótipo E; amarela indica mutações em comum com a estirpe Oka parental genótipo J; branco indica bases em comum para ambas estirpes de referência E e J.
Quadro 2 - Análise de variação genotípica utilizando dados de três ORFs do VVZ
Os genótipos 1 ou 3 foram encontrados em 11 amostras VVZ, enquanto que duas amostras apresentaram o genótipo 5 (Figura 2).

As estirpes de referência utilizadas foram: Dumas, representando o genótipo E; pOka e vOka referência do genótipo J; VZVi/Philadelphia.USA/73/V[3] referência do genótipo E; DR e Morroco 1 referência do genótipo M; e CA 123 referência do genótipo M. Os valores de bootstrap (2.000 pseudoréplicas) estão indicados nos nós da árvore.
Figura 2 - Análise filogenética utilizando o método de agrupamento de vizinhos (NJ) baseada no polimorfismo nucleotídico da ORF22 (447 pb), envolvendo 13 amostras dos Municípios de Belém, Ananindeua e Marituba
A análise filogenética mostra a distribuição das estirpes dentro das classes 1 ou 3 e classe 5. Essas amostras são oriundas dos Municípios de Ananindeua (sete casos de varicela e um de herpes-zoster), Belém (três casos de varicela e um de herpes-zoster) e Marituba (um caso de varicela). A descrição das estirpes e as sequências das amostras analisadas foram cadastradas no GenBank (Quadro 3).
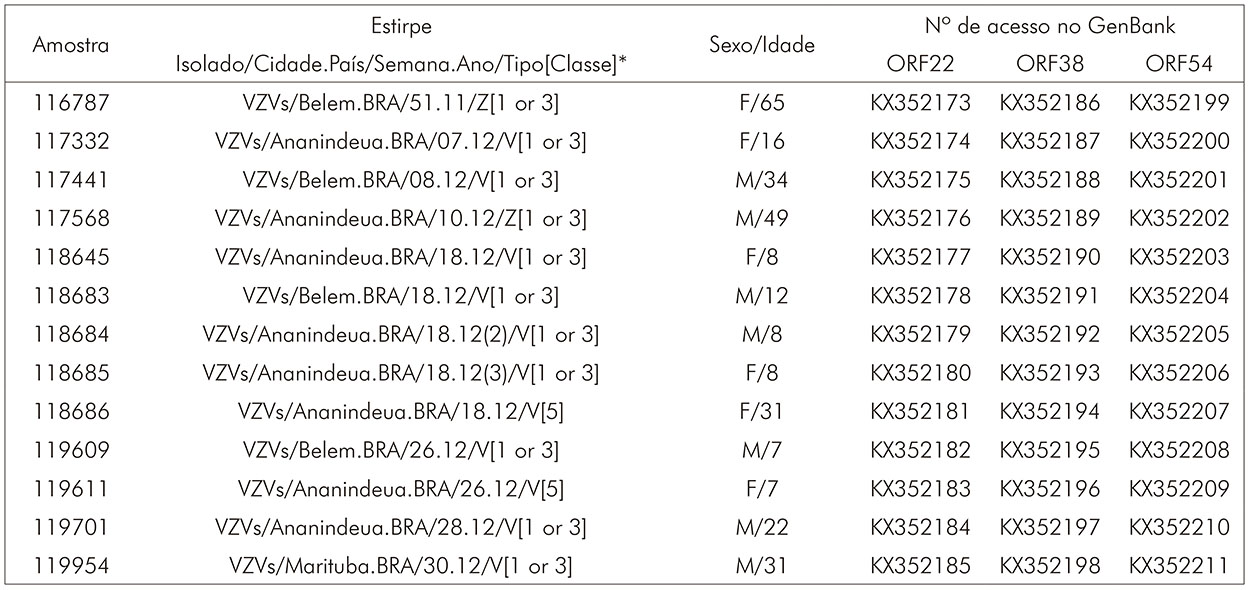
* Descrição da estirpe: espécime (representado pela sigla do vírus "VZV" e a letra "s" sigla da palavra specimen, ambas descritas em inglês), lugar de origem da amostra (cidade e país), período da coleta (semana e ano da coleta), tipo (amostra de origem de casos de varicela "V" ou zoster "Z") e a classe a que pertence a estirpe.
Quadro 3 - Descrição das estirpes e números de acesso no GenBank
DISCUSSÃO
Este é um estudo inédito de investigação genotípica do VVZ na Região Norte do Brasil, adotando SNP para análise das diferenças entre as classes 1 ou 3 e classe 52,13,15. A preferência por esse método baseou-se no fato de que as técnicas de genotipagem geralmente utilizadas no laboratório devem ser de simples execução e capazes de atingir resultados satisfatórios em amostras com quantidade limitada de DNA, como, por exemplo, a partir de vesículas e crostas.
Alguns estudos mostram que extração e purificação de ácido nucleico nem sempre são necessárias para realização da PCR36,37. A amplificação do material torna-se rápida, sensível e com um custo efetivo menor, principalmente quando muitas amostras são analisadas34. As amostras deste estudo foram imersas em solução de água destilada e armazenadas em freezer a -70º C por vários meses sem deterioração em qualidade, mesmo após vários ciclos de congelamento e descongelamento.
A PCR de material direto de vesículas é sensível e específica para detecção do VVZ, e espécimes de saliva podem funcionar como uma ferramenta de suporte para o diagnóstico em determinados casos em que, após extração com kit comercial, a PCR não funciona por baixa quantidade de DNA viral38.
Neste estudo, a PCR foi realizada em todas as amostras de swab nasal, oral e de vesículas. De 26 amostras coletadas de vesículas e processadas em água destilada submetidas a PCR, 13 (50%) foram amplificadas. As amostras foram coletadas do primeiro até 12º dia após o surgimento das vesículas na pele, sendo que as amostras amplificadas foram coletadas entre o primeiro até o nono dia após o início dos sintomas. O ideal é que essa coleta ocorra até o sétimo dia, quando as vesículas ainda estão produtivas38,39,40.
Chun et al39, em estudo com crianças que apresentaram varicela pós-vacinal, coletaram amostras de vesículas em swab, armazenaram em frasco estéril e posteriormente realizaram extração e purificação do DNA do VVZ com kit comercial, com positividade de 58%. Esses resultados foram superiores aos encontrados em outros dois estudos, que utilizavam a mesma metodologia de extração e purificação, com presença do DNA do VVZ em 46%40 e 35%41, respectivamente.
Resultados semelhantes foram encontrados em estudo realizado na Suíça em que o DNA do VVZ estava presente em 21 (47%) de 45 amostras de soro coletadas até os primeiros oito dias da fase aguda da doença42.
A análise das sequências das ORFs 22, 38 e 54 associou a semelhança das estirpes encontradas nesta pesquisa com as classificadas dentro das classes 1 ou 3 e classe 5. Do total de 13 estirpes isoladas, apenas duas apresentaram genótipo classe 5, sendo as demais classificadas em classe 1 ou 3. Vários estudos demonstraram uma distribuição geográfica distinta dos principais genótipos do VVZ quando compararam as regiões de clima temperado com as de clima tropical19,22,26,28,43,44,45. O domínio regional de genótipos específicos poderia ter sido estabelecido pelo clima e/ou outros fatores, como interações vírus-hospedeiro ou introdução de estirpes em uma população inócua ao VVZ, por meio da imigração ou viagens. Estratégias modernas de genotipagem podem ajudar a resolver essas questões. As regiões em que vários genótipos VVZ predominam, e esses dados, combinados com os padrões migratórios históricos humanos, contribuem para identificar os prováveis pontos de origem para esses genótipos19. A presença de dois tipos diferentes de genótipos neste estudo pode estar associada a um ou mais desses fatores.
Na Argentina, estudo utilizando a mesma técnica de análise da ORF22 em amostras de swab de vesículas encontrou apenas estirpes de genótipo E (classe 1 ou 3) em amostras de 13 crianças com varicela43.
Os resultados deste estudo, realizado na região metropolitana de Belém, também mostraram um predomínio da classe 1 ou 3, com 11 amostras de VVZ, e diferem, portanto, do estudo no qual, de dez amostras de VVZ coletadas no Brasil, oito estirpes foram classificadas como classe 5 e apenas duas como pertencentes à classe 1 ou 32,13.
O genoma do VVZ é muito estável e a variação na sequência do DNA observada ocorre, geralmente, como mutações pontuais individuais. Essa justificativa é plausível e indica que um pequeno número de variantes favorecidas ocorre independentemente entre as estirpes do tipo selvagem, em mais de uma localização geográfica. A literatura ressalta que não se pode descartar a possibilidade de que o VVZ de classe 5 tenha surgido, primeiramente, nas regiões tropicais, quando sofreram mutações depois que ocorreram as migrações humanas para o clima temperado. Estirpes classes 1, 2 e 3 podem ter surgido independentemente da 5 como resultado do isolamento das ilhas japonesas e as explorações e colonização dos europeus em diversos continentes, respectivamente2,13.
A análise das estirpes circulantes no Brasil permitiria, por exemplo, a associação de quadros clínicos graves à infecção por determinada estirpe. Além disso, estudos comparando as estirpes vacinais com estirpes VVZ circulantes no País possibilitariam uma avaliação mais detalhada em relação ao esclarecimento de sintomas pós-vacinais, pois permitiria avaliar se uma infecção está sendo causada pelo vírus vacinal ou se o indivíduo adquiriu, incidentalmente, o vírus selvagem próximo ao período de vacinação, associando o aparecimento de vesículas à vacina.
Na Índia, pesquisadores observaram, em avaliações clínicas, que estudantes com varicela apresentavam vesículas largas com tamanho maior que 8 mm em diâmetro, semelhante ao que ocorre com pacientes adultos do sexo masculino infectados pelo genótipo classe 5 do VVZ36. Duas regiões do VVZ que têm sido utilizadas para investigar as transmissões hospitalares são a R1 na ORF11 e OriS2,37,38. No entanto, os parâmetros precisos que informarão a sua utilização para a investigação de surtos ainda não estão estabelecidos. Os genótipos mais associados com hospitalizações de pacientes com varicela continuam desconhecidos, e a virulência de um genótipo pode ser influenciada mais pelo nível de resposta do hospedeiro e sua imunidade que por um genótipo específico46,47.
Uma vigilância quanto aos tipos de genótipos do VVZ é importante, especialmente para monitoramento de eventos adversos futuros causados pela varicela, e a análise de polimorfismos no genoma do vírus em momentos diferentes permitiria estimar sua taxa evolucionária48.
Neste estudo foi possível a avaliação do genótipo de apenas duas amostras de pacientes com herpes-zoster. A análise genotípica em amostras de pacientes com herpes-zoster poderia proporcionar uma avaliação mais detalhada de quanto tempo determinada estirpe circula no País (taxa evolucionária), além de avaliar as estirpes mais associadas à reativação viral e surgimento do herpes-zoster anos após a infecção primária pelo VVZ.
A infecção primária com VVZ ocorre, geralmente, durante a infância, por volta dos 10 anos de idade49. O herpes-zoster desenvolve-se a partir da reativação do VVZ e a estirpe isolada em casos de zoster é a mesma encontrada em infecções por esse vírus adquiridas na infância. Portanto, uma estirpe do VVZ isolado de uma paciente de 70 anos de idade com herpes-zoster, na Coreia do Sul, provavelmente circulou cerca de 60 anos atrás, na década de 194050. Neste estudo, a amostra de VVZ a partir de um caso de herpes-zoster em adulto de 65 anos de idade, oriundo de Belém, revelou estirpe do genótipo classe 1 ou 3, sugerindo que a mesma já circula por, no mínimo, cerca de cinco décadas no Brasil. Outra amostra de paciente com herpes-zoster também foi identificada como classe 1 ou 3, esse com idade de 45 anos, o qual sempre residiu em Ananindeua, mas serão necessários estudos de análise de taxa evolucionária para melhor estimar o período em que as classes 1 ou 3 foram introduzidas na região.
A análise das ORFs 21 e 50 é necessária para definição das estirpes de genótipo europeu (E1 e E2) em classe 1 ou 32,19. Outros estudos de genotipagem são necessários para acompanhar o possível impacto da vacinação na circulação de genótipos do VVZ no Brasil, além de possíveis associações com diferentes quadros clínicos e a possibilidade de análise da taxa evolucionária do vírus.
Este é um estudo inédito no Brasil, pois é a primeira vez que o VVZ tem o genótipo definido por testes realizados em uma instituição de pesquisa nacional.
CONCLUSÃO
Os genótipos do VVZ foram classificados em classes 1 ou 3 e classe 5. O genótipo classe 1 ou 3 foi encontrado nos Municípios de Ananindeua, Belém e Marituba, sendo que o classe 5 foi encontrado apenas em Ananindeua. A PCR foi sensível em 13 amostras de material direto de vesículas, enquanto que em amostras de saliva não foi obtido sucesso. A investigação das ORFs 21 e 50 é importante para definição das classes 1 e 3 nas amostras analisadas. Outros estudos são necessários para identificar os genótipos do VVZ circulantes no Brasil.










 texto em
texto em