INTRODUÇÃO
Os vírus transmitidos por artrópodes hematófagos, conhecidos como arbovírus, estão distribuídos em todos os continentes, exceto na Antártica, onde ainda não existem relatos de infecção por esses vírus. São mantidos em natureza por meio de ciclos biológicos, envolvendo hospedeiros vertebrados suscetíveis e artrópodes hematófagos, e transmitidos por repasto sanguíneo1.
Nas regiões tropicais, o ciclo de transmissão ocorre ao longo de todas as estações climáticas, uma vez que vetores e hospedeiros convivem no mesmo ambiente2, justificando assim o elevado número de isolamentos virais referentes a novos e possíveis arbovírus nessas regiões, sendo registrados no Catálogo Internacional dos Arbovírus e outros vírus de vertebrados (mais de 530 vírus). Na Região Amazônica, até o ano de 2010, cerca de 200 diferentes arbovírus foram isolados3.
Dentre as cinco principais famílias, nas quais a grande maioria dos arbovírus está distribuída (Bunyaviridae, Flaviviridae, Togaviridae, Reoviridae e Rhabdoviridae), destaca-se a família Bunyaviridae, por conter o maior número de arbovírus classificados em cinco gêneros: Tospovirus, Nairovirus, Phlebovirus, Hantavirus e Orthobunyavirus, esse último relevante para a saúde pública, pois apresenta arbovírus capazes de causar doença em humanos4,5. Os bunyavírus apresentam morfologia esférica e envelope viral, variando entre 80 a 120 nm de diâmetro. O material genético é constituído por três segmentos de RNA, sendo um pequeno (S - small), um médio (M - medium) e um grande (L - large)6.
Uma característica importante da família Bunyaviridae é o frequente rearranjo genético observado entre vírus que infectam uma mesma célula; durante o processo de montagem das partículas virais, trocam segmentos, dando origem a novos vírus rearranjados. O uso apenas de testes sorológicos clássicos dificulta a identificação desses vírus, uma vez que o teste de fixação do complemento (FC) detecta o segmento S e os testes de inibição da hemaglutinação (IH) e neutralização (TN) detectam o segmento M, demonstrando, por meio de testes diferentes, que possivelmente trata-se de um vírus rearranjado. Assim, nesses casos, o sequenciamento nucleotídico é de suma importância, pois a partir dele e pelas ferramentas moleculares, esses eventos são possíveis de serem identificados7.
O gênero Orthobunyavirus, que até o ano de 2005 era chamado de Bunyavirus8, compreende mais de 350 vírus isolados de diferentes espécies de vetores, como moscas e percevejos, muito embora a maioria dos orthobunyavírus seja transmitida por mosquitos9,10. Algumas espécies de mosquitos podem passar a transmitir determinadas arboviroses após alterações ambientais, como observado nos estudos realizados a partir da construção da usina hidrelétrica de Tucuruí, Estado do Pará, Brasil, onde o vírus Maguari, antes transmitido por sabetíneos, passou a ter mosquitos do gênero Anopheles como vetores11. Ademais, o gênero Orthobunyavirus apresenta grande diversidade de vírus e hospedeiros/vetores, o que torna a chance de isolar um vírus rearranjado muito maior. Assim, estudos de caracterização viral, com base nas características antigênicas e moleculares, são importantes, especialmente nos casos de vírus identificados nos testes sorológicos como pertencentes à família Bunyaviridae, gênero Orthobunyavirus. O objetivo deste estudo foi descrever a caracterização sorológica e molecular de três isolados virais provenientes de mosquitos capturados no Estado do Pará.
MATERIAIS E MÉTODOS
ISOLADOS VIRAIS
Neste estudo foram utilizados três isolados virais obtidos e cedidos pela Seção de Arbovirologia e Febres Hemorrágicas (SAARB) do Instituto Evandro Chagas (IEC). A cepa BeAR 544767 foi isolada a partir de um lote de mosquitos Wyeomyia sp. coletados na Floresta Nacional (Flona) de Caxiuanã, situada no nordeste do Estado do Pará (Lat. 2º15'S, Long. 52ºW / Lat. 1º30', Long. 51º15'), abrangendo os Municípios de Portel e Melgaço; enquanto que as cepas BeAR 643361 e BeAR 701402 foram isoladas a partir de mosquitos das espécies Limatus flavisetosus e Psorophora ferox, coletados em Altamira, Estado do Pará, no ano de 2001 e 2006, respectivamente. O estudo foi aprovado pelo Comitê de Ética no Uso de Animais do IEC, certificados n. 15/2009, 02/2014 e 04/2014.
ESTOQUE VIRAL
Foi produzido estoque viral em camundongos albinos suíços recém-nascidos, a partir de suspensão liofilizada de cérebros de camundongos infectados com as três cepas virais em estudo. O material liofilizado foi reconstituído com 0,5 mL de água destilada e 0,02 mL inoculado por via intracerebral nos camundongos recém-nascidos, que, após apresentarem sinais de doença, foram coletados e armazenados a -80º C para o desenvolvimento do estudo, conforme Lenette12.
CARACTERIZAÇÃO ANTIGÊNICA
A caracterização antigênica dos três isolados virais foi realizada pelos testes de FC e de TN em camundongos. O teste de FC foi realizado conforme a técnica descrita por Fulton e Dumbell13 e adaptada para microplacas por Beaty et al14. Foram utilizadas suspensões virais a partir de cérebros de camundongos infectados com os respectivos isolados, os quais foram testados em duas principais etapas: inicialmente com os diferentes pools de fluidos ascíticos imunes (FAI) da maioria dos arbovírus isolados pela SAARB/IEC (grupos antigênicos: A, B, C, Capim, Guamá, Bunyamwera, Anopheles A, Plebotomus, VSV, Timbó e Changuinola); e, em caso de reação positiva para um dos grupos antigênicos citados, realizaram-se outros testes com cada um dos integrantes virais do grupo. O teste de FC é interpretado a partir da percentagem de hemólise observada em uma escala de 0 a 4; no qual 0 é hemólise total das hemácias (100%); 1, hemólise de 75%; 2, hemólise de 50%; 3, hemólise de 25%; e 4, ausência de hemólise, 0%. Foi considerada positiva toda reação sem hemólise ou com até 25%. Os títulos do teste de FC foram classificados pelos valores da maior diluição positiva do anticorpo sobre a maior diluição positiva do antígeno.
O TN foi realizado por meio de diluições seriadas de 10 vezes do vírus e diluição constante do soro15. Os soros utilizados para essa metodologia foram selecionados a partir dos resultados do teste de FC. A interpretação dos resultados foi feita a partir do cálculo de DL50/0,02 mL, de acordo com Reed e Muench16, sendo considerados positivos os soros que apresentaram um índice logarítmico de neutralização (ILN) maior ou igual a 1,8. Esse teste é bastante utilizado com o intuito de elucidar possíveis reações cruzadas observadas entre vírus de um mesmo grupo antigênico, uma vez que apresenta maior especificidade do que o teste de FC, além de dar um indicativo de vírus rearranjados.
CARACTERIZAÇÃO MOLECULAR
Sequências genômicas
Para a obtenção do RNA viral dos três materiais analisados neste estudo, foram utilizadas culturas de células VERO infectadas apresentando 90% de efeito citopático. Todas as etapas para infecção dos cultivos celulares seguiram a técnica descrita por Lennette12. Duas plataformas de sequenciamento de nova geração - 454 FLX (Roche(r), Branford, Connecticut, EUA) e Ion PGMTM (Ion Torrent, Life Technologies, Califórnia, EUA) - foram utilizadas para a obtenção das sequências genômicas, as quais foram precedidas de uma etapa adicional que consistiu na concentração das partículas virais a partir de 10 mL de sobrenadante de células VERO infectadas com os respectivos isolados virais.
Os materiais foram centrifugados a 2.000 rotações por minuto (rpm) por 10 min e o sobrenadante foi transferido para um novo tubo e submetido a uma nova etapa de centrifugação a 4.000 rpm por 5 min. O novo sobrenadante foi filtrado em filtro estéril de 0,2 µm e transferido para um novo tubo. Em seguida, adicionou-se 1,48 mL de NaCl 5M estéril e 10,8 mL de polietilenoglicol (PEG) 8.000 (Sigma-Aldrich(r), Missouri, EUA) em 6 mL do sobrenadante filtrado. A suspensão foi então incubada a 4º C por 30 min e, posteriormente, centrifugada a 6.000 rpm por 1 h a 4º C. O sobrenadante foi descartado e o pellet resultante da centrifugação com PEG foi ressuspendido em 250 µL de tampão fosfato salino (phosphate buffered saline - PBS); posteriormente, foi adicionado 12,5 µL de RNAse para degradar o RNA de interferência uma vez que a partícula viral ainda se encontrava íntegra. A suspensão foi incubada por 30 min a 37º C. Finalmente, foram adicionados mais 238 µL de PBS, a fim de adequar a amostra para extração do RNA.
A extração do RNA viral ocorreu utilizando PureLinkTM Viral RNA Mini Kit (Invitrogen, Califórnia, EUA) para a plataforma 454 FLX e TRIzol LS Reagente (Invitrogen), e o kit QIAamp RNA Viral para plataforma Ion PGM, ambos seguindo-se orientação dos fabricantes. A quantificação da concentração do RNA viral dos três materiais foi realizada pelo equipamento QubitTM, de acordo com instruções do fabricante. A faixa de concentração mínima ideal de RNA para o sequenciamento foi de 20 ng/µL.
O genoma viral foi obtido a partir do método de pirosequenciamento (454 FLX) e sequenciamento semicondutor de íon, Ion PGMTM. Embora sejam metodologias diferentes, o processo de sequenciamento nucleotídico é semelhante entre ambas e ocorreu em três etapas: preparo da biblioteca genômica e seleção dos tamanhos dos fragmentos; PCR em emulsão e enriquecimento; e reação de sequenciamento nucleotídico. Todas as etapas foram realizadas de acordo com as instruções dos respectivos fabricantes.
Análise bioinformática
As leituras geradas pelas plataformas 454 e PGM foram montadas utilizando a metodologia De Novo por meio dos programas Newbler v3 e Mira v4, respectivamente. As sequências montadas foram comparadas com outras sequências de vírus depositadas no GenBank (http://www.ncbi.nlm.nih.gov/), com o intuito de avaliar a homologia entre as mesmas. Posteriormente, as análises de curadoria foram realizadas utilizando o programa Geneious v6 (Biomatters, Nova Zelândia).
As sequências dos isolados virais foram comparadas com sequências nucleotídicas de vírus disponíveis no GenBank. Para construção das árvores filogenéticas, as sequências foram alinhadas no programa BioEdit. As árvores foram construídas empregando-se o método de máxima verossimilhança (MV)17 em conjunto com o teste de bootstrap, fixando 1.000 réplicas para gerar maior confiabilidade aos valores dos grupamentos18.
RESULTADOS
CARACTERIZAÇÃO ANTIGÊNICA
Os três isolados virais em estudo (BeAR 544767, BeAR 643361 e BeAR 701402), inicialmente, reagiram positivamente no teste de FC para o pool de FAI do sorogrupo Bunyamwera (vírus Tucunduba - VTUC, vírus Taiassui - VTAI, vírus Iaco, vírus Sororoca, vírus Maguari, vírus Kairi, vírus Xingu e vírus Guaroa) e, após testar os vírus em estudo para todos os integrantes desse grupo antigênico, os três isolados virais apresentaram reação cruzada para VTUC e VTAI, com títulos descritos na tabela 1.
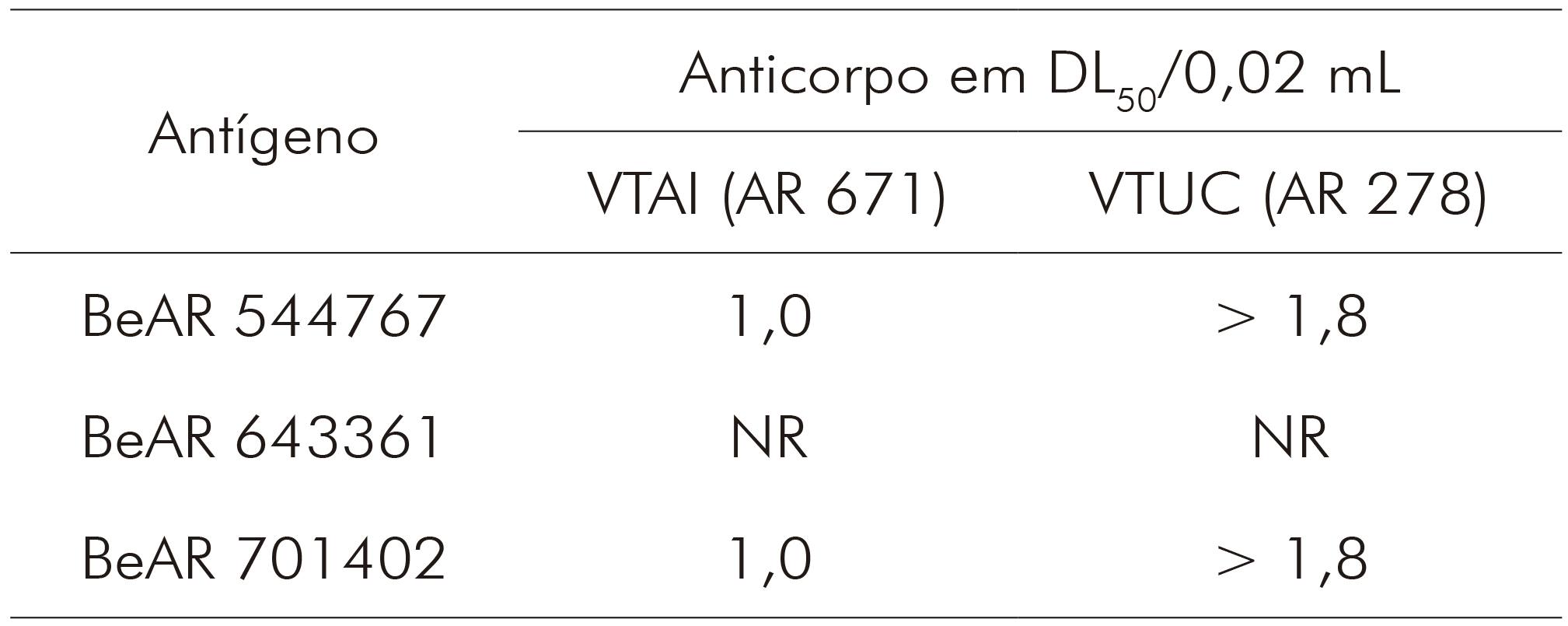
Tabela 1 - Resultados dos testes de fixação do complemento dos isolados virais BeAR 544767, BeAR 643361 e BeAR 701402 com VTAI e VTUC

No TN, os isolados virais BeAR 544767 e BeAR 701402 foram neutralizados apenas pelo soro do VTUC (AR 278) com índice logarítmico de neutralização maior ou igual a 1,8 (Tabela 2). Para o vírus BeAR 643361, não foi realizado o TN.
CARACTERIZAÇÃO MOLECULAR
A região codificante completa de cada um dos isolados virais em estudo é constituída de três segmentos de RNA fita simples: um segmento pequeno (S-RNA), um segmento médio (M-RNA) e um segmento grande (L-RNA) com tamanho semelhante entre eles (Tabela 3), flanqueada pelas regiões 5' e 3'. O segmento S-RNA codifica uma única proteína, referente ao nucleocapsídeo, a proteína N; o segmento M-RNA codifica duas proteínas estruturais, as glicoproteínas Gn e Gc, e uma proteína não estrutural, a proteína NSm; e o segmento L-RNA apresenta uma única cadeia aberta de leitura (CAL), a qual codifica a proteína L que corresponde a RNA-polimerase RNA-dependente.
Tabela 3 - Tamanho dos segmentos de RNA dos isolados BeAR 544767, BeAR 643361 e BeAR 701402

CAL: Cadeia aberta de leitura; nt: Nucleotídeo; S-RNA: Segmento pequeno; M-RNA: Segmento médio; L-RNA: Segmento grande.
Filogenia
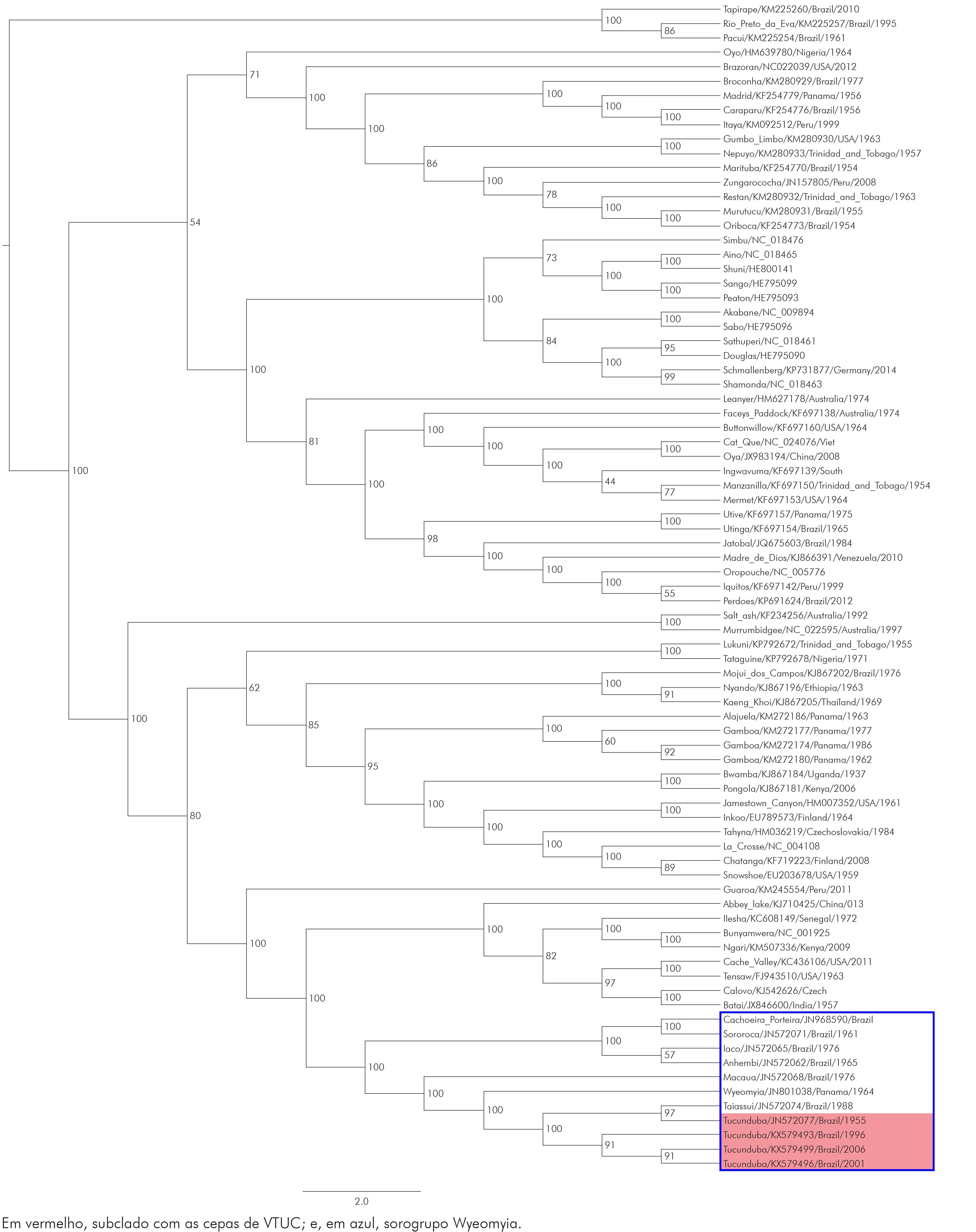
As análises filogenéticas dos isolados virais foram realizadas utilizando as sequências aminoacídicas da CAL dos três segmentos pelo método MV. Na árvore filogenética gerada para o segmento S, foi possível observar que os membros do gênero Orthobunyavirus ficaram agrupados em clados representando os diversos grupos existentes, sustentados por valores de bootstrap entre 85% a 100%.
O complexo Wyeomyia formou um clado bastante consistente, dividido em dois subclados, em que os vírus Iaco, Anhembi, Cachoeira Porteira e Sororoca formaram um desses ramos; enquanto os isolados virais em estudo e os vírus VTUC, VTAI e Wyeomyia (VWYO) formaram o outro subclado, sustentado por valor máximo de bootstrap de 100% para esse ramo (Figura 1). A análise filogenética para o segmento M (poliproteína) evidenciou um padrão de organização semelhante ao observado para o segmento S (Figura 2).
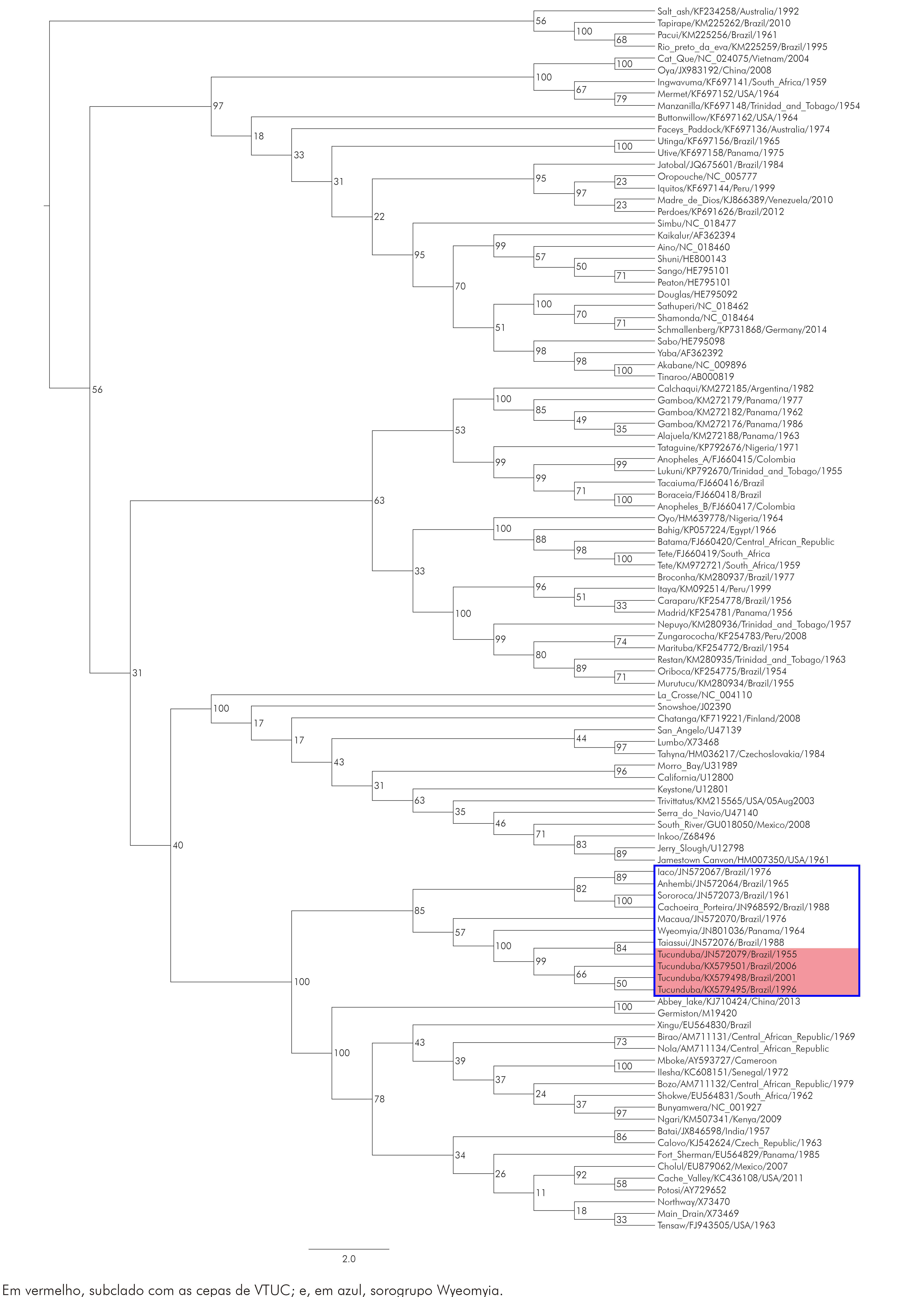
Figura 1 - Árvore filogenética com base na sequência aminoacídica do S-RNA dos isolados virais BeAR 544767, BeAR 643361 e BeAR 701402 pelo método MV
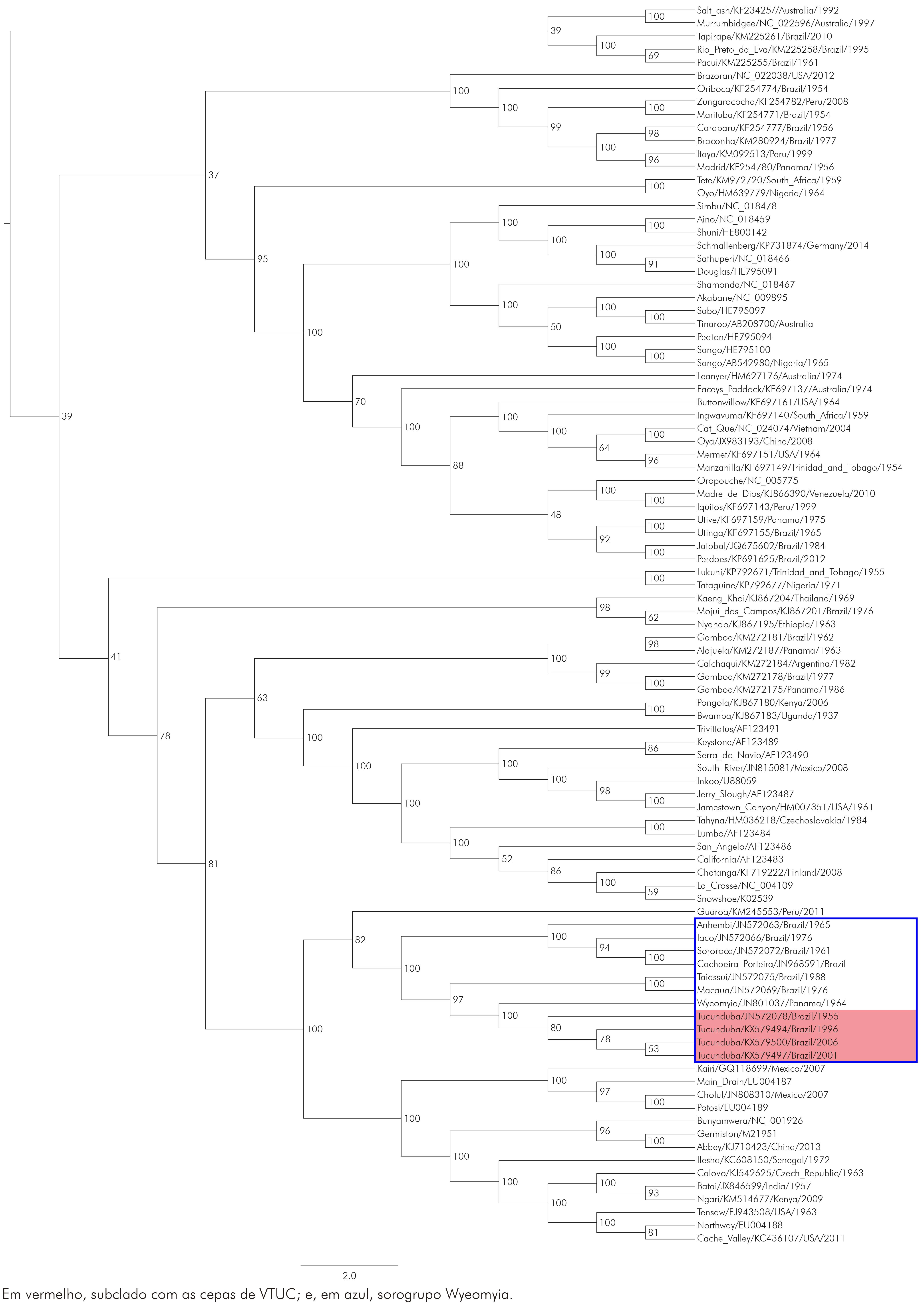
Figura 2 - Árvore filogenética com base na sequência aminoacídica do M-RNA dos isolados virais BeAR 544767, BeAR 643361 e BeAR 701402 pelo método de MV
O complexo Wyeomyia foi dividido em dois subclados, porém o segmento M parece ser o mais divergente dentre os três segmentos. Nesta análise os três isolados formaram um ramo monofilético juntamente com o VTUC e com o VWYO, apresentando valores de bootstrap igual a 100%. O mesmo padrão de agrupamento visto para os dois segmentos anteriores também é visualizado na filogenia do segmento L, mas, nesse segmento, os isolados virais são mais relacionados ao VTAI, sustentado por um valor de bootstrap de 100% (Figura 3).
DISCUSSÃO
A caracterização antigênica definida pelos testes de FC e TN é uma das ferramentas utilizadas na caracterização viral de uma cepa. Os cruzamentos sorológicos compartilhados pelas três cepas estudadas com o VTUC e com o VTAI confirmam, por sorologia, o princípio de que vírus relacionados entre si apresentam cruzamento sorológico, principalmente quando se trata de vírus trisegmentados. Além disso, mesmo os isolados virais estudados sendo cepas do VTUC, é possível observar reação positiva de título igual ou muito semelhante com o VTAI.
Esse fato está relacionado à proteína determinante no teste de FC, uma vez que o teste é determinado pelo segmento S (nucleocapsídeo) que, dentre os três segmentos dos orthobunyavírus, é o que mais compartilha características genéticas entre os vírus do grupo7,19. Reações sorológicas cruzadas também foram constatadas por Shope e Causey20, onde um relacionamento antigênico foi observado entre seis vírus pertencentes ao grupo C (Bunyaviridae, Orthobunyavirus), demonstrando que esses vírus estão relacionados entre si; e, quando analisados pelos testes FC, TN e IH, verifica-se que um mesmo vírus é identificado por um, dois ou até pelos três testes sorológicos citados. Posteriormente, aquele estudo foi complementado por Nunes et al21, os quais observaram que 13 vírus do grupo C apresentavam rearranjos naturais entre eles, corroborando e complementando o estudo de Shope e Causey20. Além disso, os vírus com estreito relacionamento ao infectar uma mesma célula suscetível podem ter seus segmentos genômicos variavelmente incorporados dentro das progênies virais7, ou seja, o segmento de um vírus passa a constituir o genoma do outro vírus, podendo com isso formar um novo vírus rearranjado.
Para complementar os resultados do teste de FC, foi realizado o TN, que detecta as glicoproteínas (Gn e Gc) localizadas no segmento M, as quais apresentam maior especificidade genética para cada vírus, uma vez que a região conservada da glicoproteína Gn possui determinantes antigênicos de tipo específico para anticorpos neutralizantes e hemaglutinantes22. Por outro lado, acredita-se que a glicoproteína Gc seja a principal determinante para a realização da etapa de adsorção durante o ciclo de replicação dos vírus nas células de mamíferos23. Dessa forma, o TN apresenta maior especificidade para a pesquisa de anticorpos neutralizantes, sendo assim o padrão ouro das provas sorológicas24.
Devido à organização genômica, essas cepas claramente referem-se a vírus pertencentes à família Bunyaviridae, obedecendo às características moleculares descritas por Elliott e Blakqori25 para incluir um vírus na referida família. A única exceção foi a ausência da proteína não estrutural NSs codificada pelo segmento S, presente em outros orthobunyavírus. Os tamanhos dos segmentos também corroboram os descritos por King et al4 e Chowdhary et al26, que observaram tamanhos semelhantes para outros orthobunyavírus.
A ausência da proteína NSs, descrita por Mohamed et al27, demonstra que alguns vírus do gênero Orthobunyavirus, como os vírus do sorogrupo Tete, Anopheles A e Anopheles B, não codificam essa proteína. Chowdhary et al26 também evidenciaram a ausência da NSs nos vírus do complexo Wyeomyia, no qual o VTUC está incluído.
A partir das análises filogenéticas é possível observar a formação do sorogrupo Wyeomyia, composto por VTAI, VTUC, VWYO, vírus Macauã, vírus Sororoca, vírus Anhembi, vírus Cachoeira Porteira e vírus Iaco, tal como descrito por Chowdhary et al26. O sequenciamento das três cepas do VTUC no presente estudo representa, ainda, mais um fortalecimento a esse clado, pois agrega representatividade à árvore filogenética. Ademais, a forma como os três segmentos desse vírus se agrupa também corrobora o que Chowdhary et al26 propuseram: o VTUC como um rearranjo de VTAI e VWYO (Darien), justificando o estreito relacionamento do VTUC ora no segmento S e L com o VTAI ora no segmento M com o VWYO.
Tendo como base as informações expostas, caracterizaram-se, sorológica e molecularmente, os isolados virais BeAR 544767, BeAR 643361 e BeAR 701402 como cepas do VTUC, vírus descrito pela primeira vez a partir de sucessivos isolamentos de mosquitos na Colômbia, Panamá, Trinidad e Guiana Francesa, além de um isolamento a partir de uma pessoa febril no Panamá1. No Pará, o VTUC foi isolado a partir de um lote de mosquitos capturados na floresta de Oriboca, região próxima à Belém, em 19553. Os hospedeiros vertebrados desse vírus ainda são desconhecidos, sendo relatado apenas um único caso humano descrito em Belém, onde a criança apresentou um quadro clínico típico de uma arbovirose com complicações neurológicas, diagnosticado como encefalite28.
CONCLUSÃO
No presente estudo, foram descritas três cepas do VTUC, ressaltando a diversidade dos vetores fontes desses isolados virais, uma vez que consistem em três espécies diferentes de mosquitos, demonstrando que esse vírus está adaptado ao meio em que circula. Outro achado importante desta pesquisa foram os anos de captura de cada lote de mosquito, visto que a cepa BeAR 544767 foi isolada em Caxiuanã, em 1996, e as cepas BeAR 643361 e BeAR 701402 foram isoladas a partir de lotes de mosquitos capturados em Altamira, nos anos de 2001 e 2006, respectivamente, demonstrando que, mesmo silenciosamente, o VTUC permanece circulando e que merece atenção, uma vez que se trata de um rearranjo viral com potencial para causar doenças em humanos.











 Curriculum ScienTI
Curriculum ScienTI