Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista Pan-Amazônica de Saúde
versão impressa ISSN 2176-6223versão On-line ISSN 2176-6223
Rev Pan-Amaz Saude v.8 n.1 Ananindeua mar. 2017
http://dx.doi.org/10.5123/s2176-62232017000100002
ARTÍCULO ORIGINAL
Caracterización molecular de Escherichia coli enteropatógena atípica (EPEC) en animales silvestres capturados en la Región Amazónica
1Instituto Evandro Chagas/SVS/MS, Ananindeua, Pará, Brasil
OBJETIVO:
Identificar animales silvestres como reservorios de Escherichia coli enteropatógena atípica (EPEC).
MATERIALES Y MÉTODOS:
Se analizaron los factores de virulencia de EPEC (eae y bfp) en 263 muestras de E. coli aisladas de 260 animales silvestres capturados en tres municipios del Estado de Pará (Marabá, Parauapebas y Canaã dos Carajás) de marzo de 2008 a diciembre de 2009. Los métodos de investigación aplicados fueron la PCR, utilizando primers específicos, seguida de la secuenciación del gen eae.
RESULTADOS:
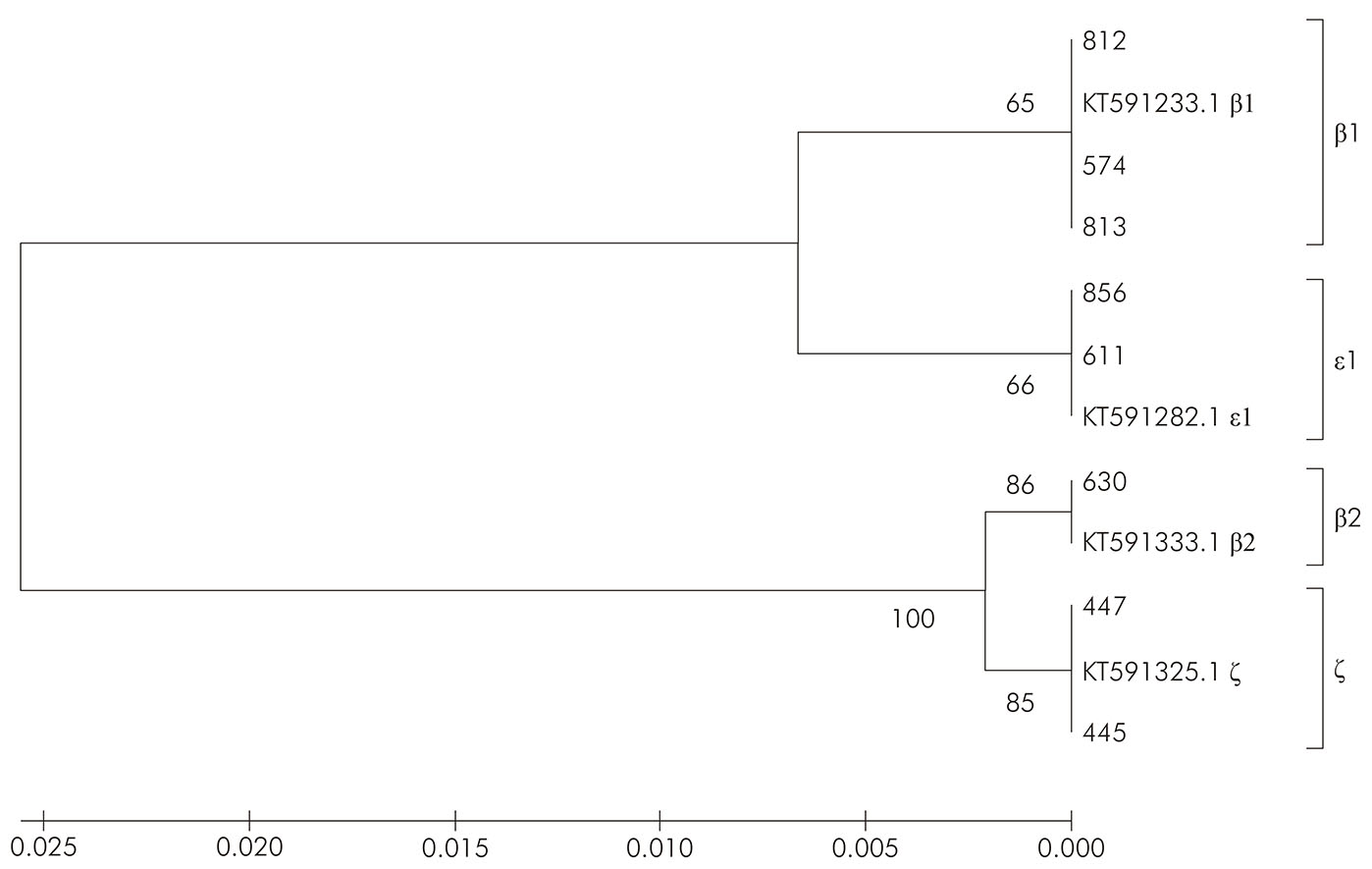
Entre las 263 muestras de E. coli evaluadas, se observaron 3,04% (8/263) como EPEC, con 2,66% (7/263) en roedores y 0,4% (1/263) en marsupiales. Entre las muestras analizadas, se observó la presencia de cuatro variantes de intimina: β1 (muestras 574, 812 y 813), β2 (muestra 630), ζ (muestras 445 e 447) y ε (muestras 611 y 856). Luego del análisis filogenético por el método UPGMA, se obtuvo el árbol consenso, que presentó la formación de dos grupos: el primero compuesto por KT591282.1, ε 1 intimina (611 y 856) con KT591233.1, β1 intimina, (574, 812 y 813); y el segundo por KT591325.1, ζ intimina (445 y 447) con KT591333.1, β2 intimina (630).
CONCLUSIÓN:
Los datos demuestran que las EPEC aisladas de los animales silvestres tienen características genéticas semejantes a las observadas en humanos, y es posible que los animales analizados estén sirviendo de reservorio para las EPEC circulantes.
Palabras clave: Escherichia coli Enteropatógena Atípica (EPEC); Animales Silvestres; Reservorios; Diversidad Genética
INTRODUCCIÓN
Cepas de Escherichia coli colonizan el tracto gastrointestinal de humanos y animales unas pocas horas después del nacimiento y son el principal miembro anaeróbico facultativo de la microbiota intestinal de estos organismos1. Esta especie, perteneciente a la familia Enterobacteriaceae, raramente causa enfermedad en sus huéspedes. A pesar de su papel como comensales, algunas cepas de E. coli son capaces de causar una variedad de enfermedades debido a la adquisición de factores de virulencia por transferencia horizontal1. Entre estas enfermedades, la diarrea es una de las principales, siendo las E. coli diarrogénicas (ECD) responsables de numerosos brotes de diarrea registrados en el mundo2.
Actualmente se conocen seis patotipos de ECD: E. coli enteroinvasiva (EIEC); E. coli enterotoxigénica (ETEC); E. coli enteropatógena (EPEC), subdividida en EPEC típica (tEPEC) y EPEC atípica (aEPEC); E. coli productora de toxina de Shiga (STEC), y su subgrupo E. coli enterohemorrágica (EHEC); E. coli enteroagregativa (EAEC); y E. coli de adherencia difusa (DAEC)1. Entre estos patotipos, las cepas EPEC se destacan como un importante agente diarrogénico presente en los países en desarrollo2.
Recientemente, una nueva clasificación fue propuesta por Clements et al.3, en la cual las E. coli se dividen en ocho patotipos: los seis anteriormente descritos, más E. coli adherente invasiva (AIEC), asociada a la enfermedad de Crohn ; y E. coli enteroagregativa productora de la toxina Shiga (STEAEC), responsable de un gran brote diarreico en 2011 en Europa.
Las EPEC pueden definirse por la presencia del gen eae, que codifica una proteína denominada intimina, que se requiere para la adhesión a la mucosa intestinal, produciendo una lesión de tipo attaching and effacing (A/E). Esta lesión se inicia por la adhesión de la bacteria a los enterocitos, llevando a la pérdida de las microvellosidades intestinales. La intimina es altamente variable entre los diferentes serotipos de EPEC, y algunas variantes antigénicas distintas han sido identificadas como α, β, γ, δ, ε, ι, κ, η, ζ, entre otras4. Las EPEC pueden clasificarse en típicas y atípicas. Los linajes que poseen el plásmido EAF (EPEC adherence factor) se llaman tEPEC y los que no poseen se llaman aEPEP. El gen bfp contenido en el plásmido EAF codifica un factor de adhesión denominado bundle-forming pili (BFP), siendo que el plásmido EAF también posee genes necesarios para la expresión de la intimina2.
Las cepas aEPEC pertenecen a un grupo diarrogénico que ha aumentado mucho su frecuencia en Brasil en los últimos años. En algunas regiones del país, estas bacterias parecen haber sustituido a las tEPEC como principal agente diarrogénico. Esto indica que, en Brasil, así como ocurrió en países desarrollados, está habiendo un cambio en el perfil epidemiológico de las EPEC de típicas para atípicas3,5.
Las cepas tEPEC raramente son aisladas de animales, siendo el hombre su principal reservorio
natural6. En contraste, las cepas aEPEC son aisladas de humanos y animales, con o sin diarrea, en frecuencias equivalentes, lo que no permite la definición de un depósito para esas bacterias7,8.
Diversos estudios asocian animales domésticos, de cría y algunos silvestres como posibles reservorios y fuente de infección de aEPEC para humanos9. A pesar de ello, ninguno comparó, de forma amplia, muestras aisladas de humanos y animales por técnicas de biología molecular, como la secuenciación, para comprobar tales hipótesis.
Orígenes clonales comunes entre cepas bacterianas aisladas de animales y humanos pueden identificar fuentes de infección y reservorios animales de diferentes patógenos. Las muestras de ECD, para los animales y para el hombre, la mayoría de las veces, poseen factores de virulencia similares, codificados por genes de origen cromosómico o plasmídico, cuyas similitudes o diferencias pueden explicar aspectos filogenéticos y patógenos de ciertos grupos de E. coli para humanos.
El análisis epidemiológico molecular de cepas bacterianas procedentes de diferentes orígenes es una evaluación de las relaciones de parentesco existentes entre ellas. La principal cuestión de estos análisis es si diferentes aislados de un determinado patógeno representan la diseminación de un organismo común o clon. En este sentido, la epidemiología ha buscado desarrollar técnicas que proporcionen una mejor evaluación de las relaciones existentes entre diferentes aislados bacterianos.
El concepto básico de las técnicas de tipificación molecular propone que muestras bacterianas, epidemiológicamente relacionadas, poseen un precursor común; de esta forma, estudios utilizando esas técnicas permiten una mejor comprensión sobre los mecanismos evolutivos, sobre el surgimiento de cepas virulentas y reservorios de cepas bacterianas10. Frente a lo expuesto, debido a los posibles riesgos para la salud pública traído por linajes de EPEC, se necesitan estudios que evalúen la distribución y la frecuencia de estas cepas en los animales, permitiendo la comparación con cepas de EPEC aisladas de humanos.
MATERIALES Y MÉTODOS
OBTENCIÓN DE LAS MUESTRAS
Se obtuvieron muestras fecales o porciones de los intestinos delgados y gruesos, linfáticos y mesentéricos y fragmentos de hígado y bazo de 630 animales silvestres (roedores, aves y marsupiales) de ambos sexos, capturados en tres municipios del estado de Pará (Marabá, Parauapebas y Canãa dos Carajás), de marzo de 2008 a diciembre de 2009 (Figura 1). Los especímenes fecales recogidos fueron acondicionados en medio de transporte Cary Blair y, junto con el pool de órganos mantenidos bajo refrigeración, encaminados directamente al Laboratorio de Enteroinfecciones Bacterianas del Instituto Evandro Chagas (IEC), en donde fueron procesadas.

Figura 1 - Localización de los municipios de Parauapebas, Marabá y Canaã dos Carajás, Estado de Pará, Brasil
Los experimentos con animales fueron aprobados por el Instituto Brasileño del Medio Ambiente y de los Recursos Naturales Renovables, proceso nº 02018001038/08-11.
AISLADO E IDENTIFICACIÓN BIOQUÍMICA
Para aislar el E. coli, el material fecal en suspensión o el pool de órganos fue sembrado en medios Agar MacConkey - MC (Difco, EE.UU.) e incubado a una temperatura de 35-37 ºC, por 18-24 h. Después de ese período, las colonias sospechosas de E. coli fueron sometidas a los medios de clasificación Triple Sugar Iron Agar - TSI (Difco, EE.UU.). Para la caracterización bioquímica de los aislados bacterianos se utilizó el kit VITEK® 2 (bioMérieux, Brasil).
IDENTIFICACIÓN DE LAS CATEGORÍAS DE E. coli DIARROGÉNICAS
Tres a cinco aislados de E. coli seleccionados de cada animal fueron mantenidos en caldo Luria y glicerol a -70 ºC para análisis de EPEC, EIEC, STEC, ETEC y EAEC, utilizando la reacción en cadena de la polimerasa (PCR) Multiplex11,12. Los controles positivos (EPEC E2348/69, EHEC EDL 931, ETEC H10407, EIEC EC 299/07, EAEC 042) y negativo (K12 DH5α) fueron gentilmente cedidos por la Dra. Tânia Vaz, del Instituto Adolfo Lutz, São Paulo.
ESTUDIO DE GENES DE VIRULENCIA DE E. coli
Las cepas se cultivaron en Agar Nutriente (Difco, EUA) para ser utilizadas en el procedimiento de extracción de ADN. El material extraído se almacenó a -20 ºC por un tiempo máximo de 20 días para ser utilizado en las reacciones de PCR Multiplex. Para esa,
se utilizaron iniciadores descritos por Aranda et al.13 y Toma et al.14 (Tabla 1). Las cepas identificadas como aEPEC tuvieron su gen eae secuenciado, utilizando la plataforma ABI 3130 (Applied Biosystems, EUA) con el kit ABI PRISM™ Dye Terminator Cycle Sequencing (Applied Biosystems, EUA). Las secuencias producidas se editaron en el programa Sample Manager, acoplado al ABI 3130 DNA Sequencer. Para el análisis de las secuencias, se utilizaron los programas BioEdit v7.1.315, Mega v5.0516 y DnaSP v517.
Tabla 1 - Iniciadores utilizados en la PCR Multiplex y sus respectivos productos de amplificación
| Iniciadores | Secuencia (5'-3') | Gen-blanco | Productos de amplificación (pb) | Referencia |
|---|---|---|---|---|
| eae-1 eae-2 | CTGAACGGCGATTACGCGAA CGAGACGATACGATCCAG | eae | 917 | Aranda et al.13 |
| BFP-1 BFP-2 | AATGGTGCTTGCGCTTGCTGC GCCGCTTTATCCAACCTGGTA | bfpA | 326 | Aranda et al.13 |
| aggRks-1 aggRksa-2 | GTATACACAAAAGAAGGAAGC ACAGAATCGTCAGCATCAGC | aggR | 254 | Toma et al.14 |
| LT-f LT-r | GGCGACAGATTATACCGTGC CGGTCTCTATATTCCCTGTT | elt | 450 | Aranda et al.13 |
| ST-f ST-r | ATTTTTMTTTCTGATTTRTCTT CACCCGGTACARGCAGGATT | est | 190 | Aranda et al.13 |
| IpaH-1 IpaH-2 | GTTCCTTGACCGCCTTTCCGATACCGTC GCCGGTCAGCCACCCTCTGAGAGTAC | ipaH | 600 | Aranda et al.13 |
| VTcom-u VTcom-d | GAGCGAAATAATTTATATGTG TGATGATGGCAATTCAGTAT | stx1/stx2 | 518 | Toma et al.14 |
EXTRACCIÓN DEL ADN Y PCR MULTIPLEX
El ADN de los aislados, caracterizados fenotípicamente como E. coli y de las cepas de referencia de los controles positivos y negativos, se extrajo por el método de ebullición y congelación, siguiendo las recomendaciones de Starnbach et al.18 y Baloda et al.19.
La PCR se hizo a partir de 2 μl de cada ADN extraído y 23 μl de la solución mix, conteniendo entre 0,5 y 1,5 μl, de acuerdo con cada iniciador (Invitrogen, Brasil); 10 mM de dNTP mix dATP, dCTP, dGTP, dTTP (Invitrogen, Brasil); 0,5 U de Platinum Taq DNA Polymerase; Casquillo Taq 1X; 50 mM de MgCl2 (Invitrogen, Brasil) y agua estéril ultra pura para un volumen final de 25 μL. Las preparaciones de la PCR Multiplex se colocaron en el termociclador automático de gradiente modelo VeritiTM 96-Well Thermal Cycler (Applied Biosystems, EE.UU.) y fueron sometidas a ciclos específicos de amplificación que consistió de un paso de 2 min a 50 ºC (hot-start), de 5 min a 95 ºC (desnaturalización inicial), seguido de 40 ciclos de 1 s a 95 ºC, 50 ºC y 72 ºC, y un paso final de extensión de 7 minutos a 72 ºC.
Los amplicones se analizaron por electroforesis en gel de agarosa al 2%, coloreado con bromuro de etideo (10 mg/ml) en tampón TBE (Tris base 0,89M, ácido bórico 0,45 M, EDTA 1 mM, pH 8,4) y visualizados bajo luz UV con ayuda de un transiluminador (Vilber Lourmat, Francia). Como marcador de tamaño molecular, se utilizó el Ladder de 1 Kb plus (Invitrogen, Brasil). Posteriormente, el gel fue fotografiado por un sistema de fotodocumentación, Bioimaging Systems (UPV, EE.UU.).
SECUENCIACIÓN DEL GEN eae DE LA aEPEC
Los fragmentos de los genes eae obtenidos de ocho aislados fueron purificados con el kit comercial Kit BigDye® X Terminator (Applied Biosystems, EUA) y secuenciados para confirmación, usando el forward
primer específico para el gen eae. Para la secuenciación se preparó un mix conteniendo 200-300 ng del ADN sospechoso y el primer específico en una concentración de 1,5 pmol/mL, y, posteriormente, alineadas y comparadas con las secuencias para el gen eae aEPEC: aisladas de pacientes diarreicos, (KT591325.1 ζ e KT591333.1 β2), aislada de animal (KT591282.1 ε1) y aislada de humano y animal (KT591233.1 β1).
RESULTADOS
De los 630 cultivos analizados, heces y agrupación de órganos, se aislaron E. coli en 263 (41,7%) muestras (Tabla 2). Después de la PCR, se observó la amplificación para el gen eae en 3,04% (8/263), distribuidos en roedores con 2,66% (7/263) y marsupiales con 0,4% (1/263) (Tabla 3; Figura 2).
Tabla 2 - Número de E. coli aisladas de animales silvestres capturados en tres municipios del Estado de Pará (Marabá, Parauapebas y Canaã dos Carajás), Brasil
| Clases | E. coli | no DEC* | |
|---|---|---|---|
| Nº de aislados | aEPEC | ||
| Aves | 34 | - | 34 |
| Marsupiales | 22 | 1 | 21 |
| Quelonios | - | - | - |
| Roedores | 195 | 7 | 188 |
| Lagomorfos | - | - | - |
| Reptiles | 12 | - | 12 |
| Total | 263 | 8 | 255 |
* E. coli no diarrogénica; Señal convencional utilizada: - Dato numérico igual a cero no resultante de redondeo.
Tabla 3 - Cepas EPEC atípicas (aEPEC) aisladas de animales silvestres (roedores y marsupiales) capturados en tres municipios del Estado de Pará (Marabá, Parauapebas y Canaã dos Carajás), Brasil
| Registro IEC | Animal | Nombre científico | Municipio |
|---|---|---|---|
| 445 | Roedor | Proechimys guyannensis | Parauapebas |
| 447 | Roedor | Proechimys guyannensis | Parauapebas |
| 574 | Marsupial | Monodelphis emiliae | Parauapebas |
| 611 | Roedor | Oligoryzomys sp. | Parauapebas |
| 630 | Roedor | Oryzomys capito | Canaã dos Carajás |
| 812 | Roedor | Oxymycterus sp. | Parauapebas |
| 813 | Roedor | Oxymycterus sp. | Parauapebas |
| 856 | Roedor | Oxymycterus sp. | Parauapebas |
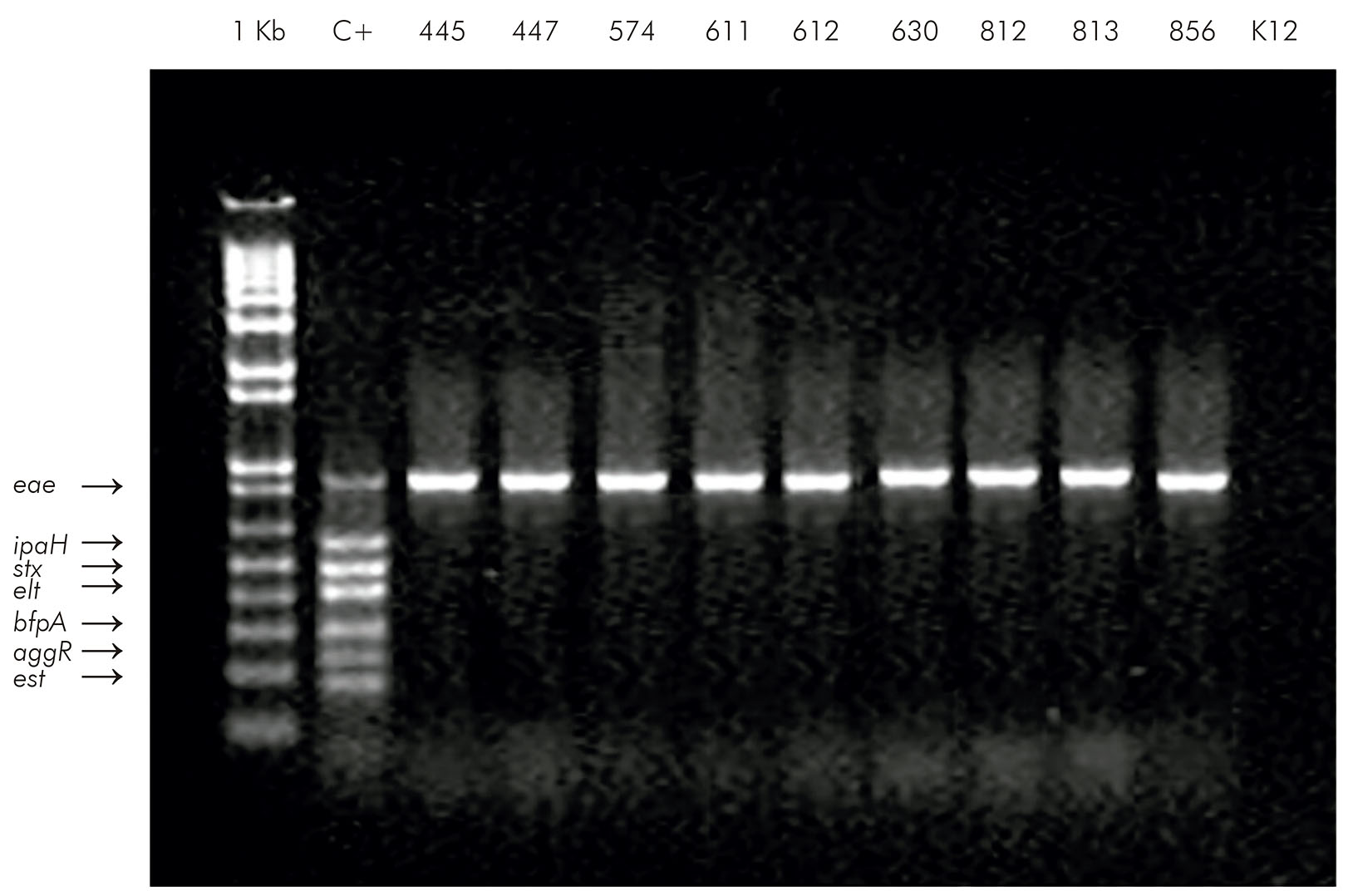
Figura 2 - PCR Multiplex: 1- ladder 1 Kb; 2- control positivo (cinco categorías de DEC); 4, 5, 6, 7, 8, 9, 10, 11 e 12- aEPEC; y 13- control negativo (E. coli K12 DH5)
Después de la PCR y la secuenciación del gen eae, las muestras fueron alineadas por el programa BioEdit v7.1.315 con las mutaciones descritas en la Tabla 4. Se observaron tasas de transición/transversión (R) igual a 7.47, según modelo de Kimura20 2-parámetros. El análisis de la prueba de Tajima's Neutrality demostró una diversidad nucleotídica (π) de 0.014298 para un número de segregaciones por sitio igual a 24 en los 917pb del gen eae de las ocho muestras analizadas. Después de analizar las secuencias del gen eae de las aEPEC de los ocho aislados de origen animal, se observó la presencia de cuatro variantes distintas para la intimina, que fueron β1 (muestras 574, 812 y 813), β2 (muestra 630), ζ (muestras 445 Y 447) y ε (muestras 611 y 856).
Tabla 4 - Alineamiento de las secuencias obtenidas con la utilización del BioEdit v7.1.3 con sus respectivos puntos de mutaciones
| Puntos polimórficos | 717 | 741 | 783 | 786 | 810 | 909 | 978 | 987 | 990 | 999 | 1012 | 1014 | 1023 | 1035 | 1040 | 1041 | 1050 | 1053 | 1089 | 1096 | 1113 | 1137 | 1257 | 1293 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| KT591325.1_zeta_intimina | A | A | G | T | T | T | A | C | C | T | C | G | C | T | G | G | T | G | T | C | T | T | T | A |
| EAE_445 | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| EAE_447 | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| KT591333.1_beta2_intimina | . | . | . | . | C | . | . | . | . | . | . | . | . | C | . | . | . | . | . | . | . | . | . | . |
| EAE_630 | . | . | . | . | C | . | . | . | . | . | . | . | . | C | . | . | . | . | . | . | . | . | . | . |
| KT591282.1_epsilon1_intimina | C | G | T | C | . | C | G | T | T | C | T | A | T | C | A | A | C | A | C | T | C | C | C | G |
| EAE_611 | C | G | T | C | . | C | G | T | T | C | T | A | T | C | A | A | C | A | C | T | C | C | C | G |
| EAE_856 | C | G | T | C | . | C | G | T | T | C | T | A | T | C | A | A | C | A | C | T | C | C | C | G |
| KT591233.1_beta1_intimina | C | T | T | . | C | C | G | T | T | C | T | A | T | C | A | A | C | A | C | T | C | C | C | G |
| EAE_574 | C | T | T | . | C | C | G | T | T | C | T | A | T | C | A | A | C | A | C | T | C | C | C | . |
| EAE_812 | C | T | T | . | C | C | G | T | T | C | T | A | T | C | A | A | C | A | C | T | C | C | C | G |
| EAE_813 | C | T | T | . | C | C | G | T | T | C | T | A | T | C | A | A | C | A | C | T | C | C | C | G |
Los nucleótidos idénticos se indican por puntos; KT591325.1, KT591333.1, KT591282.1, KT591233.1: Secuencias de referencias para las clases de intiminas analizadas.
Después del análisis filogenético por el método de agrupamiento de pares no ponderados con base en la media aritmética (unweighted pair group method with arithmetic mean - UPGMA), el árbol consenso presentó la formación de dos grupos, compuestos de dos ramas distintas cada uno, agrupando, en el primer grupo, KT591282.1, ε1 intimina (611 y 856) con KT591233.1, β1 intimina (574, 812 y 813); y en el segundo grupo, KT591325.1, ζ intimina (445 y 447) con KT591333.1, β2 intimina (630) (Figura 3).
DISCUSIÓN
Los marcadores de virulencia analizados revelaron que las aEPEC aisladas poseen características genéticas similares a las encontradas en humanos con las secuencias del gen eae y se conservan en las muestras 445, 447 y 630, provenientes de aislados de roedores. Estudios anteriores demostraron la presencia de animales como reservorio de cepas consideradas como potenciales agentes de enfermedades que pueden ser transmitidas entre seres humanos y animales21,22,23.
Estudios con aEPEC se han centrado principalmente en la comparación entre cepas aisladas de casos diarreicos y de pacientes saludables24,25,26. Más recientemente, la identificación de la presencia de tipos de EPEC potencialmente patógenos para humanos, aislados de animales, puede indicar que la transmisión de estos microorganismos entre animales y seres humanos ocurre y tiene impacto sobre la salud pública6; pero poco se ha evaluado acerca de la ocurrencia de estos patógenos en animales silvestres. De esta forma, el presente estudio buscó la identificación de las subclases de intiminas en animales silvestres y observó la ocurrencia de las subclases ζ, β1, β2 y ε, como las que están circulando en ellos. Como estas subclases ya se han descrito en seres humanos y animales de cría6,24,25,26, los datos pueden apuntar a una posible contaminación de la fauna silvestre por cepas provenientes de la contaminación ambiental o que esos especímenes están sirviendo de reservorios naturales para ese patógeno.
Entre los animales estudiados, los roedores presentaron mayor frecuencia de aEPEC, con muestras presentando secuencias altamente conservadas para el gen eae, cuando comparadas a aislados humanos6. Sólo un marsupial fue identificado como portador de la aEPEC, pero la secuencia aislada presentó un 100% de similitud con las observadas en roedores. La gran diversidad genómica de E. coli le confiere una notable plasticidad ecológica; gracias a ella, estos microorganismos se adaptan rápidamente a diferentes ambientes, pudiendo así pasar de organismo de vida libre a comensal del tracto intestinal de los animales de sangre caliente y, aún, a patógenos que infectan a humanos y a animales6,9,25,27,28,29,30.
Xu et al.6, al analizar las subclases de intiminas en humanos, animales y alimentos, identificaron las intiminas λ, β2, μ, ι2, ξ, ζ, α2 y π en pacientes diarreicos. La subclase γ1 se identificó concomitantemente en pacientes diarreicos y en cerdos; la intimina κ, identificada tanto en alimentos (carne cruda) como en pacientes diarreicos; y las clases ε2, η2 y ι1, tanto en pacientes diarreicos y asintomáticos, demostrando la gran variabilidad y distribución de las aEPEC entre las muestras analizadas. Anteriormente, Moura et al.31 describieron el subtipo β1 como el más frecuente en aislados de animales y humanos; en ese mismo sentido, Aidar-Ugrinovich et al.22 ya habían descrito que, en Brasil, el subtipo β1 se presentaba como el más frecuente en EPEC aisladas de bovinos; similar a lo observado por Cortés et al.32, que identificaron el subtipo β1 como el más prevalente en las cepas aisladas de ovinos, seguido del subtipo γ2. En el presente estudio, los subtipos más frecuentes fueron β1 (muestras 574, 812 y 813), β2 (muestra 630), ζ (muestras 445 y 447) y ε (muestras 611 y 856), presentes en ocho aEPEC de las 263 E. coli Identificadas. Sin embargo, como no se aislaron tEPEC, y 255 muestras fueron caracterizadas como no diarreicas, se sugiere que la variabilidad existente puede ser mayor, como se observa en la literatura, reforzando la necesidad de más estudios que aclaren la variabilidad genética existente entre las EPEC aisladas de humanos y animales en la Región Amazónica.
CONCLUSIÓN
Los datos demostraron que las aEPEC aisladas de los animales silvestres poseen características genéticas semejantes y mostraron proximidad con el gen eae de aEPEC encontradas en humanos y en animales de cría, posibilitando inferir que los animales evaluados sirven como posibles reservorios de cepas de origen humano, bien como ser potenciales transmisores de cepas animales con carácter zoonótico.
REFERENCIAS
1 Kapper JB, Nataro JP, Mobley HLT. Pathogenic Escherichia coli. Nat Rev Microbiol. 2004 Feb;2(2):123-40. Doi: 10.1038/nrmicro818 [Link] [ Links ]
2 Hu J, Torres AG. Enteropathogenic Escherichia coli: foe or innocent bystander? Clin Microbiol Infect. 2015 Aug;21(8):729-34. Doi: 10.1016/j.cmi.2015.01.015 [Link] [ Links ]
3 Clements A, Young JC, Constantinou N, Frankel G. Infection strategies of enteric pathogenic Escherichia coli. Gut Microbes. 2012 Mar-Apr;3(2):71-87. Doi: 10.4161/gmic.19182 [Link] [ Links ]
4 Ooka T, Seto K, Kawano K, Kobayashi H, Etoh Y, Ichihara S. Clinical significance of Escherichia albertii. Emerg Infect Dis. 2012 Mar;18(3):488-92. Doi: 10.3201/eid1803.111401 [Link] [ Links ]
5 Hernandes RT, Elias WP, Vieira MAM, Gomes TAT. An overview of atypical enteropathogenic Escherichia coli. FEMS Microbiol Lett. 2009 Aug;297(2):137-49. Doi: 10.1111/j.1574-6968.2009.01664.x [Link] [ Links ]
6 Xu Y, Bai X, Zhao A, Zhang W, Ba P, Liu K, et al. Genetic diversity of intimin gene of atypical enteropathogenic Escherichia coli isolated from human, animals and raw meats in China. PLoS One. 2016 Mar;11(3):e0152571. Doi: 10.1371/journal.pone.0152571 [Link] [ Links ]
7 Leomil L, Pestana de Castro AF, Krause G, Schmidt H, Beutin L. Characterization of two major groups of diarrheagenic Escherichia coli O26 strains which are globally spread in human patients and domestic animals of different species. FEMS Microbiol Lett. 2005 Aug;249(2):335-42. Doi: 10.1016/j.femsle.2005.06.030 [Link] [ Links ]
8 Aidar-Ugrinovich L, Blanco J, Blanco M, Blanco JE, Leomil L, Dahbi G, et al. Serotypes, virulence genes, and intimin types of Shiga toxin-producing Escherichia coli (STEC) and enteropathogenic E. coli (EPEC) isolated from calves in São Paulo, Brazil. Int J Food Microbiol. 2007 Apr;115(3):297-306. Doi: 10.1016/j.ijfoodmicro.2006.10.046 [Link] [ Links ]
9 Ishii S, Meyer KP, Sadowsky MJ. Relationship between phylogenetic groups, genotypic clusters, and virulence gene profiles of Escherichia coli strains from diverse human and animal sources. Appl Environ Microbiol. 2007 Sep;73(18):5703-10. Doi: 10.1128/AEM.00275-07 [Link] [ Links ]
10 Goering RV. Pulsed-field gel electrophoresis. In: Persing DH, Tenover FC, Versalovic J, Tang Y-W, Unger E, Relman DA, et al. Molecular microbiology: diagnostic principles and practice. Washington: ASM Press; 2004. p. 185-96. [ Links ]
11 Aranda KRS, Fabbricotti SH, Fagundes- Neto U, Scaletsky ICA. Single multiplex assay to identify simultaneously enteropathogenic, entroaggregative, enterotoxigenic, enteroinvasive and Shiga toxin-producing Escherichia coli strains in Brazilian children. FEMS Microbiol Lett. 2007 Jan;267(2):145-50. Doi: 10.1111/j.1574-6968.2006.00580.x [Link] [ Links ]
12 Sousa EB. Aspectos microbiológicos e epidemiológicos da doença diarréica aguda no município de Juruti, Pará [dissertação]. Belém (PA): Universidade Federal do Pará, Instituto de Ciências Biológicas; 2010. [Link] [ Links ]
13 Aranda KRS, Fagundes-Neto U, Scaletsky ICA. Evaluation of multiplex PCRs for diagnosis of infection with diarrheagenic Escherichia coli and Shigella spp. J Clin Microbiol. 2004 Dec;42(12):5849-53. Doi: 10.1128/JCM.42.12.5849-5853.2004 [Link] [ Links ]
14 Toma C, Lu Y, Higa N, Nakasone N, Chinen I, Baschkier A, et al. Multiplex PCR assay for identification of human diarrheagenic Escherichia coli. J Clin Microbiol. 2003 Jun;41(6):2669-71. Doi: 10.1128/JCM.41.6.2669-2671.2003 [Link] [ Links ]
15 Hall TA. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucl Acids Sym Ser. 1999;41:95-8. [Link] [ Links ]
16 Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. Mega5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Mol Biol Evol. 2011 Oct;28(10):2731-9. Doi: 10.1093/molbev/msr121 [Link] [ Links ]
17 Librado P, Rozas J. DnaSP v5: a software for comprehensive analysis of DNA polymorphism data. Bioinformatics. 2009 Apr;25(11):1451-2. Doi: 10.1093/bioinformatics/btp187 [Link] [ Links ]
18 Starnbach MN, Falkow S, Tompkins LS. Species-specific detection of Legionella pneumophila in water by DNA amplification and hibridization. J Clin Microbiol. 1989 Jun;27(6):1257-61. [Link] [ Links ]
19 Baloda SB, Krovacek K, Eriksson L, Linné T, Månsson I. Detection of aerolysin gene in Aeromonas strains isolated from drinking water, fish, and foods by polymerase chain reaction. Comp Immunol Microbiol Infect Dis. 1995 Jan;18(1):17-26. Doi: 10.1016/0147-9571(94)E0001-A [Link] [ Links ]
20 Kimura M. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences. J Mol Evol. 1980 Dec;16(2):111-20. Doi: 10.1007/BF01731581 [Link] [ Links ]
21 Rodrigues J, Thomazini CM, Morelli A, Batista GCM. Reduced etiological role for enteropathogenic Escherichia coli in cases of diarrhea in Brazilian infants. J Clin Microbiol. 2004 Jan;42(1):398-400. Doi: 10.1128/JCM.42.1.398-400.2004 [Link] [ Links ]
22 Aidar-Ugrinovich L, Blanco J, Blanco M, Blanco JE, Leomil L, Dahbi G, et al. Serotypes, virulence genes, and intimin types of Shiga toxin-producing Escherichia coli (STEC) and enteropathogenic E. coli (EPEC) isolated from calves in São Paulo, Brazil. Int J Food Microbiol. 2007 Apr;115(3):297-306. Doi: 10.1016/j.ijfoodmicro.2006.10.046 [Link] [ Links ]
23 Carvalho VM, Irino K, Onuma D, Pestana de Castro AF. Random amplification of polymorphic DNA reveals clonal relationships among enteropathogenic Escherichia coli isolated from non-human primates and humans. Braz J Med Biol Res. 2007 Feb;40(2):237-41. Doi: 10.1590/S0100-879X2006005000043 [Link] [ Links ]
24 Afset JE, Anderssen E, Bruant G, Harel J, Wieler L, Bergh K. Phylogenetic backgrounds and virulence profiles of atypical enteropathogenic Escherichia coli strains from a case-control study using multilocus sequence typing and DNA microarray analysis. J Clin Microbiol. 2008 Jul;46(7):2280-90. Doi: 10.1128/JCM.01752-07 [Link] [ Links ]
25 Contreras CA, Ochoa TJ, Lacher DW, DebRoy C, Navarro A, Talledo M, et al. Allelic variability of critical virulence genes (eae, bfpA and perA) in typical and atypical enteropathogenic Escherichia coli in Peruvian children. J Med Microbiol. 2010 Jan;59(Pt 1):25-31. Doi: 10.1099/jmm.0.013706-0 [Link] [ Links ]
26 Ramachandran V, Brett K, Hornitzky MA, Dowton M, Bettelheim KA, Walker MJ, et al. Distribution of intimin subtypes among Escherichia coli isolates from ruminant and human sources. J Clin Microbiol. 2003 Nov;41(11):5022-32. Doi: 10.1128/JCM.41.11.5022-5032.2003 [Link] [ Links ]
27 Carvalho VM, Gyles CL, Ziebell K, Ribeiro MA, Catão-Dias JL, Sinhorini IL, et al. Characterization of monkey enteropathogenic Escherichia coli (EPEC) and human typical and atypical EPEC serotype isolates from neotropical nonhuman primates. J Clin Microbiol. 2003 Mar;41(3):1225-34. Doi: 10.1128/JCM.41.3.1225-1234.2003 [Link] [ Links ]
28 Nakazato G, Gyles C, Ziebell K, Keller R, Trabulsi LR, Gomes TAT, et al. Attaching and effacing Escherichia coli isolated from dogs in Brazil: characteristics and serotypic relationship to human enteropathogenic E. coli (EPEC). Vet Microbiol. 2004 Aug;101(4):269-77. Doi: 10.1016/j.vetmic.2004.04.009 [Link] [ Links ]
29 Krause G, Zimmermann S, Beutin L. Investigation of domestic animals and pets as a reservoir for intimin- (eae) gene positive Escherichia coli types. Vet Microbiol. 2005 Mar;106(1-2):87-95. Doi: 10.1016/j.vetmic.2004.11.012 [Link] [ Links ]
30 Pitondo-Silva A, Nakazato G, Falcão JP, Irino K, Martinez R, Darini AL, et al. Phenotypic and genetic features of enteropathogenic Escherichia coli isolates from diarrheal children in the Ribeirão Preto metropolitan area, São Paulo State, Brazil. APMIS. 2015 Feb;123(2):128-35. Doi: 10.1111/apm.12314 [Link] [ Links ]
31 Moura RA, Sircili MP, Leomil L, Matté MH, Trabulsi LR, Elias WP, et al. Clonal relationship among atypical enteropathogenic Escherichia coli strains isolated from different animal species and humans. Appl Environ Microbiol. 2009 Dec;75(23):7399-408. Doi: 10.1128/AEM.00636-09 [Link] [ Links ]
32 Cortés C, De la Fuente R, Blanco J, Blanco M, Blanco JE, Dhabi G, et al. Serotypes, virulence genes and intimin types of verotoxin-producing Escherichia coli and enteropathogenic E. coli isolated from healthy dairy goats in Spain. Vet Microbiol. 2005 Sep;110(1-2):67-76. Doi: 10.1016/j.vetmic.2005.06.009 [Link] [ Links ]
Se refiere al doi: 10.5123/S2176-62232017000100002, publicado originalmente en portugués. Traducido por: Lota Moncada
Cómo citar este artículo / How to cite this article: Rocha DCC, Marinho ANR, Santos SD, Loureiro ECB. Caracterización molecular de Escherichia coli enteropatógena atípica (EPEC) en animales silvestres capturados en la Región Amazónica. Rev Pan-Amaz Saude. 2017 enero-marzo;8(1):1-8. Doi: http://dx.doi.org/10.5123/S2176-62232017000100002
Recibido: 29 de Abril de 2016; Aprobado: 07 de Octubre de 2016










 texto em
texto em