Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista Pan-Amazônica de Saúde
versão impressa ISSN 2176-6223versão On-line ISSN 2176-6223
Rev Pan-Amaz Saude vol.8 no.4 Ananindeua out./dez. 2017
http://dx.doi.org/10.5123/s2176-62232017000400006
ARTIGO DE OPINIÃO
Epigenômica, epigenética e câncer*
1Universidade Católica de Brasília, Programa de Pós-Graduação em Ciências Genômicas e Biotecnologia, Brasília, Distrito Federal, Brasil
2Northwestern University, Feinberg School of Medicine, Cancer Biology and Epigenomics Program, Chicago, Illinois, USA
3Genomic Enterprise, Chicago, Illinois, USA
4Genome Connect, Chicago, Illinois, USA
O Projeto Genoma Humano identificou que existem aproximadamente 22.000-25.000 genes codificadores de proteínas anotados em um genoma haploide de 3 bilhões de pares de bases, uma lista quase completa dos genes humanos. No entanto, existe novas informações além da sequência de DNA. As Epigenética e epigenômica são campos emergentes que começaram a ter um enorme impacto na biomedicina. Amplamente definida, a epigenética refere-se aos mecanismos baseados na cromatina importantes na regulação da expressão gênica que não envolvem alterações na sequência de DNA1. A epigenômica se refere às modificações epigenéticas em um tipo de célula em um tempo específico2. Recentemente, a epigenética foi considerada o epicentro da biomedicina moderna, devido ao estudo da hereditariedade não relacionada à sequência de DNA que pode ajudar a explicar a relação entre a origem genética de um indivíduo, do meio ambiente, envelhecimento e de uma doença3. A modificação epigenética mais estudada é a metilação do DNA, uma adição covalente de um grupo metil (CH3) à citosina. Além disso, a densidade de histonas, modificações pós-traducionais e mecanismos baseados no RNA também estão incluídos1. A importância da metilação do DNA é enfatizada pelo número crescente de doenças humanas que ocorrem quando essa informação epigenética não é devidamente estabelecida; e há um interesse crescente no desenvolvimento de formas de reverter farmacologicamente essas anormalidades, algumas já aprovadas pela Food and Drug Administration (FDA)2. A metilação do DNA pode ser observada em doenças como câncer, retardo mental, doenças psiquiátricas, doenças neonatais, doença de Alzheimer, entre outras2,4. A seguir, resumo da maquinaria molecular associada à epigenômica, com foco no câncer e na oncologia (Figura 1).
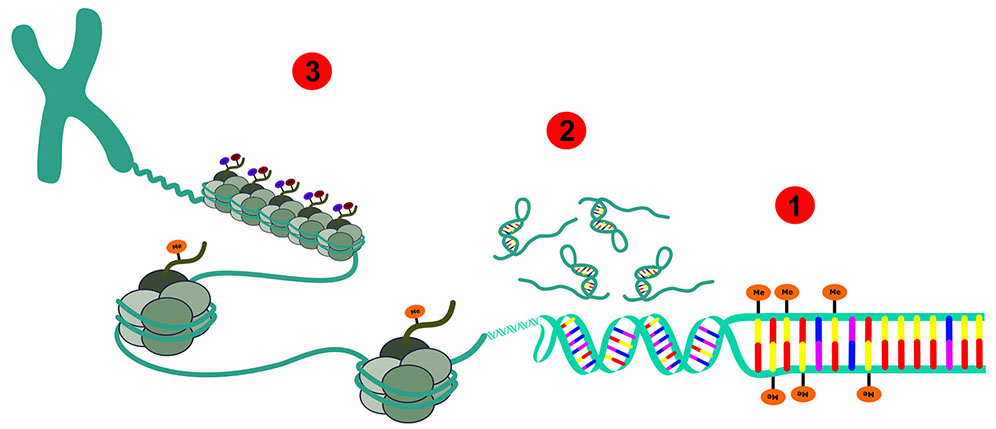
Representação esquemática: 1 - Metilação do DNA; 2 - RNAs não codificantes; 3 - Modificações de histonas.
Figura 1 - Representação de todas as modificações epigenéticas que podem ocorrer em um epigenoma eucariótico de células normais e cancerosas e a cromatina
METILAÇÃO DE DNA E CÂNCER
A metilação do DNA foi descoberta por Hotchkiss (1948) no timo de bezerros e ocorre principalmente no quinto carbono das citosinas5. O nucleosídeo 5-metil-desoxicitidina (5mC) é frequentemente considerado a quinta base do genoma. A distribuição da metilação do DNA no genoma eucariótico, estudada por Sinsheimer em 1955, revelou > 90% de 5mC metilada na sequência CpG6. Nos primórdios dos estudos epigenéticos, afirmava-se que a metilação da citosina no DNA eucariótico poderia ser uma modificação herdada de forma estável que afetava a diferenciação celular. Recentemente muitos processos envolvendo a metilação do DNA foram estudados, como a variação específica do tipo de célula em padrões de metilação, envolvimento de metilação em processos celulares, regulação de genes, interações de proteínas de DNA, diferenciação celular, supressão de elementos transponíveis, embriogênese, inativação do X, imprinting genômico e tumorigênese2,4. Além disso, medicamentos desenvolvidos para atingir o epigenoma das células cancerosas já foram aprovados pelo FDA para alguns tipos de doenças malignas2.
Resumidamente, os padrões de metilação do DNA são apagados no estágio embrionário inicial e restabelecidos em cada indivíduo no momento da implantação. Como consequência, isso permite que as células-tronco embrionárias se diferenciem em qualquer célula especializada possível. Os dados de 5mC do relatório de Sinsheimer (1955) sugeriram que > 90% da metilação do DNA no timo de bezerros é determinada pela sequência de dinucleotídeo CpG6. A hipótese de que os resíduos 5mC são locais preferenciais para uma mutação espontânea de DNA poderia explicar parcialmente a sub-representação do dinucleotídeo CG (formas metiladas mais as não metiladas). No DNA total e nos genes estruturais de eucariotos superiores, há uma ocorrência preferencial de 5mC no DNA não codificador altamente repetitivo7. Acredita-se que a base 5mC sofra uma mutação de transição via desaminação para produzir timina, que por sua vez pode resultar no acúmulo de TpG e CpA, com depleção de CpG, durante a evolução7.
A desregulação da metilação do DNA estaria associada a vários tipos de câncer e diferentes estágios da carcinogênese2,4,8. Recentemente, foi demonstrado que o DNA genômico de mamíferos contém não apenas 5-metilcitosina, mas também 5-hidroximetilcitosina (5hmC), que agora é considerada a sexta base do genoma em organismos superiores9. Curiosamente, o hmC foi encontrado no DNA de mamíferos em 197210, embora sua presença permanecesse incerta porque os estudos posteriores não foram capazes de reproduzir os dados. Ainda há informações limitadas sobre a nova sexta base que foi detectada pela primeira vez nos neurônios e não está (ou está fracamente) presente nas linhas de células cancerosas (por exemplo, HeLa)9.
MODIFICAÇÕES DE HISTONA E DINÂMICA DE CROMATINA NO CÂNCER
A cromatina, associada a promotores ativos, é composta por nucleossomos espaçados com histonas centrais que estão em estado altamente acetilado11. Essa situação, junto com muitas outras modificações pós-traducionais, fornece máxima ativação e transcrição de genes. Existem muitos mecanismos propostos para o silenciamento de genes. A outra situação, trabalhando em conjunto com a metilação do DNA, são modificações de histonas que também são conhecidas como "Código das Histonas".
O genoma eucariótico é empacotado em cromatina, uma estrutura altamente ordenada que contém DNA, RNA, histonas e outras proteínas cromossômicas. A cromatina foi originalmente classificada em eucromatina e heterocromatina, regiões ativas e inativas, respectivamente. Os principais determinantes da estrutura da cromatina são as proteínas histonas. O DNA se envolve ao redor das histonas, formando nucleossomos, que compactam o genoma e ainda permitem o acesso a processos ativos, como transcrição, replicação e reparo de DNA. Cada partícula do núcleo do nucleossomo compreende ~ 147 bp de DNA envolto em 1,7 voltas em torno de um octâmero de proteína, formado por duas moléculas, cada uma das quatro histonas altamente conservadas H2A, H2B, H3 e H4. Os domínios das histonas se dobram em pares antiparalelos: H3 com H4 e H2A com H2B12. Todas as quatro histonas também têm 'caudas' que se estendem para fora do centro do nucleossomo e estão sujeitas a diversas modificações pós-traducionais que são importantes na regulação do gene e compactação da cromatina. Essas modificações covalentes, principalmente nas caudas N-terminais das histonas, incluem metilação, acetilação, fosforilação, ubiquitilação, sumoilação e ADP-ribosilação. Os resíduos de lisina nas caudas das histonas são um substrato comum para modificações, onde a acetilação envolve pequenos grupos químicos, enquanto a ubiquitilação e a sumoilação adicionam dois terços do tamanho das próprias proteínas histonas. Outro grau de complexidade é que a metilação pode ocorrer várias vezes (mono, di ou trimetilação)13. Alguns dos resultados funcionais dessas modificações são evidentes, como a ativação da acetilação, ao passo que a sumoilação parece reprimir, e essas duas modificações podem interferir mutuamente. Em contraste, a metilação e ubiquitilação podem ter efeitos variáveis, dependendo dos resíduos e do contexto precisos. Por exemplo, a trimetilação da lisina 4 na histona H3 (H3K4me3) ocorre nas extremidades 5' dos genes que são induzidos, enquanto a H3K9me3 ocorre na heterocromatina pericentromérica compacta, que é transcricionalmente inativa. Vários modelos, como o modelo de neutralização de carga e o código das histonas14, bem como o de via de sinalização são importantes nesse processo. Novamente, a desregulação dos mecanismos associados ao chamado "código das histonas" já foi descrita em diferentes tipos de câncer.
RNAs NÃO CODIFICADORES E CÂNCER
Os RNAs não codificadores de proteínas (ncRNAs) dominam a produção transcricional em mamíferos e outros organismos complexos. Na realidade, o genoma eucariótico, ao invés de ser visto como uma ilha de genes codificadores de proteínas em um mar em expansão de DNA "lixo" evolucionário, pode ser considerado como uma máquina de RNA15,16. Existem muitos processos em que os ncRNAs (RNAs que não são traduzidos em proteínas) têm uma função específica no epigenoma. Além disso, a expressão desregulada de vários desses RNAs está associada à tumorigênese e ao desenvolvimento do câncer em diferentes tipos da doença16.
Os ncRNAs podem ser classificados de acordo com seu tamanho como pequenos RNAs não codificadores (< 200 bp) ou longos RNAs não codificadores (lncRNAs) (> 200 bp)16. Os microRNAs estão envolvidos em uma série de funções celulares, como regulação da proliferação, diferenciação celular e apoptose em células saudáveis e cancerosas. Aparentemente, ocorre uma rede de interações entre as vias lncRNA e microRNA (miRNA, um tipo de pequeno RNA não codificador)17. Os lncRNAs podem atuar como esponjas moleculares, capturando os miRNAs, evitando a ação destes18. Os ncRNAs também podem atuar em competição com RNAs endógenos, promovendo a proliferação celular no câncer de próstata19.
Portanto, acredita-se que, em um futuro próximo, os ncRNAs poderão ser explorados para identificar novos biomarcadores de câncer para a previsão e progressão da doença, e também para desenvolver novas alternativas terapêuticas aos métodos convencionais usados atualmente em oncologia. Todas essas mudanças epigenéticas são relevantes para manter a estabilidade do chamado epigenoma nas células normais, e os problemas nesses processos estão diretamente ligados ao desenvolvimento e progressão do câncer.
REFERÊNCIAS
1 Bird A. Perceptions of epigenetics. Nature. 2007 May;447(7143):396-8. Doi: 10.1038/nature05913 [Link] [ Links ]
2 Hamm CA, Costa FF. Epigenomes as therapeutic targets. Pharmacol Ther. 2015 Jul;151:72-86. Doi: 10.1016/j.pharmthera.2015.03.003 [Link] [ Links ]
3 Feinberg AP. Epigenetics at the epicenter of modern medicine. JAMA. 2008 Mar;299(11):1345-50. Doi: 10.1001/jama.299.11.1345 [Link] [ Links ]
4 Hamm CA, Costa FF. The impact of epigenomics on future drug design and new therapies. Drug Discov Today. 2011 Jul;16(13-14):626-35. Doi: 10.1016/j.drudis.2011.04.007 [Link] [ Links ]
5 Hotchkiss RD. The quantitative separation of purines, pyrimidines, and nucleosides by paper chromatography. J Biol Chem. 1948 Aug;175(1): 315-32. [Link] [ Links ]
6 Sinsheimer RL. The action of pancreatic deoxyribonuclease. II. Isomeric dinucleotides. J Biol Chem. 1955 Aug;215(2):579-83. [Link] [ Links ]
7 Ehrlich M, Wang RY. 5-Methylcytosine in eukaryotic DNA. Science. 1981 Jun;212(4501):1350-7. Doi: 10.1126/science.6262918 [Link] [ Links ]
8 Costa FF. Epigenomics in cancer management. Cancer Manag Res. 2010 Oct;2:255-65. Doi: 10.2147/CMR.S7280 [Link] [ Links ]
9 Kriaucionis S, Heintz N. The nuclear DNA base 5-hydroxymethylcytosine is present in Purkinje neurons and the brain. Science. 2009 May;324(5929):929-30. Doi: 10.1126/science.1169786 [Link] [ Links ]
10 Penn NW, Suwalski R, O'Riley C, Bojanowski K, Yura R. The presence of 5-hydroxymethylcytosine in animal deoxyribonucleic acid. Biochem J. 1972 Feb;126(4):781-90. Doi: 10.1042/bj1260781 [Link] [ Links ]
11 Bird A. DNA methylation patterns and epigenetic memory. Genes Dev. 2002 Jan;16(1):6-21. Doi: 10.1101/gad.947102 [Link] [ Links ]
12 Luger K, Mäder AW, Richmond RK, Sargent DF, Richmond TJ. Crystal structure of the nucleosome core particle at 2.8 A resolution. Nature. 1997 Sep;389(6648):251-60. Doi: 10.1038/38444 [Link] [ Links ]
13 Berger SL. The complex language of chromatin regulation during transcription. Nature. 2007 May;447(7143):407-12. Doi: 10.1038/nature05915 [Link] [ Links ]
14 Strahl BD, Allis CD. The language of covalent histone modifications. Nature. 2000 Jan;403(6765):41-5. Doi: 10.1038/47412 [Link] [ Links ]
15 Costa FF. Non-coding RNAs, epigenetics and complexity. Gene. 2008 Feb;410(1):9-17. Doi: 10.1016/j.gene.2007.12.008 [Link] [ Links ]
16 Costa FF. Non-coding RNAs: meet thy masters. Bioessays. 2010 Jul;32(7):599-608. Doi: 10.1002/bies.200900112 [Link] [ Links ]
17 Dykes IM, Emanueli C. Transcriptional and post-transcriptional gene regulation by long non-coding RNA. Genomics Proteomics Bioinformatics. 2017 Jun;15(3):177-86. Doi: 10.1016/j.gpb.2016.12.005 [Link] [ Links ]
18 Zhu M, Liu J, Xiao J, Yang L, Cai M, Shen H, et al. Lnc-mg is long non-coding RNA that promotes myogenesis. Nat Commun. 2017 Mar;8:14718. Doi: 10.1038/ncomms14718 [Link] [ Links ]
19 Zhang S, Dong X, Ji T, Chen G, Shan L. Long non-coding RNA UCA1 promotes cell progression by acting as a competing endogenous RNA of ATF2 in prostate cancer. Am J Transl Res. 2017 Feb;9(2):366-75. [Link] [ Links ]
Se refere ao doi: 10.5123/S2176-62232017000400006, publicado originalmente em inglês. Traduzido por: Patrícia Campelo Haick
*Artigo de opinião de Palestrante para o II Encontro Científico Internacional do Instituto Evandro Chagas, realizado de 25 a 27 de outubro de 2017, em Ananindeua, Pará, Brasil. Todos os artigos desta modalidade foram analisados pela Comissão Científica do Evento e, posteriormente, pelos Editores da RPAS
Recebido: 09 de Outubro de 2017; Aceito: 11 de Outubro de 2017










 texto em
texto em