Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista Pan-Amazônica de Saúde
versão impressa ISSN 2176-6215versão On-line ISSN 2176-6223
Rev Pan-Amaz Saude vol.9 no.4 Ananindeua dez. 2018
http://dx.doi.org/10.5123/s2176-62232018000400003
ARTIGO ORIGINAL
Perfis de infecção do vírus Mayaro e do vírus Chikungunya em linhagens de células de mamíferos e mosquitos
1Instituto Evandro Chagas/SVS/MS, Seção de Arbovirologia e Febres Hemorrágicas, Ananindeua, Pará, Brasil
OBJETIVO:
Traçar o perfil infeccioso do vírus Mayaro (MAYV) e do vírus Chikungunya (CHIKV) em quatro diferentes linhagens de células.
MATERIAIS E MÉTODOS:
Os isolados desses vírus foram utilizados para infectar culturas das seguintes linhagens celulares - C6/36, VERO, BHK-21 e LLC-MK2 - sob a multiplicidade de infecção de 1 PFU/célula, as quais foram monitoradas por até 96 h pós-infecção (hpi) por microscopia de contraste de fase para registrar o efeito citopático (CPE); e, em seguida, foram submetidos a ensaios de imunofluorescência indireta (iIF) e reação em cadeia da polimerase de transcrição reversa quantitativa (qRT-PCR) para detecção de antígenos virais e determinação da carga de RNA, respectivamente.
RESULTADOS:
Para ambos os vírus, as células VERO e BHK-21 apresentaram CPE de 24 hpi, as LLC-MK2 apresentaram CPE a partir de 48 hpi e as C6/36 não apresentaram qualquer CPE. Nos ensaios de iIF, todas as células já se tornaram positivas a 6 hpi, com exceção das células LLC-MK2 e BHK-21 infectadas com CHIKV. Finalmente, os ensaios qRT-PCR mostraram que, em geral, as células infectadas com MAYV apresentaram aumento da carga de RNA, embora as células LLC-MK2 infectadas pelo CHIKV tenham apresentado a maior carga de RNA.
CONCLUSÃO:
Ao avaliar o comportamento desses arbovírus em diferentes tipos de culturas de células, incluindo alguns não comumente utilizados na rotina do diagnóstico laboratorial, o presente estudo fornece não apenas uma melhor compreensão da cinética de replicação de MAYV e CHIKV, mas também oportunidades para a otimização de seu diagnóstico em amostras biológicas.
Palavras-chave: Arbovírus; Alphavirus; Vírus Mayaro; Vírus Chikungunya; Linhagem Celular
INTRODUÇÃO
O vírus Mayaro (MAYV) e o vírus Chikungunya (CHIKV) são arbovírus pertencentes à família Togaviridae, gênero Alphavirus. O MAYV foi isolado pela primeira vez em 1954, em amostras de trabalhadores rurais em Trinidad e Tobago e, posteriormente isolado na área do rio Guamá, no estado do Pará, Brasil. O CHIKV foi isolado pela primeira vez na Tanzânia, em 1952, durante um surto atribuído ao vírus da Dengue (DENV)1,2,3,4,5.
Surtos e epidemias de febre de Mayaro foram registrados no Brasil, Bolívia e Peru. No norte do Brasil, existem relatos de vários surtos ocorridos em cidades do estado do Pará, como Belterra (1978), Conceição do Araguaia (1981) e Benevides (1991) e no estado do Amazonas, em Manaus (2007-2008)6. No período de 2014 a 2016, foram notificados e distribuídos 343 casos suspeitos humanos de febre de Mayaro em 11 estados brasileiros, destacando-se o estado de Goiás que apresentou a maior frequência7. Por outro lado, o primeiro caso autóctone de febre Chikungunya ocorreu nas Américas em 2013, na região do Caribe, chegando ao Brasil em 2014 e causando dezenas de milhares de casos8,9. De 2014 a 2017, 505.723 casos humanos suspeitos de febre de Chikungunya foram relatados no Brasil9,10,11,12,13.
Nos seres humanos, a infecção pelo MAYV é esporádica, afetando principalmente pessoas provenientes de florestas tropicais, onde seu principal vetor é o mosquito Haemagogus janthinomys. Em relação ao CHIKV, um ciclo selvagem e periurbano da doença foi descrito na África, transmitido pelo Aedes aegypti, e sua disseminação para o Oceano Índico, Ásia e Europa estava relacionada à presença do vetor Aedes albopictus3,4,5,7,14.
A condição clínica do paciente afetado pela febre de Mayaro é de início repentino, caracterizada por febre atingindo 39 °C, dor de cabeça, mialgia, erupção cutânea (principalmente no peito, costas, braços e pernas), dor intensa nas articulações dos pulsos, tornozelos, dedos, joelhos e cotovelos, bem como na coluna lombar e sacral. Da mesma forma, a febre de Chikungunya é caracterizada por poliartrite/artralgia simétrica nos pulsos, tornozelos e cotovelos, e pode regredir após 10 dias de infecção ou progredir para cronicidade4,5,7,14,15,16,17.
O método mais específico para o diagnóstico de infecções por arbovírus é a reação em cadeia da polimerase com transcrição reversa (RT-PCR), mas o isolamento do vírus seguido de imunofluorescência (IF) é considerado o método padrão-ouro para detectar o agente infeccioso. Tentativas de isolamento de vírus podem ser realizadas por inoculação em culturas de células de camundongos albinos suíços recém-nascidos (2 a 3 dias) e, eventualmente, também em mosquitos1,18.
As culturas de células de artrópodes têm sido cada vez mais utilizadas, pois demonstram sensibilidade ao isolamento de vírus semelhante a camundongos lactantes. Entre as linhagens celulares de artrópodes mais utilizadas estão as células C6/36 de Aedes albopictus, células AP-61 de Aedes pseudoscutellaris e células TRA-284 de Toxorhynchites amboinensis1,16,19. As culturas de células vertebradas também são amplamente utilizadas e incluem células VERO do rim de macaco verde africano (Chlorocebus aethiops), células BHK-21 do rim de hamster bebê (Mesocricetus auratus) e células LLC-MK2 do rim de macaco rhesus (Macaca mulatta)16,20.
Sobre muitos arbovírus, pouco ou nada se sabe sobre suas características ecopidemiológicas e moleculares e sua capacidade de infectar várias linhagens celulares disponíveis atualmente. Assim, este trabalho teve como objetivo traçar o perfil de infecção de MAYV e CHIKV em quatro linhagens celulares, analisando a suscetibilidade e a permissividade das células BHK-21 e LLC-MK2 às infecções por alphavirus em comparação às células C6/36 e VERO, que são mais frequentemente utilizadas em laboratórios específicos. Além disso, foram realizados estudos da cinética de replicação viral para acompanhar a progressão da infecção pelo vírus nessas linhagens celulares, avaliando o efeito citopático (CPE), detecção de antígeno do vírus e a progênie de carga RNA.
MATERIAIS E MÉTODOS
AMOSTRAS DE VÍRUS
Os isolados de vírus brasileiros utilizados neste estudo foram fornecidos pela coleta de arbovírus da Seção de Arbovirologia e Febre Hemorrágica (SAARB) do Instituto Evandro Chagas (IEC). A cepa MAYV (BeAr20290; passagem 2; genótipo L; número de acesso genbank KY618127) foi isolada de um artrópode hematófago (Haemagogus sp.) em 1960 e CHIKV (BeH803609; passagem 2; genótipo asiático; número de acesso genbank KP164571) foi isolada a partir do sangue de um ser humano doente em 2014. Os estoques de CHIKV e MAYV utilizados foram originários de fluidos de cultura de células e cérebro de camundongos, respectivamente, mantidos em freezer a -70 ºC.
CULTURA DE CÉLULAS
As células utilizadas neste estudo foram cultivadas de acordo com as particularidades de cada linhagem. As células C6/36 (ATCC, EUA) foram cultivadas a 28 °C em meio Leibovitz L-15 com L-glutamina (Gibco, EUA) suplementada com soro bovino fetal (FBS) a 5% (FBS) (Gibco, EUA), fosfato de triptose a 3% (Himedia, Índia), 10 mL/L de aminoácidos não essenciais (Baktron Microbiology, Brasil) e 1% de solução antibiótica (10.000 U/mL de penicilina e 10.000 µg/mL de estreptomicina) (Gibco, EUA)16,21. As células VERO (ATCC) foram cultivadas a 37 °C e 5% de CO2 no meio 199 (Gibco, EUA) suplementado com 2,2 g/L de bicarbonato de sódio (Sigma, EUA), 5% de SFB e 1% de solução antibiótica20,22. As células BHK-21 (ATCC) foram cultivadas a 37 °C e 5% de CO2 em MEM23 e as células LLC-MK2 (ATCC) foram cultivadas a 37 °C e 5% de CO2 em DMEM24, ambas suplementadas com 3,7 g/L e 2,2 g/L de bicarbonato de sódio, respectivamente, 5% de FBS e 1% de solução antibiótica.
PREPARAÇÃO E TITULAÇÃO DE ESTOQUES DE VÍRUS
Os estoques de vírus foram preparados, inoculando 200 μL dos isolados de vírus em células VERO cultivadas em frascos de cultura de 25 cm2. A infecção foi realizada pelo método de adsorção viral. Após a infecção, as células eram visualizadas diariamente por microscopia óptica (LM) para a observação do CPE. O sobrenadante da cultura celular foi obtido de culturas com CPE e fracionado em alíquotas de 500 μL e armazenado a -70 °C.
Os títulos infecciosos dos estoques de vírus foram determinados pelo ensaio de placa25 usando diluições seriadas de 10 vezes (101 a 107) de cada amostra preparada no meio 199; 200 μL de cada diluição foram inoculados em placas de 6 poços e, após 1 h, foram adicionados 3 mL de meio de cultura semissólido (1,6% de carboximetilcelulose no meio 199) suplementado com 2% de FBS e 1% de solução antibiótica. As placas foram incubadas a 37 °C e 5% de CO2 por 48 h e, simultaneamente, fixadas e coradas por 3 h em solução contendo 1% de cristal violeta, 30% de etanol e 20% de formalina em solução salina tamponada com fosfato (PBS). As placas foram contadas e os títulos de vírus foram expressos como unidades formadoras de placas (PFU)/mL.
INFECÇÃO EXPERIMENTAL DAS LINHAS CELULARES
Para padronizar a dose infectante, foi utilizada a multiplicidade de infecção (MOI) de 1 PFU/célula para todas as linhagens celulares e preparadas placas de 6 poços contando as células de acordo com o título de vírus obtido em C6/36, VERO, Células BHK-21 ou LLC-MK2; e então cada placa de poço foi infectada com MAYV ou CHIKV por adsorção de 1 h para realizar as análises da cinética de replicação de vírus nas 6, 12, 24, 48, 72 e 96 h pós-infecção (hpi). Um controle negativo foi utilizado para cada linha celular. Todas as infecções foram realizadas de acordo com os padrões de nível três de biossegurança.
CONTRASTE DE FASE LM (PCLM)
As células foram observadas para CPE por pcLM. As imagens de contraste de fase foram adquiridas em um microscópio Axiovert 25 usando lente objetiva 10x/0,25 CP-Achromat (Zeiss, Alemanha). Os conjuntos de imagens foram processados usando o software ImageJ 1.48 (NIH, EUA).
ENSAIO DE IMUNOFLUORESCÊNCIA INDIRETA (IIF)
As células foram colhidas por raspagem e submetidas a IF seguindo um protocolo adaptado de Gubler et al.26. Foram utilizados, como anticorpo primário, anticorpos policlonais anti-alfavírus produzidos em camundongos e, como anticorpos secundários, anticorpos anti-camundongos conjugados com isotiocianato de fluoresceína (Cappel). As imagens IF foram adquiridas em um microscópio BX51 usando uma câmera com lente objetiva UPlanFL N 20x/0.5 e filtros WB e U-25ND (Olympus, Japão). Os conjuntos de imagens foram processados usando o software ImageJ 1.48 (NIH, EUA). Uma quantidade de classificação de pontuação positiva baseada em células estabelecida por inspeção visual foi usada para a expressão dos níveis de intensidade de fluorescência das imagens de IF da seguinte forma: (1+ = 1 a 25% das células positivas, 2+ = aproximadamente 50% das células positivas, 3+ = aproximadamente 75% das células positivas e 4+ = aproximadamente 100% das células positivas).
RT-PCR QUANTITATIVO (QRT-PCR)
Os sobrenadantes das culturas celulares foram colhidos por aspiração e submetidos a qRT-PCR seguindo os protocolos padronizados por Celestino Júnior27 e Lanciotti et al.28. Os sobrenadantes das culturas de células infectadas foram submetidos à extração de RNA usando o Maxwell 16 Total RNA Purification kit no instrumento Maxwell 16 (Promega, EUA). Para a execução da técnica de qRT-PCR, foi utilizado o kit qRT-PCR de uma etapa SuperScript III Platinum com sondas TaqMan (Invitrogen, EUA) no sistema de PCR em tempo real ViiA 7 (Applied Biosystems, EUA). Os gráficos exponenciais gerados pelas amplificações foram analisados no software Prism 6.0 (GraphPad, EUA).
RESULTADOS
PROGRESSÃO DO CPE E TITULAÇÃO DO ESTOQUE DE VÍRUS
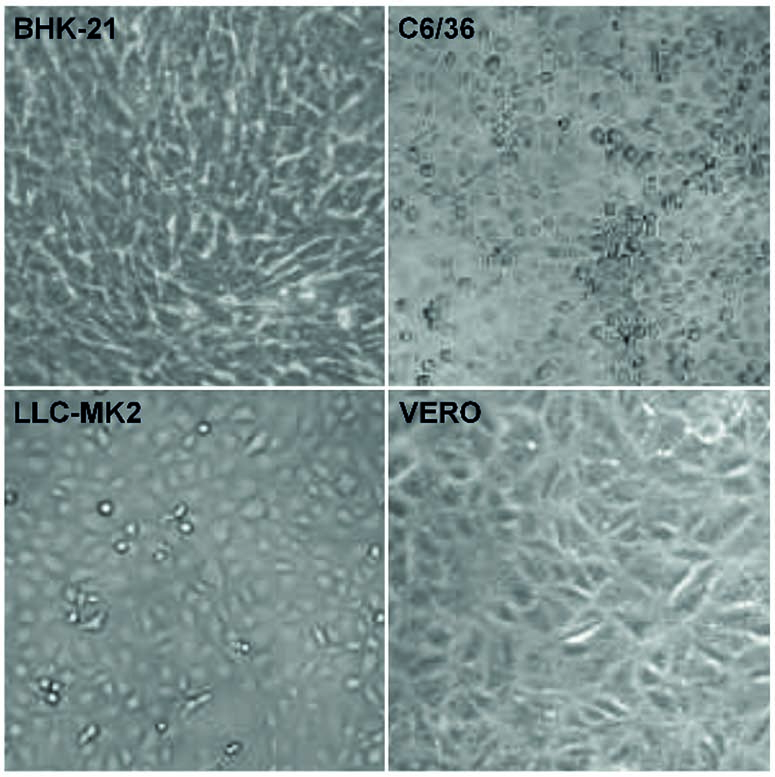
O título CHIKV foi de 5,4 x 107 PFU/mL, enquanto o título MAYV foi de 3,2 x 108 PFU/mL. Nas células BHK-21 (Figura 1A) infectadas com CHIKV, a manifestação de CPE ocorreu a partir de 24 hpi; aos 48 hpi, a presença de CPE era mais evidente; e, a partir de 72 hpi, a monocamada foi totalmente destruída. O mesmo padrão foi observado no MAYV nessas células, porém o CPE foi detectado precocemente com apenas 12 hpi. Nas células C6/36 infectadas por vírus (Figura 1B), não foi observado CPE. As células LLC-MK2 infectadas por vírus (Figura 1C) apresentaram CPE relativamente mais tarde, de 48 hpi em culturas MAYV e 72 hpi em células infectadas por CHIKV, que progrediram com a destruição da monocamada a 96 hpi. As células VERO infectadas por vírus (Figura 1D) mostraram manifestação de CPE de 24 hpi com efeito mais proeminente nas culturas de MAYV neste momento; e uma rápida progressão para ambos os vírus em direção à destruição da monocamada nos pontos de tempo subsequentes.

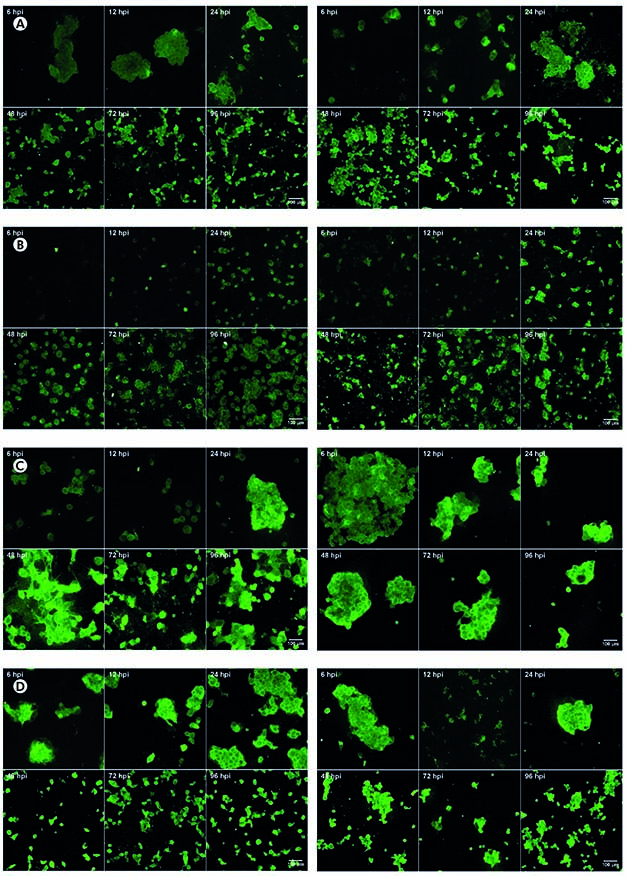
As células BHK-21 (A), C6/36 (B), LLC-MK2 (C), e VERO (D) foram infectadas com CHIKV (painéis a esquerda) ou MAYV (painéis a direita) sob o MOI de 1 PFU/célula para os tempos indicados após a infecção, quando suas imagens de luz transmitidas foram registradas para medir o CPE pelo LM, usando um anel de contraste de fase. As imagens representam três campos visuais selecionados aleatoriamente para cada condição.
Figura 1 - Progressão da CPE em células infectadas por alphavirus
Nas células BHK-21, VERO e LLC-MK2 infectadas com MAYV e CHIKV, foi observada a ocorrência de tipo lítico de CPE, causando a pilhagem das células mortas, bem como a sua refração até a completa destruição da monocamada. Não foram observadas diferenças morfológicas entre as células infectadas com MAYV e CHIKV. Os controles negativos de cada linha de células são apresentados na figura 2.
As células BHK-21, C6/36, LLC-MK2, e VERO foram cultivadas por 48 h, quando suas imagens de luz transmitidas foram registradas para medir o CPE pelo LM, usando um anel de contraste de fase. As imagens representam três campos visuais selecionados aleatoriamente para cada condição.
Figura 2 - O CPE em células não infectadas
PROGRESSÃO DA DETECÇÃO DE ANTÍGENOS POR VÍRUS
Em relação ao IF das células BHK-21 infectadas com CHIKV (Figura 3A, painéis esquerdos), a positividade de 1+ foi observada a 12 hpi e 2+ a 24 hpi; às 48 hpi, a fluorescência progrediu para a positividade de 3+; e, nos momentos subsequentes, houve estabilização da positividade em 3+. Por outro lado, as células BHK-21 infectadas com MAYV (Figura 3A, painéis à direita) já mostravam a positividade de 1+ a 6 hpi; às 12 e 24 hpi, houve positividade de 2+; às 48 hpi, a fluorescência progrediu para a positividade de 3+; a partir de 72 hpi, houve positividade de 4+.
As células C6/36 infectadas com CHIKV (Figura 3B, painéis à esquerda) mostraram apenas uma célula positiva a 6 hpi no FII, com progressão nas horas subsequentes: a 12 hpi com a positividade de 2+ e a 24, 48, 72, e 96 hpi com positividade de 3+. As células C6/36 infectadas com MAYV (Figura 3B, painéis à direita) mostraram a positividade de 1+ a 6 hpi e 2+ a 12 hpi, tendo uma progressão diretamente para 3+ a 24 hpi; a partir de 48 hpi, houve positividade de 4+.
Em relação às células LLC-MK2 infectadas com CHIKV (Figura 3C, painéis à esquerda), a positividade de 1+ foi observada a 24 hpi, aumentando diretamente para 3+ de 48 hpi a 96 hpi. Em contraste, as células LLC-MK2 infectadas com MAYV (Figura 3C, painéis à direita) já haviam mostrado a positividade de 1+ a 6 hpi e 12 hpi; às 24 hpi e 48 hpi foi observada positividade de 2+ e 3+, respectivamente; houve positividade de 4+ a partir de 72 hpi.
As células VERO infectadas com CHIKV (Figura 3D, painéis à esquerda) ou MAYV (Figura 3D, painéis à direita) já haviam mostrado positividades semelhantes no ensaio iIF, com 2+ a 6 hpi e 12 hpi, crescendo posteriormente para 3+ a 24 hpi. Ambos os vírus resultaram na mesma positividade de 4+ às 48 hpi, com a estabilização da positividade em 4+ nas horas subsequentes.
As células BHK-21 (A), C6/36 (B), LLC-MK2 (C), and VERO (D) foram infectadas com CHIKV (painéis à esquerda) ou MAYV (painéis à direita) sob o MOI de 1 PFU/célula para os tempos indicados após a infecção, quando foram fixados para medir a detecção de antígenos por IF, usando anticorpos primários específicos e anticorpos secundários marcados com FITC. As imagens representam três campos visuais selecionados aleatoriamente para cada condição.
Figura 3 - Progressão da detecção de antígeno do vírus em células infectadas por alphavirus
Todas as análises de cultura de células mostraram maior intensidade de fluorescência nas células infectadas pelo MAYV.
Os controles negativos do IF de cada linha de células são apresentados na figura 4.
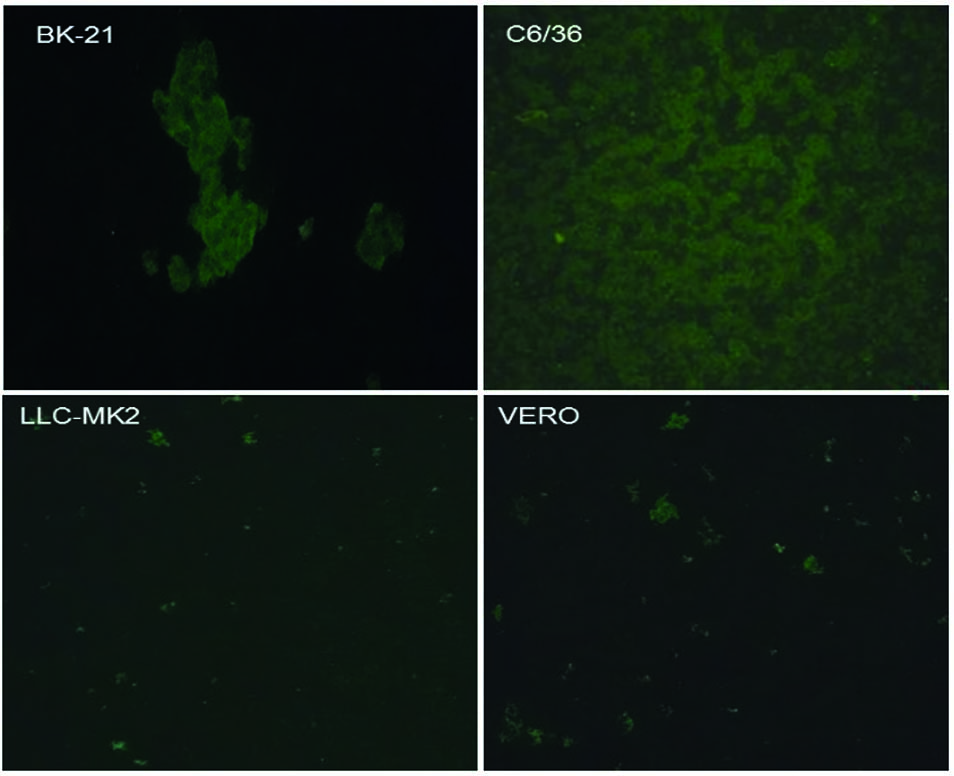
As células BHK-21, C6/36, LLC-MK2, e VERO foram cultivadas por 48 h, quando fixadas para medir a detecção de antígeno por IF usando anticorpos primários antialphavirus e anticorpos secundários marcados com FITC. As imagens representam três campos visuais selecionados aleatoriamente para cada condição.
Figura 4 - A detecção do antígeno do vírus em células não infectadas
PROGRESSÃO DA CARGA DE RNA NO SOBRENADANTE
Conforme avaliado por qRT-PCR no sobrenadante de culturas celulares, em células BHK-21 infectadas com CHIKV (Figura 5A, barras pretas), o RNA viral foi detectado a 6 hpi (3,60 x 103 cópias/μL), com aumento nesse valor nos tempos subsequentes de 12 hpi (8,68 x 103 cópias/μL), 24 hpi (2,60 x 104 cópias/μL), 48 hpi (3,31 x 105 cópias/μL), e 72 hpi (3,81 x 105 cópias/μL). Eventualmente, houve uma queda na carga viral em 96 hpi (2,84 x 105 cópias/μL). Por outro lado, as células BHK-21 infectadas com MAYV (Figura 5A, barras cinza) apresentaram carga viral crescente - 4,87 x 102 cópias/μL a 6 hpi, 6,85 x 103 cópias/μL a 12 hpi, 9,82 x 104 cópias/μL a 24 hpi, 6,63 x 105 cópias/μL a 48 hpi, 6,87 x 105 cópias/μL a 72 hpi e 1,28 x 106 cópias/μL a 96 hpi.
As células C6/36 infectadas com CHIKV (Figura 5B, barras pretas) apresentaram cargas de RNA de 2,76 x 102 cópias/μL a 6 hpi, 9,36 x 102 cópias/μL a 12 hpi, 1,27 x 103 cópias/μL a 24 hpi, 1,14 x 104 cópias/μL a 48 hpi, 3,60 x 105 cópias/μL a 72 hpi e 8,76 x 105 cópias/μL a 96 hpi. As células C6/36 infectadas pelo MAYV (Figura 5B, barras cinza) mostraram 3,36 x 102 cópias/μL a 6 hpi, 1,08 x 103 cópias/μL a 12 hpi, 2,25 x 103 cópias/μL a 24 hpi, 2,14 x 104 cópias/μL a 48 hpi, 4,42 x 105 cópias/μL a 72 hpi e 6,86 x 105 cópias/μL a 96 hpi.
As células LLC-MK2 infectadas com CHIKV (Figura 5C, barras pretas) mostraram um aumento na carga de RNA ao longo do tempo: 2,62 x 102 cópias/μL a 6 hpi, 6,45 x 105 cópias/μL a 12 hpi, 4,54 x 106 cópias/μL a 24 hpi, 8,89 x 106 cópias/μL a 48 hpi, 9,16 x 106 cópias/μL a 72 hpi e 1,26 x 107 cópias/μL a 96 hpi. As células LLC-MK2 infectadas com MAYV (Figura 5C, barras cinza) também mostraram carga viral progressiva, exibindo 2,00 x 102 cópias/μL a 6 hpi, 1,84 x 103 cópias/μL a 12 hpi, 5,31 x 105 cópias/μL a 24 hpi, 8,74 x 105 cópias/μL a 48 hpi, 9,97 x 105 cópias/μL a 72 hpi e 1,97 x 106 cópias/μL a 96 hpi.
As células VERO infectadas com CHIKV (Figura 5D, barras pretas) apresentaram cargas de RNA de 3,98 x 102 cópias/μL, 7,00 x 103 cópias/μL, 1,28 x 104 cópias/μL, 3,98 x 104 cópias/μL, 2,48 x 105 cópias/μL e 2,55 x 105 cópias/μL a 6 hpi, 12 hpi, 24 hpi, 48 hpi, 72 hpi e 96 hpi, respectivamente. As células VERO infectadas com MAYV (Figura 5D, barras pretas) apresentaram as seguintes cargas de RNA: 1,82 x 102 cópias/μL a 6 hpi, 9,03 x 102 cópias/μL a 12 hpi, 2,24 x 104 cópias/μL a 24 hpi, 6,91 x 104 cópias/μL a 48 hpi, 3,48 x 105 cópias/μL a 72 hpi e 4,88 x 105 cópias/μL a 96 hpi.
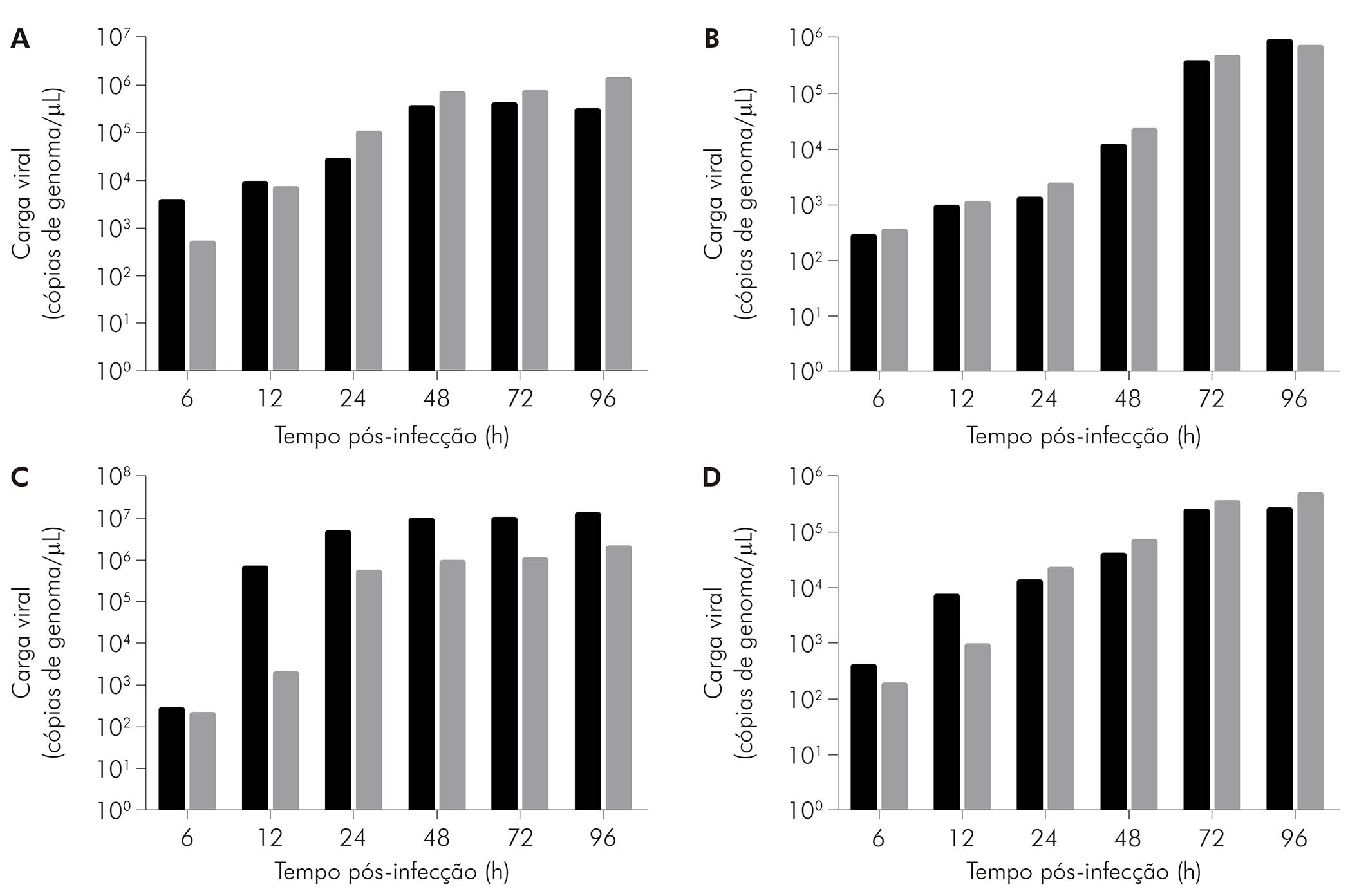
As células BHK-21 (A), C6/36 (B), LLC-MK2 (C), e VERO (D) foram infectadas com CHIKV (barras pretas) ou MAYV (barras cinzas) sob o MOI de 1 PFU/célula para os tempos indicados após a infecção, quando seus sobrenadantes foram colhidos para medir a carga de RNA por RT-qPCR, usando conjuntos específicos de iniciador/sonda. Os dados foram obtidos de três experiências e plotados como média ± desvio padrão para cada condição.
Figura 5 - A progressão da carga de RNA no sobrenadante de células infectadas por alphavirus
DISCUSSÃO
Este estudo teve como objetivo comparar o perfil de duas infecções por alphavirus de importância em saúde pública na região amazônica brasileira: o MAYV, detectado pela primeira vez no Brasil em 195529, e o CHIKV detectado pela primeira vez no Brasil em 201430. As principais linhagens celulares usadas para o diagnóstico laboratorial desses arbovírus são C6/36 e VERO, que certamente representam bons sistemas de isolamento de vírus. No entanto, poucos estudos tentaram analisar a suscetibilidade de outras linhagens celulares, como BHK-21 e LLC-MK2, em comparação com os mencionados anteriormente, além de analisar o início da detecção por meio dos testes IF e qRT-PCR.
Este estudo demonstra que, para ambos os alphavirus, as células VERO e BHK-21 apresentaram CPE anterior, o que já era observado a 12 hpi em ambas as células, quando comparado às células C6/36 e LLC-MK2. As células C6/36 não apresentaram CPE, como já era esperado, já que a maioria das infecções por arbovírus não levou à morte nessa linhagem celular, e as células LLC-MK2 começaram a apresentar CPE a partir de 48 hpi. Vale ressaltar que o CPE observado em todas as culturas que mostraram ser do tipo lítico, com destruição completa da monocamada e grande quantidade de células mortas arredondadas.
Ao comparar as infecções por MAYV e CHIKV, observou-se um CPE mais pronunciado em culturas de células infectadas com MAYV (VERO, BHK-21 e LLC-MK2), a partir de 12 hpi. Nas culturas de células infectadas com CHIKV, o aparecimento de CPE às 24 hpi foi um pouco mais suave. No entanto, a partir de 48 hpi em geral, as culturas celulares infectadas por um ou outro vírus apresentaram um padrão semelhante de CPE, exceto as células LLC-MK2, que ainda continuavam apresentando um CPE um pouco mais avançado quando infectadas com MAYV.
Em relação aos resultados da detecção de vírus por IF para CHIKV, as células VERO apresentaram a positividade mais avançada (a 6 hpi), seguida pelas células C6/36, indicando que para esse vírus essas culturas, muito usadas na rotina laboratorial, são melhores para o início detecção de vírus por iIF. Quando as outras culturas celulares foram analisadas, observou-se que, para fins de diagnóstico, para obter positividade em poucos dias, a cultura das células BHK-21 (2+ positividade a 12 hpi) era melhor que a das células LLC-MK2 (1 + positividade a 12 hpi).
Por outro lado, em relação ao MAYV, o teste de IF mostrou uma evolução da positividade até atingir 4+ em todas as linhagens celulares estudadas, mencionando que a maioria dessas culturas celulares apresentava padrão de positividade semelhante, atingindo a positividade de 4+ já a 48 hpi. Assim, pode-se sugerir que qualquer uma das linhagens celulares estudadas possa ser utilizada para fins de diagnóstico, visando à detecção precoce do MAYV, e que as células LLC-MK2 e BHK-21 são boas alternativas à falta de células C6/36 ou VERO em laboratório.
Em relação às cargas de RNA nos sobrenadantes da cultura de células, comparando células BHK-21 infectadas com MAYV ou CHIKV, foram obtidos valores mais altos para MAYV. Tal resultado corrobora os dados obtidos pelo IF, nos quais o MAYV mostrou a positividade de 4+ e o CHIKV, a positividade de 3+ nos últimos momentos estudados. Nas células LLC-MK2, a carga de RNA foi maior no CHIKV do que no MAYV, essa foi a linhagem celular que apresentou o maior título entre todas as culturas celulares analisadas, apontando que as células que não são frequentemente utilizadas na rotina laboratorial apresentam bons resultados na detecção de vírus. Os resultados de qRT-PCR para células LLC-MK2, quando comparados com os resultados de IF, podem parecer inconsistentes, observa-se que o ensaio de qRT-PCR é mais específico que o ensaio de iIF, pois o primeiro é quantitativo e o último é semiquantitativo e depende da especificidade dos anticorpos utilizados.
Quanto às cargas de RNA da cultura de células C6/36, o CHIKV atingiu um valor mais alto no último momento analisado (96 hpi); no entanto, nos demais momentos estudados, o MAYV apresentou valores mais altos. Esses resultados corroboram os do teste de IF em relação à maior positividade para o MAYV às 48 e 72 h, quando comparados ao CHIKV nos mesmos momentos. Por outro lado, as cargas de RNA obtidas com a cultura de células VERO foram mais altas para MAYV durante a maior parte do estudo (24 a 96 hpi), embora os resultados do teste IF indicassem semelhança entre MAYV e CHIKV desde o início detecção de vírus a 6 hpi até o último momento testado. No entanto, o CHIKV apresentou maiores valores de carga de RNA por RT-qPCR em relação ao MAYV.
Simon et al.31 relataram que o diagnóstico de CHIKV foi realizado por RT-PCR e também destacaram o uso de células C6/36 e VERO para o isolamento e estudo desse vírus. Dado o uso quase exclusivo dessas duas linhagens celulares para o diagnóstico de arbovírus, nota-se a relevância deste estudo na tentativa de compreender o comportamento dos alphavirus em outras linhagens celulares que não são comumente usadas na rotina laboratorial, como BHK-21 e principalmente células LLC-MK2.
No estudo de Way et al.32, 20 arbovírus - incluindo nove alphavirus - foram testados em ambos os sistemas de isolamento de vírus (camundongos e cultura de células), e as células VERO e LLC-MK2 apresentaram o maior grau de infecção quando comparadas às células BHK-21. Isso corrobora o presente estudo, mostrando que os maiores valores de carga de RNA foram obtidos em células LLC-MK2.
Segundo Tesh33, a linhagem celular mais utilizada na rotina laboratorial para propagação de arbovírus é o C6/36, devido à sua fácil manutenção e crescimento a 28 °C. Figueiredo34 realizou a propagação de 23 arbovírus em células C6/36, sendo o MAYV um dos arbovírus estudados. Não foi observado CPE nessas células, e o IF para alphavirus mostrou positividade após dois a três dias de infecção. Esses resultados corroboram o presente estudo, que mostrou que não foi observado CPE para os alphavirus estudados em células C6/36; no entanto, neste estudo, a detecção do antígeno do vírus pelo IF foi possível a partir de 6 hpi, o que provavelmente pode ser explicado pelo MOI ser menor nas amostras do estudo de Figueiredo.
Devido à boa adaptação do CHIKV às células BHK-21, essa linhagem celular está sendo usada atualmente para realizar estudos de pequenas mutações nas glicoproteínas do envelope viral que facilitam a adaptação do vírus ao vetor artrópode35.
Lim e Chu36 realizaram uma cinética de replicação do CHIKV em células VERO infectadas sob o MOI de 10, com pontos de tempo de 0, 12, 24, 36 e 48 hpi, e observaram CPE de 36 hpi e maior intensificação a 48 hpi; no mesmo estudo, também foi realizada a quantificação do RNA viral por meio do qRT-PCR, e observou-se a maior carga de RNA às 24 hpi, com 5,0 x 108 PFU/mL. No presente estudo, foi realizada a cinética de replicação com 6, 12, 24, 48, 72 e 96 hpi, com início da CPE em células VERO infectadas com CHIKV já a 24 hpi, o que corrobora o estudo citado36, visto que o CPE começa a se desenvolver a partir de 24 hpi, porém a 48 hpi encontra-se avançado. No entanto, dado o MOI inicial dez vezes menor, a carga de RNA, neste estudo, também foi menor que a do estudo de Lim e Chu, atingindo um pico de 96 hpi com 2,55 x 105 cópias/μL para CHIKV em células VERO.
Lee e Chu37 conseguiram replicar o CHIKV em células C6/36 para estudar várias proteínas que interferem na replicação do vírus em células artrópodes, corroborando o presente estudo, demonstrando que as células C6/36, embora não apresentem CPE, fornecem bons resultados em testes de IF e qRT-PCR.
Em conjunto, esses achados sugerem que as células VERO, BHK-21 e LLC-MK2 são as células de escolha para os ensaios de monitoração e titulação de CPE e formação de placas. Embora as células C6/36 não demonstrem CPE para os dois vírus experimentados em questão, elas apresentam excelentes resultados com detecção precoce, justificando seu amplo uso no diagnóstico laboratorial. No entanto, este trabalho também mostra que outras linhagens celulares menos utilizadas na rotina de diagnóstico, como BHK-21 e especialmente LLC-MK2, permitem excelentes cargas de RNA. Além disso, ao comparar o MAYV com o CHIKV, foi geralmente observado que o MAYV alcançou cargas de RNA mais altas, bem como início precoce da detecção de CPE e IF - provavelmente porque a amostra do MAYV foi isolada em 1960, sofrendo um número maior de passagens do que a cepa CHIKV isolada mais recentemente.
Além disso, estudos como este são importantes para mostrar o melhor momento para colher células infectadas que servirão como fontes de vírus para técnicas como o sequenciamento de nucleotídeos, que requer uma grande quantidade de RNA viral; por exemplo, com a linhagem celular BHK-21, a maior carga de RNA alcançada para CHIKV foi de 72 hpi e, para MAYV, de 96 hpi. Isso indica que existem diferentes perfis de replicação entre vírus em diferentes linhagens celulares.
Por fim, é sabido que muitas células utilizadas na rotina laboratorial já são padronizadas para o diagnóstico de arbovírus, porém é necessário que novos estudos sejam realizados com outras linhagens celulares para analisar sua suscetibilidade e permissividade ao crescente número de arbovírus medicamente importantes para a melhoria do diagnóstico virológico.
AGRADECIMENTOS
Os autores agradecem à equipe do Laboratório de Isolamento Viral em Cultura de Células e Laboratório de Biologia Molecular (SAARB/IEC), especialmente Maissa Maia, Ercilia Gonçalves, Jamilla Pantoja, Maxwell Lima, Eliana Silva e Karla Melo, pela assistência técnica ao presente trabalho
REFERÊNCIAS
1 Azevedo RSS, Martins LC, Rodrigues SG, Travassos da Rosa JFS, Vasconcelos PFC. Arboviroses. In: Farhat CK, Carvalho LHFR, Succi RCM, editores. Infectologia pediátrica. 3. ed. São Paulo: Atheneu; 2007. p. 534-51. [ Links ]
2 Causey OR, Maroja O, Azevedo MC. Epidemia pelo vírus "Mayaro" no estado do Pará. Rev Serv Esp Saude Publica. 1958;10(1):152-4. [ Links ]
3 Donalisio MR, Freitas ARR. Chikungunya no Brasil: um desafio emergente. Rev Bras Epidemiol. 2015 jan-mar;18(1):283-5. Doi: 10.1590/1980-5497201500010022 [Link] [ Links ]
4 Lopes N, Nozawa C, Linhares REC. Características gerais e epidemiologia dos arbovírus emergentes no Brasil. Rev Pan-Amaz Saude. 2014 set;5(3):55-64. Doi: 10.5123/S2176-62232014000300007 [Link] [ Links ]
5 Tauil PL. Condições para a transmissão da febre do vírus chikungunya. Epidemiol Serv Saude. 2014 out-dez;23(4):773-4. Doi: 10.5123/S1679-49742014000400020 [Link] [ Links ]
6 Mourão MPG, Bastos MS, Figueiredo RP, Gimaque JBL, Galusso EL, Kramer VM, et al. Mayaro fever in the city of Manaus, Brazil, 2007-2008. Vector Borne Zoonotic Dis. 2012 Jan;12(1):42-6. Doi: 10.1089/vbz.2011.0669 [Link] [ Links ]
7 Ministério da Saúde (BR). Situação epidemiológica - Dados [Internet]. Brasília (DF): Ministério da Saúde; 2015 nov 25 [atualizado 2016 nov 8; citado 2017 set 11]. Disponível em: Disponível em: http://portalms.saude.gov.br/saude-de-a-z/febre-do-mayaro/20950-situacao%20epidemiologica-dados . [ Links ]
8 Vasconcelos PFC, Azevedo RSS, Rodrigues SG, Martins LC, Chiang JO, Travassos da Rosa APA. Arboviroses. In: Leão RNQ, coordenador. Medicina tropical e infectologia na Amazônia. Belém: Samauma; 2013. Vol. 1; cap. 23; p. 481-503. [ Links ]
9 Ministério da Saúde (BR). Secretaria de Vigilância em Saúde. Monitoramento dos casos de dengue, febre de Chikungunya e febre pelo vírus Zika até a Semana Epidemiológica 5, 2016. Bol Epidemiol. 2016;47(8):1-7. [Link] [ Links ]
10 Ministério da Saúde (BR). Secretaria de Vigilância em Saúde. Monitoramento dos casos de dengue e febre de Chikungunya até a Semana Epidemiológica (SE) 53 de 2014. Bol Epidemiol. 2015;46(3):1-7. [Link] [ Links ]
11 Ministério da Saúde (BR). Secretaria de Vigilância em Saúde. Monitoramento dos casos de dengue, febre de Chikungunya e febre pelo vírus Zika até a Semana Epidemiológica 52, 2015. Bol Epidemiol. 2016;47(3):1-10. [Link] [ Links ]
12 Ministério da Saúde (BR). Secretaria de Vigilância em Saúde. Monitoramento dos casos de dengue, febre de Chikungunya e febre pelo vírus Zika até a Semana Epidemiológica 52, 2016. Bol Epidemiol. 2017;48(3):1-11. [Link] [ Links ]
13 Ministério da Saúde (BR). Secretaria de Vigilância em Saúde. Monitoramento dos casos de dengue, febre de Chikungunya e febre pelo vírus Zika até a Semana Epidemiológica 52, 2017. Bol Epidemiol. 2018;49(2):1-13. [Link] [ Links ]
14 Travassos da Rosa APA, Travassos da Rosa JFS, Pinheiro FP, Vasconcelos PFC. Arboviroses. In: Leão RNQ, coordenador. Doenças infecciosas e parasitárias: enfoque amazônico. Belém: CEJUP; 1997. p. 207-25. [ Links ]
15 Pinheiro FP, Freitas RB; Travassos da Rosa JF, Gabbay YB, Mello WA, LeDuc JW. An outbreak of Mayaro virus disease in Belterra, Brazil: I. Clinical and virological findings. Am J Trop Med Hyg. 1981 May;30(3):674-81. Doi: 10.4269/ajtmh.1981.30.674 [Link] [ Links ]
16 Vasconcelos PFC, Travassos da Rosa APA, Pinheiro FP, Travassos da Rosa JFS. Arboviroses. In: Veronesi R, Focaccia R, editores. Tratado de infectologia. 4. ed. São Paulo: Atheneu; 2009. p. 339-51. [ Links ]
17 Vasconcelos PFC. Emergência do vírus Chikungunya: risco de introdução no Brasil. Rev Pan-Amaz Saude. 2014 set;5(3):9-10. Doi: 10.5123/S2176-62232014000300001 [Link] [ Links ]
18 Korsman SNJ, Zyl GV, Nutt L, Andersson MI, Preiser W. Virologia. Rio de Janeiro: Elsevier; 2014. p. 117-61. [ Links ]
19 Travassos da Rosa APA, Pinheiro FP, Travassos da Rosa ES, Rodrigues SG, Travassos da Rosa JFS, Vasconcelos PFC. Arboviroses. In: Tonelli E, Freire LMS, editores. Doenças infecciosas na infância e adolescência. 2. ed. Belo Horizonte: Medsi; 2000. p. 986-1015. [ Links ]
20 Travassos da Rosa APA, Pinheiro FP, Travassos da Rosa ES, Rodrigues SG, Travassos da Rosa JFS, Vasconcelos PFC. Arboviroses. In: Farhat CK, Carvalho ES, Carvalho LHFR, Succi RCM, editores. Infectologia pediátrica. 2. ed. São Paulo: Atheneu ; 1998. p. 358-67. [ Links ]
21 Barbosa ML, Rocco IM, Felippe JMMS, Cruz AS. Growth and maintenance of Aedes albopictus cell line, clone C6/36, in different media. Rev Inst Adolf Lutz. 1993;53(1-2):63-70. [ Links ]
22 Ammerman NC, Beier-Sexton M, Azad AF. Growth and maintenance of Vero cell lines. Curr Protoc Microbiol. 2008 Nov;11(1):A.4E.1-A.4E.7. Doi: 10.1002/9780471729259.mca04es11 [Link] [ Links ]
23 Macpherson I, Stoker M. Polyoma transformation of hamster cell clones - an investigation of genetic factors affecting cell competence. Virology. 1962 Feb;16(2):147-51. Doi: 10.1016/0042-6822(62)90290-8 [Link] [ Links ]
24 Hull RN, Cherry WR, Tritch OJ. Growth characteristics of monkey kidney cell strains LLC-MK1, LLC-MK2, and LLC-MK2(NCTC-3196) and their utility in virus research. J Exp Med. 1962 May;115(5):903-18. Doi: 10.1084/jem.115.5.903 [Link] [ Links ]
25 Dulbecco R, Vogt M. Some problems of animal virology as studied by the plaque technique. Cold Spring Harb Symp Quant Biol. 1953;18:273-9. Doi: 10.1101/SQB.1953.018.01.039 [Link] [ Links ]
26 Gubler DJ, Kuno G, Sather GE, Velez M, Oliver A. Mosquito cell cultures and specific monoclonal antibodies in surveillance for dengue viruses. Am J Trop Med Hyg. 1984 Jan;33(1):158-65. Doi: 10.4269/ajtmh.1984.33.158 [Link] [ Links ]
27 Celestino Jr AF. Desenvolvimento de técnica de RT-PCR em tempo real para detecção do vírus Mayaro (Togaviridae: Alphavirus) [dissertação]. Belém (PA): Universidade Federal do Pará, Instituto de Ciências Biológicas; 2013. 64 p. [Link] [ Links ]
28 Lanciotti RS, Calisher CH, Gubler DJ, Chang GJ, Vorndam AV. Rapid detection and typing of dengue viruses from clinical samples by using reverse transcriptase-polymerase chain reaction. J Clin Microbiol. 1992 Mar;30(3):545-51. [Link] [ Links ]
29 Causey OR, Maroja OM. Mayaro virus: a new human disease agent. III. Investigation of an epidemic of acute febrile illness on the river Guama in Pará, Brazil, and isolation of Mayaro virus as causative agent. Am J Trop Med Hyg. 1957 Nov;6(6):1017-23. Doi: 10.4269/ajtmh.1957.6.1017 [Link] [ Links ]
30 Nunes MRT, Faria NR, Vasconcelos JM, Golding N, Kraemer MUG, Oliveira LF, et al. Emergence and potential for spread of Chikungunya virus in Brazil. BMC Med. 2015 Apr;13:102. Doi: 10.1186/s12916-015-0348-x [Link] [ Links ]
31 Simon F, Javelle E, Oliver M, Leparc-Goffart I, Marimoutou C. Chikungunya virus infection. Curr Infect Dis Rep. 2011 Jun;13:218. Doi: 10.1007/s11908-011-0180-1 [Link] [ Links ]
32 Way HJ, Bowen ETW, Platt GS. Comparative studies of some African arboviruses in cell culture and in mice. J Gen Virol. 1976 Jan;30(1):123-30. Doi: 10.1099/0022-1317-30-1-123 [Link] [ Links ]
33 Tesh RB. A method for the isolation and identification of dengue viruses, using mosquito cell cultures. Am J Trop Med Hyg. 1979 Nov;28(6):1053-9. Doi: 10.4269/ajtmh.1979.28.1053 [Link] [ Links ]
34 Figueiredo LTM. Uso de células de Aedes albopictus C6/36 na propagação e classificação de arbovírus das famílias Togaviridae, Flaviviridae, Bunyaviridae e Rhabdoviridae. Rev Soc Bras Med Trop. 1990 jan-mar;23(1):13-8. Doi: 10.1590/S0037-86821990000100003 [Link] [ Links ]
35 Arias-Goeta C, Moutailler S, Mousson L, Zouache K, Thiberge JM, Caro V, et al. Chikungunya virus adaptation to a mosquito vector correlates with only few point mutations in the viral envelope glycoprotein. Infect Genet Evol. 2014 Jun;24:116-26. Doi: 10.1016/j.meegid.2014.03.015 [Link] [ Links ]
36 Lim PJ, Chu JJH. A polarized cell model for Chikungunya virus infection: entry and egress of virus occurs at the apical domain of polarized cells. PLoS Negl Trop Dis. 2014 Feb;8(2):e2661. Doi: 10.1371/journal.pntd.0002661 [Link] [ Links ]
37 Lee RCH, Chu JJH. Proteomics profiling of Chikungunya-infected Aedes albopictus C6/36 cells reveal important mosquito cell factors in virus replication. PLoS Negl Trop Dis. 2015 Mar;9(3):e0003544. Doi: 10.1371/journal.pntd.0003544 [Link] [ Links ]
AJUDA FINANCEIRA Este trabalho foi financiado por doações do Ministério da Saúde, Brasil, Instituto Nacional de Ciência e Tecnologia de Febres Hemorrágicas Virais (concessão nº 573739/2008-0) e Conselho Nacional de Desenvolvimento Científico e Tecnológico (concessão nº 457664/2013-4).
Se refere ao doi: 10.5123/S2176-62232018000400003, publicado originalmente em inglês. Traduzido por: Patrícia Campelo Haick
Como citar este artigo / How to cite this article: Ribeiro ACS, Carvalho CAM, Casseb SMM, Rodrigues SG, Vasconcelos PFC, Carvalho VL, et al. Perfis de infecção do vírus Mayaro e do vírus Chikungunya em linhagens de células de mamíferos e mosquitos. Rev Pan-Amaz Saude. 2018 out-dez;9(4):1-11. Doi: http://dx.doi.org/10.5123/S2176-62232018000400003
Recebido: 11 de Setembro de 2017; Aceito: 03 de Agosto de 2018










 texto em
texto em 
 Curriculum ScienTI
Curriculum ScienTI