INTRODUÇÃO
As enterobactérias são um grupo de bacilos Gram-negativos constituintes da microbiota de seres humanos e animais e estão associadas a diversos processos patogênicos1. São isoladas de diferentes ambientes e, quando presentes em águas para consumo humano, são indicativas de contaminação por fezes2.
Desde os anos 1980, as infecções causadas por esses microrganismos se tornaram um problema de saúde pública, devido ao surgimento de amostras multirresistentes a diversas classes de antimicrobianos, limitando as opções terapêuticas3. O aumento do número de amostras bacterianas resistentes pode ser explicado pela pressão seletiva decorrente do uso indiscriminado de antimicrobianos na prática clínica, na pecuária e a consequente contaminação ambiental4.
A presença de bactérias resistentes aos antimicrobianos em águas é um fenômeno bem descrito na literatura científica. Woodford et al.5 descreveram a presença de bactérias multirresistentes e genes que codificam resistência aos antimicrobianos em várias fontes de águas residuais e para consumo humano. Essas bactérias podem ser originárias de contaminação antrópica e resultantes da pressão de seleção decorrente da contaminação ambiental por antibióticos6. No ambiente aquático, pode haver também a transferência de elementos genéticos que codificam resistência, principalmente em sedimentos, o que colabora para a disseminação de bactérias multirresistentes, bem como para a manutenção desses elementos no ambiente6.
Os mecanismos de resistência em enterobactérias, como a produção de β-lactamases de espectro expandido (extended-spectrum β-lactamase - ESBL), metalo-β-lactamases e carbapenemases, são mediados principalmente por elementos genéticos móveis, como plasmídios7, o que justifica a preocupação com o isolamento de enterobactérias resistentes a antimicrobianos em águas, uma vez que existe a possibilidade de contaminação e posterior colonização de indivíduos por esses microrganismos8, assim como a transferência de plasmídios e integrons contendo elementos de resistência antimicrobiana no intestino humano9.
Existem diversos relatos de isolamento de bactérias resistentes e multirresistentes aos antibióticos em águas para abastecimento público e em soluções alternativas de abastecimento. Em estudo realizado por Coleman et al.10, 16,9% dos isolados de Escherichia coli, provenientes de poços do estado de Alberta, no Canadá, foram resistentes a pelo menos um antibiótico. Bactérias multirresistentes também estão presentes em águas para consumo humano. O isolamento de enterobactérias produtoras de ESBL, enzima que hidrolisa cefalosporinas, penicilinas e monobactâmicos, foi relatado por Abera et al.11 em amostras de água colhidas em domicílios atendidos pelo sistema de distribuição de águas de Bahi Dar, na Etiópia. Subba et al.12 também descreveram altas taxas de multirresistência de E. coli isolada de águas encanadas, poços e nascentes utilizadas para consumo no Nepal. O isolamento de E. coli produtora de ESBL em águas para consumo também foi descrito em Bangladesh, com positividade de 26% em 233 cepas de E. coli provenientes de águas de tubulação para abastecimento, além de 36% dos isolados classificados como multirresistentes13.
No Brasil, alguns autores relataram a presença de enterobactérias resistentes aos antimicrobianos em águas para consumo humano e corpos d'água. Gomes-Freitas et al.14 descreveram a presença de amostras bacterianas resistentes à ampicilina, tetraciclina e ciprofloxacina em amostras de águas de bebedouros escolares. A presença de bactérias oportunistas resistentes e multirresistentes também foi descrita em amostras de água mineral à venda em supermercados do interior do estado de São Paulo15. No estado do Rio de Janeiro, foram isoladas bactérias multirresistentes no sistema lacustre de Jacarepaguá e da baía da Guanabara, com a presença de patógenos, como Shigella sp. e Vibrio cholerae, resistentes a atimicrobianos16. A presença de bactérias produtoras de ESBL, em águas contaminadas por efluentes hospitalares, foi relatada na Região Sul do Brasil17,18. No estado de São Paulo, foram detectadas enterobactérias produtoras de carbapenemase em efluentes hospitalares e nos Rios Tietê e Pinheiros, os mais importantes da cidade de São Paulo19,20.
Genes responsáveis por resistência aos antibióticos provenientes de bactérias patogênicas e ambientais podem ser isolados em águas para abastecimento. Embora a presença desses genes na natureza seja anterior ao surgimento da antibioticoterapia21, sua presença também pode ser relacionada a uma pressão de seleção pela contaminação por antimicrobianos ou à transferência horizontal de genes entre diferentes gêneros de bactérias21. Su et al.22 detectaram genes responsáveis pela resistência à tetraciclina, sulfonamidas, macrolídeos, cloranfenicol e quinolonas em águas para consumo humano no sul da China em todas as fases de tratamento até a distribuição domiciliar. Genes codificadores de β-lactamases também foram detectados em águas de poços da região norte da Itália23, e em uma amostra bacteriana de E. coli proveniente de uma amostra de água tratada do nordeste da França24.
O isolamento de genes codificadores de carbapenemases em águas para abastecimento também já foi descrito. Em estudo realizado por Rathinasabapathi et al.25, foram detectados genes responsáveis pela produção da New Delhi metallo-β-lactamase em três amostras de águas tratadas com cloro antes da distribuição para consumo.
Dessa forma, a detecção de enterobactérias resistentes aos antimicrobianos em águas para consumo humano é um indicador de contaminação antrópica no ambiente, bem como um potencial problema de saúde pública. Assim, o objetivo deste trabalho foi isolar e identificar enterobactérias resistentes a antimicrobianos e produtoras de ESBL, isoladas de águas para consumo humano, provenientes de diferentes sistemas de
abastecimento público e de soluções alternativas de abastecimento da região centro-oeste do estado de São Paulo.
MATERIAIS E MÉTODOS
COLETA DAS AMOSTRAS E DETERMINAÇÃO DA COLIMETRIA
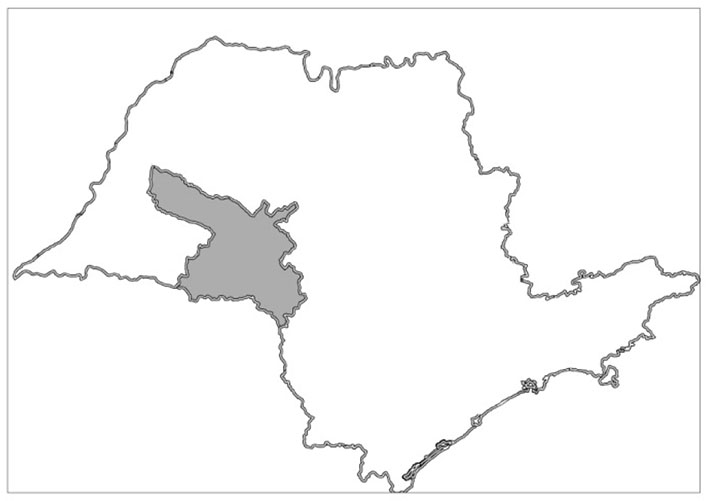
No período de fevereiro a novembro de 2015, 3.729 amostras de água de 62 municípios da região centro-oeste do estado de São Paulo (Figura 1) foram analisadas, pelo Instituto Adolfo Lutz de Marília, quanto à positividade para coliformes totais e/ou E. coli pela técnica de substrato cromogênico e fluorogênico Colilert (IDEXX Laboratories Inc., Westbrook, Maine, EUA). As amostras provenientes do sistema de abastecimento público foram coletadas após o tratamento nos pontos de entrada das edificações (cavalete) ou nos casos de soluções alternativas coletivas, direto do poço, em frascos estéreis de 120 mL, mantidos sob refrigeração até o momento do processamento.
ISOLAMENTO E IDENTIFICAÇÃO
As amostras de água positivas para coliformes totais e/ou E. coli foram repicadas por estriamento em placas de Petri contendo Ágar MacConkey e incubadas por 18-24 h para isolamento. Uma única colônia de cada população morfologicamente semelhante foi identificada por técnicas bioquímicas clássicas de microbiologia até o nível de gênero1,26.
DETECÇÃO DA RESISTÊNCIA A ANTIMICROBIANOS E PRODUÇÃO DE ESBL
Para a detecção de resistência às drogas, foi empregada a técnica de difusão a partir do disco27. Foram avaliadas as seguintes classes de antimicrobianos: aminoglicosídeos (amicacina e gentamicina), monobactâmicos (aztreonam), cefalosporinas (ceftazidima, cefpodoxima, cefotaxima, cefepima, cefoxitina e cefalotina), carbapenêmicos (ertapeném, imipeném e meropeném), fluoroquinolonas (ácido nalidíxico e norfloxacina) e associações de penicilinas e inibidores de β-lactamase (amoxacilina + ácido clavulânico, ampicilina + sulbactam e ticarcilina + clavulanato). As amostras de enterobactérias foram consideradas sensíveis, com resistência intermediária ou resistentes, de acordo com os pontos de corte definidos pelo manual do Clinical and Laboratory Standards Institute (CLSI) de 201728, exceto para a cefalotina, que não possui ponto de corte determinado por esse manual, onde foram empregados os pontos de corte determinados pelo CLSI publicado em 201529. A detecção de ESBL foi realizada pela técnica de disco aproximação, com disco de amoxacilina + ácido clavulânico30.
RESULTADOS
AMOSTRAS
Do total de 3.726 amostras de água analisadas no período do estudo, 67 amostras de 29 municípios da região centro-oeste do Estado foram positivas para coliformes totais e/ou E. coli e incluídas neste estudo. Dessas, apenas duas foram obtidas de poços de soluções alternativas coletivas.
ISOLAMENTO E IDENTIFICAÇÃO
Os gêneros isolados com maior frequência foram: Enterobacter sp. (28), E. coli (19), Klebsiella sp. (sete), Edwardsiella sp. (quatro), Proteus sp. (três), Citrobacter sp. (dois), Providencia sp. (dois), Salmonella sp. (um) e Serratia sp. (um) (Tabela 1).
Tabela 1 - Enterobactérias resistentes aos antimicrobianos isoladas de águas para consumo humano na região centro-oeste do estado de São Paulo, Brasil
| Enterobactérias | Aminoglicosídeos* | Carbapenêmicos† | Cefalosporinas | Penicilinas | Fluoroquinolonas | Monobactâmicos | |||||||||
| Gênero | N | KF (1ª) | FOX (2ª) | CAZ (3ª) | CPD (3ª) | CTX (3ª) | FEP (4ª) | AMC | SAM | TIM | NA | NOR | ATM | ||
| Citrobacter sp. | 2 | - | - | ERI | ERI | - | - | - | - | - | ERI | - | - | - | - |
| Escherichia coli | 19 | - | - | 3 | 3 | - | - | 1 | - | - | 1 | 1 | 2 | 1 | 1 |
| Edwardsiella sp. | 4 | - | - | 2 | 4 | - | - | - | - | 1 | - | - | - | - | - |
| Enterobacter sp. | 28 | - | - | ERI | ERI | - | - | 1 | - | ERI | ERI | 3 | - | - | 1 |
| Klebsiella sp. | 7 | - | - | 2 | 2 | - | - | - | - | - | 1 | - | - | - | - |
| Proteus sp. | 3 | - | - | ERI | 2 | - | 1 | 1 | - | - | 1 | - | - | - | 1 |
| Providencia sp. | 2 | - | - | ERI | - | - | - | 1 | - | - | - | - | - | - | 1 |
| Salmonella sp. | 1 | - | - | 1 | 1 | - | - | 1 | - | - | - | - | 1 | - | - |
| Serratia sp. | 1 | - | - | ERI | - | - | - | - | - | - | - | - | - | - | - |
| Total | 67 | - | - | 8 | 12 | - | 1 | 5 | - | 1 | 3 | 4 | 3 | 1 | 4 |
Sinal convencional utilizado: - Dado numérico igual a zero, não resultante de arredondamento; N: Número de isolados bacterianos; *: Amicacina e gentamicina; †: Ertapeném, imipeném e meropeném; KF: Cefalotina; FOX: Cefoxitina; CAZ: Ceftazidima; CPD: Cefpodoxima; CTX: Cefotaxima; FEP: Cefepima; AMC: Amoxacilina + ácido clavulânico; SAM: Ampicilina + sulbactam; TIM: Ticarcilina + clavulanato; NA: Ácido nalidíxico; NOR: Norfloxacina; ATM: Aztreonam; ERI: Espécie que apresenta resistência intrínseca ao antimicrobiano avaliado.
DETECÇÃO DA RESISTÊNCIA AOS ANTIMICROBIANOS E PRODUÇÃO DE ESBL
Das 67 amostras bacterianas analisadas, 21 (31,34%) foram resistentes a pelo menos um agente antimicrobiano. As cefalosporinas de segunda geração apresentaram o maior número de cepas resistentes (32,43%), seguidas pelas penicilinas (14,92%) (Tabela 2). Quando agrupados por classes, três amostras bacterianas (4,47%) foram resistentes a cefalosporinas de primeira e segunda gerações, duas (2,99%) a cefalosporinas de primeira e segunda gerações + penicilinas, duas (2,99%) a cefalosporinas de terceira geração + monbactâmicos, uma (1,49%) a cefalosporinas de segunda geração + penicilinas, uma (1,49%) a cefalosporinas de primeira, segunda e terceira gerações + ácido nalidíxico, uma (1,49%) a cefalosporinas de segunda e terceira gerações + penicilinas + monobactâmicos e uma (1,49%) a cefalosporinas de primeira, segunda e terceira gerações + penicilinas + fluoroquinolonas + monobactâmicos.
Tabela 2 - Resistência, por classe de antimicrobianos, de enterobactérias isoladas de águas para consumo humano na região centro-oeste do estado de São Paulo, Brasil
| Classe de antimicrobiano | Sensível | Resistência intermediária | Resistente | |||
| N | % | N | % | N | % | |
| Aminoglicosídeos | 65 | 97,01 | 2 | 2,99 | - | - |
| Carbapenêmicos | 63 | 94,03 | 4 | 5,97 | - | - |
| Cefalosporinas 1ª geração* | 16 | 51,61 | 7 | 22,58 | 8 | 25,81 |
| Cefalosporinas 2ª geração* | 24 | 64,87 | 1 | 2,70 | 12 | 32,43 |
| Cefalosporinas 3ª geração | 62 | 92,54 | - | - | 5 | 7,46 |
| Cefalosporinas 4ª geração | 66 | 98,51 | 1 | 1,49 | - | - |
| Fluoroquinolonas | 57 | 85,08 | 5 | 7,46 | 5 | 7,46 |
| Monobactâmicos | 62 | 92,54 | 1 | 1,49 | 4 | 5,97 |
| Penicilinas | 57 | 85,08 | - | - | 10 | 14,92 |
Sinal convencional utilizado: - Dado numérico igual a zero, não resultante de arredondamento; *: Espécies com resistência intrínseca não foram avaliadas; N: Número de amostras bacterianas.
A cefoxitina foi o antimicrobiano testado que apresentou o maior número de cepas resistentes (12), seguido por cefalotina (oito), cefotaxima (cinco), ticarcilina + clavulanato e aztreonam (quatro cada), ampicilina + sulbactam e ácido nalidíxico (três cada), amoxacilina + ácido clavulânico, norfloxacina e cefpodoxima (um, cada) (Tabela 1). A cefalotina apresentou o maior número de isolados com resistência intermediária, seguido por ácido nalidíxico, imipeném e gentamicina (Tabela 2).
A espécie E. coli apresentou o maior número de cepas com resistência intermediária (oito), sendo cinco dessas apenas à cefalotina, uma à gentamicina e ao meropeném, respectivamente, e uma ao imipeném e ao ácido nalidíxico. Dos cinco isolados de Enterobacter sp. com resistência intermediária, dois foram resistentes à cefpodoxima, um ao ácido nalidíxico e à cefpodoxima, um ao acido nalidíxico e um ao imipeném; uma cepa de Edwardisiella sp. expressou resistência intermediária à cefpodoxima e à cefalotina e uma apenas à cefalotina. Foi detectado um isolado de Proteus sp. com resistência intermediária à amicacina, gentamicina, ertapeném e imipeném; um de Providencia sp. com resistência intermediária à cefpodoxima, cefepima e norfloxacina; e um de Salmonella sp. expressou resistência intermediária ao aztreonam. As outras espécies isoladas no estudo não apresentaram cepas com resistência intermediária a qualquer dos antimicrobianos testados.
Nenhum dos isolados foi produtor de ESBL, e a maioria (14,94%) das amostras de enterobactérias que apresentou algum tipo de resistência foi resistente a apenas uma classe de antimicrobiano; contudo, uma (1,49%) amostra de E. coli foi resistente a quatro classes (Tabela 3).
Tabela 3 - Resistência e multirresistência às classes de antimicrobianos testadas em enterobactérias provenientes de águas para abastecimento público da região centro-oeste do estado de São Paulo, Brasil
| Enterobactérias | 0R | 1R | 2R | 3R | 4R | Total | ||||||||||||
| N | % | N | % | N | % | N | % | N | % | N | % | |||||||
| Citrobacter sp. | 2 | 2,98 | - | - | - | - | - | - | - | - | 2 | 2,98 | ||||||
| Escherichia coli | 13 | 19,40 | 4 | 6,00 | 1 | 1,49 | - | - | 1 | 1,49 | 19 | 28,38 | ||||||
| Edwardsiella sp. | 1 | 1,49 | 2 | 2,98 | 1 | 1,49 | 1 | 1,49 | - | - | 5 | 7,45 | ||||||
| Enterobacter sp. | 24 | 35,82 | 3 | 4,47 | 1 | 1,49 | - | - | - | - | 28 | 41,78 | ||||||
| Klebsiella sp. | 4 | 6,00 | - | - | 1 | 1,49 | 1 | 1,49 | - | - | 6 | 8,98 | ||||||
| Proteus sp. | - | - | 1 | 1,49 | 1 | 1,49 | 1 | 1,49 | - | - | 3 | 4,47 | ||||||
| Providencia sp. | 1 | 1,49 | - | - | 1 | 1,49 | - | - | - | - | 2 | 2,98 | ||||||
| Salmonella sp. | - | - | - | - | 1 | 1,49 | - | - | - | - | 1 | 1,49 | ||||||
| Serratia sp. | 1 | 1,49 | - | - | - | - | - | - | - | - | 1 | 1,49 | ||||||
| Total | 46 | 68,67 | 10 | 14,94 | 7 | 10,43 | 3 | 4,47 | 1 | 1,49 | 67 | 100,00 | ||||||
0R: Sensível a todos os antimicrobianos; 1R: Resistente a uma classe de antimicrobianos; 2R: Resistente a duas classes de antimicrobianos; 3R: Resistente a três classes de antimicrobianos; 4R: Resistente a quatro classes de antimicrobianos; N: Número de amostras bacterianas. Sinal convencional utilizado: - Dado numérico igual a zero, não resultante de arredondamento.
DISCUSSÃO
A presença de microrganismos resistentes aos antimicrobianos em águas para consumo humano se caracteriza como um problema de saúde pública, pelo perigo de disseminação de cepas resistentes e consequente colonização dos indivíduos expostos8. Neste estudo, o gênero Enterobacter sp. foi o mais isolado de águas para consumo humano, o que pode estar relacionado com a capacidade de formação de biofilme durante o processo de tratamento, dificultando a ação do cloro no tratamento da água31. Mais da metade das amostras de enterobactérias desta pesquisa foram resistentes a pelo menos uma classe de antimicrobianos, resultado inferior ao descrito na África e na Europa2,32, em águas para consumo humano, e superior ao encontrado por Gomes-Freitas et al.14 no Brasil.
A cefalotina e a cefoxitina (cefalosporinas de primeira e segunda gerações) foram as drogas que tiveram a maior taxa de amostras bacterianas resistentes, possivelmente por serem antibióticos de uso amplo há muitos anos para o tratamento de infecções comunitárias e hospitalares, e com mecanismos genéticos de resistência bacteriana disseminados entre as espécies da família Enterobacteriaceae33. A resistência às cefalosporinas de terceira geração foi detectada em cinco amostras e nenhuma foi resistente à cefepima, uma cefalosporina de quarta geração. Dados similares foram relatados por Talukdar et al.13, em estudo com amostras de E. coli provenientes de águas encanadas de Daka, Bangladesh, onde descreveram 9% de resistência à cefotaxima, uma cefalosporina de terceira geração. Embora, neste trabalho, a prevalência de resistência às cefalosporinas mais recentes tenha sido baixa, AbdelRahim et al.34 descreveram 81,5% de resistência à cefepima em amostras de enterobactérias isoladas em águas para consumo no Egito. As taxas de resistência ao grupo das penicilinas foram baixas, similares aos dados descritos por Gomes-Freitas et al.14 em amostras de águas provenientes de bebedouros de uma escola no interior do estado de Goiás, Brasil.
Apenas duas amostras bacterianas foram resistentes ao ácido nalidíxico, número inferior ao descrito por Talukdar et al.13 na cidade de Daca, Bangladesh, que apresentou um total de 37% de E. coli resistentes a essa droga; mas similar ao obtido por Coleman et al.10 com E. coli, que relataram apenas 0,5% de resistência a esse antimicrobiano. Os mesmos autores10 descrevem baixa taxa de resistência para os aminoglicosídeos, o que diverge do presente estudo, que descreveu a totalidade de isolados sensíveis a esse grupo de antibióticos. A resistência ao aztreonam foi detectada em 5,97% das amostras, dado inferior ao descrito por Oliveira e Van Der Sand17 em isolados de águas de superfícies, em um estudo no sul do Brasil.
A resistência a três ou mais classes de antimicrobianos é denominada multirresistência e se caracteriza como um problema de saúde pública por diminuir as opções terapêuticas em um processo infeccioso35. Neste trabalho, uma amostra de Proteus sp. e uma de Salmonella sp. foram resistentes a três classes de
antimicrobianos, respectivamente; e uma amostra de E. coli apresentou resistência a quatro classes de antimicrobianos. Isolados multirresistentes são descritos com maior frequência em águas residuais6, mas também podem ser isolados de águas de superfície e para consumo humano, como nascentes e água mineral engarrafada15,36, o que pode expor a população à colonização por bactérias resistentes e multirresistentes a antibióticos8. Em um estudo com cepas de E. coli provenientes de soluções alternativas, Coleman et al.10 descreveram 22% de resistência a uma ou duas classes de antibióticos e 14% de isolados multirresistentes, valores superiores aos descritos neste estudo.
Muitas bactérias tornam-se multirresistentes pela aquisição de elementos genéticos codificadores de enzimas que hidrolisam antibióticos, como é o caso da ESBL e das carbapenemases7. Neste trabalho, nenhuma amostra foi produtora de ESBL pelo método fenotípico utilizado; contudo, estudos detectaram a presença de genes codificadores de ESBL em diversos grupos bacterianos isolados de águas no Brasil e em outros países6,15,22, bem como do DNA bacteriano extraído diretamente da água2,32,36. A ausência de amostras produtoras de ESBL neste estudo pode estar relacionada com a falta de acesso às técnicas moleculares capazes de detectar o gene produtor da enzima, que muitas vezes pode não ser expressa fenotipicamente.
Um dado interessante obtido neste estudo foi a presença de amostras de enterobactérias com resistência intermediária aos carbapenêmicos. Uma cepa de Proteus sp., isolada de solução alternativa de abastecimento, apresentou resistência intermediária ao imipeném e ertapeném, uma de Enterobacter sp. e uma de E. coli ao imipeném e um de E. coli ao meropeném. O isolamento de bactérias resistentes aos carbapenêmicos e detecção de genes produtores de carbapenemase já foi descrito em águas residuais, baías e lagos das Regiões Sul e Sudeste do Brasil16,18,19, o que ressalta a importância do monitoramento de bactérias resistentes em águas, como as captadas no centro-oeste do estado de São Paulo.
CONCLUSÃO
Os achados descritos neste estudo mostram que as águas para consumo podem ser fonte de disseminação de bactérias Gram-negativas resistentes e multirresistentes, o que caracteriza um problema de saúde pública, e ressaltam a importância do tratamento adequado das águas, para impedir a exposição da população a enterobactérias resistentes e multirresistentes, bem como do monitoramento dos sistemas de abastecimento público, para a detecção de bactérias resistentes a antimicrobianos.










 text in
text in