Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista Pan-Amazônica de Saúde
versão impressa ISSN 2176-6215versão On-line ISSN 2176-6223
Rev Pan-Amaz Saude vol.10 Ananindeua 2019 Epub 08-Nov-2019
http://dx.doi.org/10.5123/s2176-6223201900065
ARTÍCULO ORIGINAL
Resistencia a antimicrobianos de enterobacterias aisladas de aguas destinadas al abastecimiento público en la región centro-oeste del estado de São Paulo, Brasil
1 Instituto Adolfo Lutz, Núcleo de Ciências Biomédicas, Marília, São Paulo, Brasil
2 Instituto Adolfo Lutz, Núcleo de Ciências Químicas e Bromatológicas, Marília, São Paulo, Brasil
OBJETIVOS:
Aislar e identificar enterobacterias resistentes a antimicrobianos de aguas para consumo humano en la región centro-oeste del estado de São Paulo, Brasil.
MATERIALES Y MÉTODOS:
Se analizaron 3.726 muestras de aguas de 62 municipios de esa región, provenientes del sistema público de abastecimiento y de pozos como soluciones alternativas colectivas. Las muestras positivas para coliformes totales y/o Escherichia coli fueron sembradas en Agar MacConkey, para aislado e identificación. Se evaluaron las enterobacterias aisladas para detección de la resistencia a antimicrobianos y producción de β-lactamasa de espectro extendido.
RESULTADOS:
De las 67 muestras de aguas de 29 municipios con crecimiento de enterobacterias fueron resistentes: 21 (31,34%) a, al menos, un antimicrobiano, siete (10,43%) a dos clases de antimicrobianos, tres (4,47%) a tres clases y una (1,49%) a cuatro clases. La droga que presentó el mayor número de muestras bacterianas resistentes fue la cefoxitina (12), seguida por cefalotina (ocho), cefotaxima (cinco), ticarcilina + clavulanato (cuatro), aztreonam (cuatro), ácido nalidíxico (tres) y ampicilina + sulbactam (tres). Las cefalosporinas de segunda generación presentaron el mayor número de muestras bacterianas resistentes (12; 32,43%), seguidas por las penicilinas (10; 14,92%) y cefalosporinas de primera generación (8; 25,81%). Tres (4,47%) muestras fueron resistentes a tres clases de antimicrobianos y una (1,49%), a cuatro clases.
CONCLUSIÓN:
Las aguas para consumo pueden ser fuente de diseminación de bacterias gramnegativas resistentes y multirresistentes, caracterizando un problema de salud pública y tornando importante el tratamiento adecuado de las aguas y el monitoreo de los sistemas de abastecimiento público, para la detección de bacterias resistentes a antimicrobianos.
Palabras clave: Antibacterianos; Farmacorresistencia Bacteriana; Agua Potable; Enterobacteriaceae
INTRODUCCIÓN
Las enterobacterias son un grupo de bacilos gramnegativos que constituyen la microbiota de humanos y animales y están asociadas con varios procesos patógenos1. Son aisladas de diferentes ambientes y, cuando están presentes en el agua para consumo humano, son indicativos de contaminación por heces2.
Desde la década de 1980, las infecciones causadas por estos microorganismos se han convertido en un problema de salud pública, debido a la aparición de muestras multirresistentes a diferentes clases de antimicrobianos, lo que limita las opciones terapéuticas3. El aumento en el número de muestras bacterianas resistentes puede explicarse por la presión selectiva resultante del uso indiscriminado de antimicrobianos en la práctica clínica, en la pecuaria y la consiguiente contaminación ambiental4.
La presencia de bacterias resistentes a los antimicrobianos en las aguas es un fenómeno bien descrito en la literatura científica. Woodford et al.5 describieron la presencia de bacterias y genes multirresistentes que codifican la resistencia a los antimicrobianos en diversas fuentes de aguas residuales y para consumo humano. Estas bacterias pueden originarse a partir de la contaminación antrópica y resultar de la presión de selección resultante de la contaminación ambiental por antibióticos6. En el medio ambiente acuático, también puede haber transferencia de elementos genéticos que codifican resistencia, principalmente en sedimentos, lo que contribuye a la diseminación de bacterias multirresistentes, así como al mantenimiento de estos elementos en el medio ambiente6.
Los mecanismos de resistencia en las enterobacterias, como la producción de β-lactamasas de espectro extendido (β-lactamasas de espectro extendido - BLEE), metalo-betalactamasas y carbapenemasas, están mediadas principalmente por elementos genéticos móviles, como los plásmidos7, lo que justifica la preocupación con el aislado de enterobacterias resistentes a los antimicrobianos en el agua, ya que existe la posibilidad de contaminación y posterior colonización de individuos por estos microorganismos8, así como la transferencia de plásmidos e integrones que contienen elementos de resistencia antimicrobiana en el intestino humano9.
Hay varios informes de aislados de bacterias resistentes y multirresistentes a antibióticos en aguas para abastecimiento público y en soluciones de alternativas de abastecimiento. En un estudio de Coleman et al.10, el 16.9% de los aislados de Escherichia coli, provenientes de pozos en el estado de Alberta, Canadá, eran resistentes a, al menos, un antibiótico. Las bacterias multirresistentes también están presentes en el agua para consumo humano. Abera et al.11 relataron sobre el aislado de enterobacterias productoras de BLEE, una enzima que hidroliza cefalosporinas, penicilinas y monobactámicos11 en muestras de agua colectadas en hogares servidos por el sistema de distribución de agua en Bahi Dar, Etiopía. Subba et al.12 también describieron altas tasas de resistencia a múltiples fármacos de E. coli aislado de agua entubada, pozos y manantiales utilizados para consumo en Nepal. El aislado de E. coli productora de BLEE en el agua potable también se ha descrito en Bangladesh, con un 26% positivo en 233 cepas de E. coli de las tuberías para abastecimiento de agua, además del 36% de los aislados clasificados como multirresistentes13.
En Brasil, algunos autores informaron la presencia de enterobacterias resistentes a los antimicrobianos en el agua para consumo humano y cuerpos de agua. Gomes-Freitas et al.14 describieron la presencia de muestras bacterianas resistentes a la ampicilina, la tetraciclina y la ciprofloxacina en muestras de agua en los dispensadores de agua potable de las escuelas. La presencia de bacterias oportunistas resistentes y multirresistentes también se ha descrito en muestras de agua mineral a la venta en supermercados en el interior del estado de São Paulo15. En el estado de Rio de Janeiro, se aislaron bacterias multirresistentes en el sistema lacustre de Jacarepaguá y de la bahía de Guanabara, con la presencia de patógenos, como Shigella sp. y Vibrio cholerae, resistentes a los antimicrobianos16. La presencia de bacterias productoras de BLEE en aguas contaminadas por efluentes hospitalarios ha sido reportada en el sur de Brasil17,18. En el estado de São Paulo, se detectaron enterobacterias productoras de carbapenemasas en efluentes hospitalarios y en los ríos Tietê y Pinheiros, los más importantes en la ciudad de São Paulo19,20.
Los genes responsables por la resistencia a los antibióticos de las bacterias patógenas y ambientales pueden ser aislados en el agua abastecimiento. Aunque la presencia de estos genes en la naturaleza es anterior a la aparición de la antibioticoterapia21, su presencia también puede estar relacionada con una presión de selección debido a la contaminación antimicrobiana o con la transferencia horizontal de genes entre diferentes géneros de bacterias21. Su et al.22 detectaron genes responsables por la resistencia a la tetraciclina, sulfonamidas, macrólidos, cloranfenicol y quinolonas en agua para consumo humano en el sur de China en todas las etapas del tratamiento hasta la distribución en el hogar. También se han detectado genes codificadores de β-lactamasas en agua de pozo en el norte de Italia23, y en una muestra bacteriana de E. coli proveniente de una muestra de agua tratada del nordeste de Francia24.
También ha sido descrito el aislado de genes codificadores de carbapenemasas en aguas para abastecimiento. En estudio realizado por Rathinasabapathi et al.25, se detectaron genes responsables por la producción de New Delhi metalo-betalactamasas (NDM) en tres muestras de aguas tratadas con cloro antes de su distribución para consumo.
De esa forma, la detección de enterobacterias resistentes a los antimicrobianos en aguas para consumo humano es un indicador de contaminación antrópica en el ambiente, bien como un potencial problema de salud pública. De esta forma, el objetivo de este trabajo fue de aislar e identificar enterobacterias resistentes a antimicrobianos y productoras de BLEE, aisladas de aguas para consumo humano, provenientes de diferentes sistemas de abastecimiento público y de soluciones alternativas de abastecimiento de la región centro-oeste del estado de São Paulo.
MATERIALES Y MÉTODOS
COLECTA DE LAS MUESTRAS Y DETERMINACIÓN DE LA COLIMETRIA
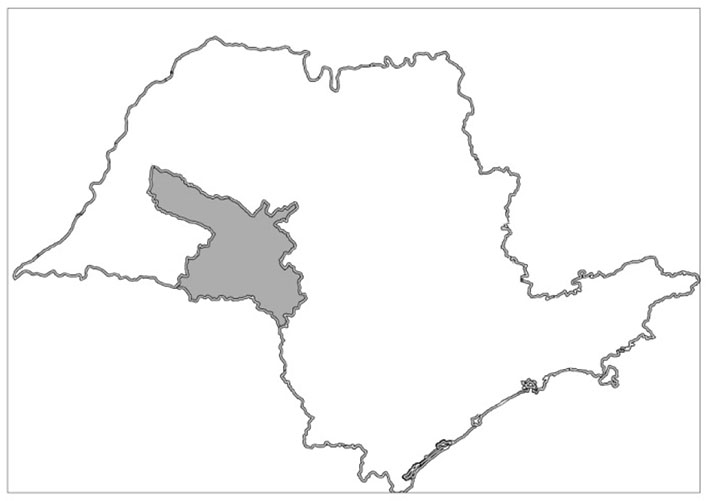
En el período de febrero a noviembre de 2015, el Instituto Adolfo Lutz de Marília analizó 3.729 muestras de agua de 62 municipios de la región centro-oeste del estado de São Paulo (Figura 1), en relación a la positividad para coliformes totales y/o E coli utilizando la técnica de sustrato cromogénico y fluorogénico Colilert (IDEXX Laboratories Inc., Westbrook, Maine, EE. UU.). Las muestras del sistema de abastecimiento público se recogieron después del tratamiento en los puntos de entrada de los edificios (caballete) o en el caso de soluciones alternativas colectivas, directamente del pozo, en matraces estériles de 120ml, se mantuvieron refrigeradas hasta el momento del procesamiento.
AISLADO E IDENTIFICACIÓN
Las muestras de agua positivas para coliformes totales y/o E. coli fueron repicadas por estriado en placas de Petri con Ágar MacConkey e incubadas por 18-24 h para aislado. Una única colonia de cada población morfológicamente semejante fue identificada por técnicas bioquímicas clásicas de microbiología hasta el nivel de género1,26.
DETECCIÓN DE LA RESISTENCIA A ANTIMICROBIANOS Y PRODUCCIÓN DE BLEE
Para la detección de resistencia a los medicamentos, se utilizó la técnica de difusión en disco27. Se evaluaron las siguientes clases de antimicrobianos: aminoglucósidos (amikacina y gentamicina), monobactámicas (aztreonam), cefalosporinas (ceftazidima, cefpodoxima, cefotaxima, cefepima, cefoxitina y cefalotina), carbapénemicos (ertapenem, imipenem y meropenem) y combinaciones de penicilinas e inhibidores de la β-lactamasa (amoxicilina + ácido clavulánico, ampicilina + sulbactam y ticarcilina + clavulanato). Las mutaciones enterobacterianas se consideraron sensibles, con resistencia intermedia o resistente, de acuerdo con los puntos de corte definidos por el manual del Instituto de Normas Clínicas y de Laboratorio (CLSI) de 201728, a excepción de la cefalotina, que no tiene un límite determinado por este manual, donde se utilizaron los puntos de corte determinados por el CLSI publicado en 201529. La detección de BLEE se realizó utilizando la técnica de disco de aproximación, utilizando amoxicilina + ácido clavulánico30.
RESULTADOS
MUESTRAS
Del total de 3.726 muestras de agua analizadas en el período del estudio, 67 muestras de 29 municipios de la región centro-oeste del Estado fueron positivas para coliformes totales y/o E. coli e incluidas en este estudio. De esas, apenas dos se obtuvieron de pozos de soluciones alternativas colectivas.
AISLADO E IDENTIFICACIÓN
Los géneros aislados con más frecuencia fueron: Enterobacter sp. (28), E. coli (19), Klebsiella sp. (siete), Edwardsiella sp. (cuatro), Proteus sp. (tres), Citrobacter sp. (dos), Providencia sp. (dos), Salmonella sp. (uno) e Serratia sp. (uno) (Tabla 1).
Tabla 1 - Enterobacterias resistentes a los antimicrobianos aisladas de aguas para consumo humano en la región centro-oeste del estado de São Paulo, Brasil
| Enterobacterias | Aminoglucósidos* | Carbapenémicos† | Cefalosporinas | Penicilinas | Fluoroquinolonas | Monobactámicos | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Género | N | KF (1ª) | FOX (2ª) | CAZ (3ª) | CPD (3ª) | CTX (3ª) | FEP (4ª) | AMC | SAM | TIM | NA | NOR | ATM | ||
| Citrobacter sp. | 2 | - | - | ERI | ERI | - | - | - | - | - | ERI | - | - | - | - |
| Escherichia coli | 19 | - | - | 3 | 3 | - | - | 1 | - | - | 1 | 1 | 2 | 1 | 1 |
| Edwardsiella sp. | 4 | - | - | 2 | 4 | - | - | - | - | 1 | - | - | - | - | - |
| Enterobacter sp. | 28 | - | - | ERI | ERI | - | - | 1 | - | ERI | ERI | 3 | - | - | 1 |
| Klebsiella sp. | 7 | - | - | 2 | 2 | - | - | - | - | - | 1 | - | - | - | - |
| Proteus sp. | 3 | - | - | ERI | 2 | - | 1 | 1 | - | - | 1 | - | - | - | 1 |
| Providencia sp. | 2 | - | - | ERI | - | - | - | 1 | - | - | - | - | - | - | 1 |
| Salmonella sp. | 1 | - | - | 1 | 1 | - | - | 1 | - | - | - | - | 1 | - | - |
| Serratia sp. | 1 | - | - | ERI | - | - | - | - | - | - | - | - | - | - | - |
| Total | 67 | - | - | 8 | 12 | - | 1 | 5 | - | 1 | 3 | 4 | 3 | 1 | 4 |
Señal convencional utilizada: - Dato numérico igual a cero, no resultante de redondeo; N: Número de aislados bacterianos; *: Amikacina y gentamicina; †: Ertapenem, imipenem y meropenem; KF: Cefalotina; FOX: Cefoxitina; CAZ: Ceftazidima; CPD: Cefpodoxima; CTX: Cefotaxima; FEP: Cefepima; AMC: Amoxicilina + ácido clavulánico; SAM: Ampicilina + sulbactam; TIM: Ticarcilina + clavulanato; NA: Ácido nalidíxico; NOR: Norfloxacina; ATM: Aztreonam; ERI: Especie que presenta resistencia intrínseca al antimicrobiano evaluado.
DETECCIÓN DE LA RESISTENCIA A LOS ANTIMICROBIANOS Y PRODUCCIÓN DE BLEE
De las 67 muestras bacterianas analizadas, 21 (31,34%) eran resistentes a al menos un agente antimicrobiano. Las cefalosporinas de segunda generación tuvieron el mayor número de cepas resistentes (32.43%), seguidas de las penicilinas (14.92%) (Tabla 2). Cuando se agruparon por clase, tres muestras bacterianas (4.47%) fueron resistentes a cefalosporinas de primera y segunda generación, dos (2.99%) a cefalosporinas + penicilinas de primera y segunda generación, dos (2.99%) a cefalosporinas tercera generación + monobactámicos, una (1.49%) a cefalosporinas + penicilinas de segunda generación, una (1.49%) a cefalosporinas de primera, segunda y tercera generación + ácido nalidíxico, una (1.49%) a cefalosporinas segunda y tercera generación + penicilinas + monobactámicos y una (1.49%) a cefalosporinas de primera, segunda y tercera generación + penicilinas + fluoroquinolonas + monobactámicos.
Tabla 2 - Resistencia, por clase de antimicrobianos, de enterobacterias aisladas de agua para consumo humano en la región centro-oeste del estado de São Paulo, Brasil
| Clase de antimicrobiano | Sensible | Resistencia intermediaria | Resistente | |||
|---|---|---|---|---|---|---|
| N | % | N | % | N | % | |
| Aminoglucósidos | 65 | 97,01 | 2 | 2,99 | - | - |
| Carbapenémicos | 63 | 94,03 | 4 | 5,97 | - | - |
| Cefalosporinas 1ª generación* | 16 | 51,61 | 7 | 22,58 | 8 | 25,81 |
| Cefalosporinas 2ª generación* | 24 | 64,87 | 1 | 2,70 | 12 | 32,43 |
| Cefalosporinas 3ª generación | 62 | 92,54 | - | - | 5 | 7,46 |
| Cefalosporinas 4ª generación | 66 | 98,51 | 1 | 1,49 | - | - |
| Fluoroquinolonas | 57 | 85,08 | 5 | 7,46 | 5 | 7,46 |
| Monobactámicos | 62 | 92,54 | 1 | 1,49 | 4 | 5,97 |
| Penicilinas | 57 | 85,08 | - | - | 10 | 14,92 |
Señal convencional utilizada: - Dato numérico igual a cero, no resultante de redondeo; *: Especies con resistencia intrínseca no fueron evaluadas; N: Número de muestras bacterianas.
La cefoxitina fue el antimicrobiano probado que tuvo el mayor número de cepas resistentes (12), seguido de cefalotina (ocho), cefotaxima (cinco), ticarcilina + clavulanato y aztreonam (cuatro cada uno), ampicilina + sulbactam y ácido nalidíxico (tres cada uno), amoxicilina + ácido clavulánico, norfloxacina y cefpodoxima (uno, cada uno) (Tabla 1). La cefalotina presentó el mayor número de aislamientos con resistencia intermedia, seguida de ácido nalidíxico, imipenem y gentamicina (Tabla 2).
Las especies de E. coli mostraron el mayor número de cepas con resistencia intermedia (ocho), cinco de ellas solo a la cefalotina, una a la gentamicina y meropenem, respectivamente, y una a imipenem y ácido nalidíxico. De los cinco aislados de Enterobacter sp. con resistencia intermedia, dos fueron resistentes a la cefpodoxima, uno al ácido nalidíxico y la cefpodoxima, uno al ácido nalidíxico y uno al imipenem; una cepa de Edwardisiella sp. expresó resistencia intermedia a la cefpodoxima y cefalotina y una solo a la cefalotina. Se detectó un aislado de Proteus sp. con resistencia intermedia a la amikacina, gentamicina, ertapenem e imipenem; uno de Providencia sp. con resistencia intermedia a cefpodoxima, cefepima y norfloxacina; y uno de Salmonella sp. resistencia intermedia al aztreonam. Las otras especies aisladas en el estudio no mostraron cepas con resistencia intermedia a ninguno de los antimicrobianos probados.
Ninguno de los aislados fue productor de BLEE, y la mayoría (14,94%) de las muestras de enterobacterias que presentó algún tipo de resistencia fue resistente a apenas una clase de antimicrobiano; sin embargo, una (1,49%) muestra de E. coli fue resistente a cuatro clases (Tabla 3).
Tabla 3 - Resistencia y multirresistencia a clases de antimicrobianos probadas en enterobacterias provenientes de aguas para abastecimiento público de la región centro-oeste del estado de São Paulo, Brasil
| Enterobacterias | 0R | 1R | 2R | 3R | 4R | Total | ||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| N | % | N | % | N | % | N | % | N | % | N | % | |
| Citrobacter sp. | 2 | 2,98 | - | - | - | - | - | - | - | - | 2 | 2,98 |
| Escherichia coli | 13 | 19,40 | 4 | 6,00 | 1 | 1,49 | - | - | 1 | 1,49 | 19 | 28,38 |
| Edwardsiella sp. | 1 | 1,49 | 2 | 2,98 | 1 | 1,49 | 1 | 1,49 | - | - | 5 | 7,45 |
| Enterobacter sp. | 24 | 35,82 | 3 | 4,47 | 1 | 1,49 | - | - | - | - | 28 | 41,78 |
| Klebsiella sp. | 4 | 6,00 | - | - | 1 | 1,49 | 1 | 1,49 | - | - | 6 | 8,98 |
| Proteus sp. | - | - | 1 | 1,49 | 1 | 1,49 | 1 | 1,49 | - | - | 3 | 4,47 |
| Providencia sp. | 1 | 1,49 | - | - | 1 | 1,49 | - | - | - | - | 2 | 2,98 |
| Salmonella sp. | - | - | - | - | 1 | 1,49 | - | - | - | - | 1 | 1,49 |
| Serratia sp. | 1 | 1,49 | - | - | - | - | - | - | - | - | 1 | 1,49 |
| Total | 46 | 68,67 | 10 | 14,94 | 7 | 10,43 | 3 | 4,47 | 1 | 1,49 | 67 | 100,00 |
0R: Sensible a todos los antimicrobianos; 1R: Resistente a una clase de antimicrobianos; 2R: Resistente a dos clases de antimicrobianos; 3R: Resistente a tres clases de antimicrobianos; 4R: Resistente a cuatro clases de antimicrobianos; N: Número de muestras bacterianas. Señal convencional utilizada: - Dato numérico igual a cero, no resultante de redondeo.
DISCUSIÓN
La presencia de microorganismos resistentes a los antimicrobianos en el agua para consumo humano se caracteriza como un problema de salud pública, debido al peligro de propagación de cepas resistentes y la consiguiente colonización de individuos expuestos8. En este estudio, el género Enterobacter sp. fue el más aislado de aguas para el consumo humano, lo que puede estar relacionado con la capacidad de formación de biopelículas durante el proceso de tratamiento, lo que dificulta la acción del cloro en el tratamiento del agua31. Más de la mitad de las muestras de enterobacterias en esta investigación fueron resistentes a al menos una clase de antimicrobianos, un resultado inferior que el descrito en África y Europa2,32, en agua para consumo humano, y más alto que el encontrado por Gomes-Freitas et al.14 en Brasil.
La cefalotina y la cefoxitina (cefalosporinas de primera y segunda generación) fueron los medicamentos que tuvieron la tasa más alta de muestras bacterianas resistentes, posiblemente porque han sido ampliamente utilizados antibióticos durante muchos años para tratar infecciones comunitarias y hospitalarias y con mecanismos genéticos de resistencia bacteriana diseminada entre especies de la familia Enterobacteriaceae33. Se detectó resistencia a las cefalosporinas de tercera generación en cinco muestras y ninguna fue resistente a la cefepima, una cefalosporina de cuarta generación. Talukdar et al.13 informaron datos similares, en un estudio con E. coli con muestras de aguas entubadas de Daka, Bangladesh, donde describieron un 9% de resistencia a la cefotaxima, una cefalosporina de tercera generación. Aunque, en este estudio, la prevalencia de resistencia a las cefalosporinas más recientes haya sido baja, AbdelRahim et al.34 describieron el 81.5% de resistencia a la cefepima en muestras de enterobacterias aisladas en agua potable en Egipto. Las tasas de resistencia para el grupo de penicilina fueron bajas, similares a los datos descritos por Gomes-Freitas et al.14 en muestras de agua de dispensadores de agua potable en una escuela en el interior del estado de Goiás, Brasil.
Apenas dos muestras bacterianas fueron resistentes al ácido nalidíxico, número inferior al descrito por Talukdar et al.13 en la ciudad de Daca, Bangladesh, que presentó un total de 37% de E. coli resistentes a esa droga; aunque similar al obtenido por Coleman et al.10 con E. coli, que relataron apenas 0,5% de resistencia a ese antimicrobiano. Los mismos autores10 describen una tasa baja de resistencia para os aminoglucósidos, lo que diverge del presente estudio, que describió la totalidad de aislados sensibles a ese grupo de antibióticos. La resistencia al aztreonam se detectó en 5,97% de las muestras, dato inferior al descrito por Oliveira y Van Der Sand17 en aislados de aguas de superficies, en un estudio en el sur de Brasil.
La resistencia a tres o más clases de antimicrobianos se denomina multirresistencia y se caracteriza como un problema de salud pública porque reduce las opciones terapéuticas en un proceso infeccioso35. En este trabajo, una muestra de Proteus sp. y una de Salmonella sp. fueron resistentes a tres clases de antimicrobianos, respectivamente; y una muestra de E. coli mostró resistencia a cuatro clases de antimicrobianos. Los aislados multirresistentes se describen, con mayor frecuencia, en aguas residuales6, pero también se pueden aislar de agua de superficie y para consumo humano, como manantiales y agua mineral embotellada15,36, lo que puede exponer a la población a la colonización por bacterias resistentes y multirresistentes a antibióticos8. En un estudio con cepas de E. coli de soluciones alternativas, Coleman et al.10 describieron un 22% de resistencia a una o dos clases de antibióticos y un 14% de aislados multirresistentes, valores superiores a los descritos en este estudio.
Muchas bacterias se vuelven multirresistentes debido a la adquisición de elementos genéticos codificadores de enzimas que hidrolizan antibióticos, como es el caso de BLEE y carbapenemasas7. En este estudio, ninguna de las muestras produjo BLEE por el método fenotípico utilizado; sin embargo, los estudios han detectado la presencia de genes que codifican BLEE en varios grupos bacterianos aislados del agua en Brasil y en otros países6,15,22, así como ADN bacteriano extraído directamente del agua2,32,36. La ausencia de muestras productoras de BLEE en este estudio puede estar relacionada a la falta de acceso a técnicas moleculares capaces de detectar el gen productor de la enzima, que a menudo no puede expresarse fenotípicamente.
Un dato interesante obtenido en este estudio fue la presencia de mutaciones de enterobacterias con resistencia intermedia a los carbapenémicos. Una cepa de Proteus sp., aislada de una solución de alternativa de abastecimiento mostró resistencia intermedia a imipenem y ertapenem, uno de Enterobacter sp. y uno de E. coli al imipenem y uno de E. coli al meropenem. El aislado de bacterias resistentes a los carbapenémicos y la detección de genes que producen carbapenemasas ya se han descrito en aguas residuales, bahías y lagos en las Regiones Sur y Sudeste de Brasil16,18,19, lo que destaca la importancia de monitorear bacterias resistentes en el agua, como las captadas en el centro-oeste del estado de São Paulo.
CONCLUSIÓN
Los hallazgos descritos en este estudio muestran que las aguas para consumo pueden ser fuente de diseminación de bacterias gramnegativas resistentes y multirresistentes, lo que caracteriza un problema de salud pública, y destacan la importancia del tratamiento adecuado de las aguas, para impedir la exposición de la población a enterobacterias resistentes y multirresistentes, bien como del monitoreo de los sistemas de abastecimiento público, para la detección de bacterias resistentes a antimicrobianos.
REFERENCIAS
1 Winn Jr. WC, Allen SD, Janda WM, Koneman EW, Procop GW, Schreckenberger PC, et al. As Enterobacteriaceae. In: Koneman, diagnóstico microbiológico: texto e atlas colorido. 6. ed. Rio de Janeiro: Guanabara Koogan; 2008. p. 208-99. [ Links ]
2 Stange C, Sidhu JPS, Tiehm A, Toze S. Antibiotic resistance and virulence genes in coliform water isolates. Int J Hyg Environ Health. 2016 Nov;219(8):823-31. [ Links ]
3 Nordmann P. Carbapenemase-producing Enterobacteriaceae: overview of a major public health challenge. Med Mal Infect. 2014 Feb;44(2):51-6. [ Links ]
4 Bai X, Ma X, Xu F, Li J, Zhang H, Xiao X. The drinking water treatment process as a potential source of affecting the bacterial antibiotic resistance. Sci Total Environ. 2015 Nov; 533:24-31. [ Links ]
5 Woodford N, Wareham DW, Guerra B, Teale C. Carbapenemase-producing Enterobacteriaceae and non-Enterobacteriaceae from animals and the environment: an emerging public health risk of our own making? J Antimicrob Chemother. 2014 Feb;69(2):287-91. [ Links ]
6 Baquero F, Martínez JL, Cantón R. Antibiotics and antibiotic resistance in water environments. Curr Opin Biotechnol. 2008 Jun;19(3):260-5. [ Links ]
7 Potter RF, D'Souza AW, Dantas G. The rapid spread of carbapenem-resistant Enterobacteriaceae. Drug Resist Updat. 2016 Nov;29:30-46. [ Links ]
8 Lee J, Lee CS, Hugunin KM, Maute CJ, Dysko RC. Bacteria from drinking water supply and their fate in gastrointestinal tracts of germ-free mice: a phylogenetic comparison study. Water Res. 2010 Sep;44(17):5050-8. [ Links ]
9 Huddleston JR. Horizontal gene transfer in the human gastrointestinal tract: potential spread of antibiotic resistance genes. Infect Drug Resist. 2014 Jun;7:167-76. [ Links ]
10 Coleman BL, Louie M, Salvadori MI, McEwen SA, Neumann N, Sibley K, et al. Contamination of Canadian private drinking water sources with antimicrobial resistant Escherichia coli. Water Res. 2013 Jun;47(9):3026-36. [ Links ]
11 Abera B, Kibret M, Mulu W. Extended-Spectrum beta (β)-Lactamases and antibiogram in Enterobacteriaceae from clinical and drinking water sources from Bahir Dar City, Ethiopia. PLoS One. 2016 Nov;11(11):e0166519. [ Links ]
12 Subba P, Joshi DR, Bhatta DR. Antibiotic resistance pattern and plasmid profiling of thermotolerant Escherichia coli isolates in drinking water. J Nepal Health Res Counc. 2013 Jan;11(23):44-8. [ Links ]
13 Talukdar PK, Rahman M, Rahman M, Nabi A, Islam Z, Hoque MM, et al. Antimicrobial resistance, virulence factors and genetic diversity of Escherichia coli isolates from household water supply in Dhaka, Bangladesh. PLoS One. 2013;8(4):e61090. [ Links ]
14 Gomes-Freitas D, Silva RDR, Bataus LAM, Barbosa MS, Braga CASB, Carneiro LC. Bacteriological water quality in school's drinking fountains and detection antibiotic resistance genes. Ann Clin Microbiol Antimicrob. 2017 Feb;16(1):5. [ Links ]
15 Falcone-Dias MF, Centrón D, Pavan F, Moura ACS, Naveca FG, Souza VC, et al. Opportunistic pathogens and elements of the resistome that are common in bottled mineral water support the need for continuous surveillance. PloS One. 2015 Mar;10(3):e0121284. [ Links ]
16 Coutinho FH, Silveira CB, Pinto LH, Salloto GRB, Cardoso AM, Martins OB, et al. Antibiotic resistance is widespread in urban aquatic environments of Rio de Janeiro, Brazil. Microb Ecol. 2014 Oct;68(3):441-52. [ Links ]
17 Oliveira DV, Van Der Sand ST. Phenotypic tests for the detection of ß-lactamase-producing Enterobacteriaceae isolated from different environments. Curr Microbiol. 2016 Jul;73(1): 132-8. [ Links ]
18 Conte D, Palmeiro JK, Nogueira KS, Lima TMR, Cardoso MA, Pontarolo R, et al. Characterization of CTX-M enzymes, quinolone resistance determinants, and antimicrobial residues from hospital sewage, wastewater treatment plant, and river water. Ecotoxicol Environ Saf. 2017 Feb;136:62-9. [ Links ]
19 Picão RC, Cardoso JP, Campana EH, Nicoletti AG, Petrolini FVB, Assis DM, et al. The route of antimicrobial resistance from the hospital effluent to the environment: focus on the occurrence of KPC-producing Aeromonas spp. and Enterobacteriaceae in sewage. Diagn Microbiol Infect Dis. 2013 May;76(1):80-5. [ Links ]
20 Oliveira S, Moura RA, Silva KC, Pavez M, McCulloch JA, Dropa M, et al. Isolation of KPC-2-producing Klebsiella pneumoniae strains belonging to the high-risk multiresistant clonal complex 11 (ST437 and ST340) in urban rivers. J Antimicrob Chemother. 2014 Mar;69(3):849-52. [ Links ]
21 Berglund, B. Environmental dissemination of antibiotic resistance genes and correlation to anthropogenic contamination with antibiotics. Infect Ecol Epidemiol. 2015 Sep;5:28564. [ Links ]
22 Su HC, Liu YS, Pan CG, Chen J, He LY, Ying GG. Persistence of antibiotic resistance genes and bacterial community changes in drinking water treatment system: from drinking water source to tap water. Sci Total Environ. 2018 Mar;616-617:453-61. [ Links ]
23 Caltagirone M, Nucleo E, Spalla M, Zara F, Novazzi F, Marchetti VM, et al. Occurrence of extended spectrum β-lactamases, KPC-type, and MCR-1.2-producing Enterobacteriaceae from wells, river water, and wastewater treatment plants in Oltrepò Pavese area, Northern Italy. Front Microbiol. 2017 Nov;8:2232. [ Links ]
24 Madec JY, Haenni M, Ponsin C, Kieffer N, Rion E, Gassilloud B. Sequence type 48 Escherichia coli carrying the blaCTX-M-1 IncI1/ST3 plasmid in drinking water in France. Antimicrob Agents Chemother. 2016 Sep;60(10):6430-2. [ Links ]
25 Rathinasabapathi P, Hiremath DS, Arunraj R, Parani M. Molecular detection of New Delhi metallo-beta-lactamase-1 (NDM-1) positive bacteria from environmental and drinking water samples by loop mediated isothermal amplification of bla NDM-1 . Indian J Microbiol. 2015 Dec;55(4): 400-5. [ Links ]
26 Pessôa GVA, Silva EAM. Meios de Rugai e lisina-motilidade combinados em um só tubo para a identificação presuntiva de enterobactérias. Rev Inst Adolfo Lutz. 1972;32:97-100. [ Links ]
27 Bauer AW, Kirby WM, Sherris JC, Turck M. Antibiotic susceptibility testing by a standardized single disk method. Am J Clin Pathol. 1966 Apr;45(4): 493-6. [ Links ]
28 Clinical and Laboratory Standards Institute. Performance standards for antimicrobial susceptibility testing. Approved Standard M100-S22. Wayne: CLSI; 2017. [ Links ]
29 Clinical and Laboratory Standards Institute. Performance standards for antimicrobial susceptibility testing. Approved Standard M100-S22. Wayne: CLSI; 2015. [ Links ]
30 Gales AC, Frigatto EAM, Andrade SS. Teste de sensibilidade aos antimicrobianos [Internet]. Brasília: Agência Nacional de Vigilância Sanitária; 2008 [citado 2017 fev 28]. Disponível em: Disponível em: http://www.anvisa.gov.br/servicosaude/controle/rede_rm/cursos/boas_praticas/modulo5/gram_negativos4.htm . [ Links ]
31 Herson DS, McGonigle B, Payer MA, Baker KH. Attachment as a factor in the protection of Enterobacter cloacae from chlorination. Appl Environ Microbiol. 1987 May;53(5):1178-80. [ Links ]
32 Lyimo B, Buza J, Subbiah M, Smith W, Call DR. Comparison of antibiotic resistant Escherichia coli obtained from drinking water sources in northern Tanzania: a cross-sectional study. BMC Microbiol. 2016 Nov;16(1):254. [ Links ]
33 von Wintersdorff CJ, Penders J, van Niekerk JM, Mills ND, Majumder S, van Alphen LB, et al. Dissemination of antimicrobial resistance in microbial ecosystems through horizontal gene transfer. Front Microbiol. 2016 Feb;7:173. [ Links ]
34 AbdelRahim KAA, Hassanein AM, Abd El Azeiz HAEH. Prevalence, plasmids and antibiotic resistance correlation of enteric bacteria in different drinking water resources in Sohag, Egypt. Jundishapur J Microbiol. 2015 Jan;8(1):e18648. [ Links ]
35 Magiorakos AP, Srinivasan A, Carey RB, Carmeli Y, Falagas ME, Giske CG, et al. Multidrug-resistant, extensively drug-resistant and pandrug-resistant bacteria: an international expert proposal for interim standard definitions for acquired resistance. Clin Microbiol Infect. 2012 Mar;18(3):268-81. [ Links ]
36 Li S, Zhu ZC, Wang L, Zhou YF, Tang YJ, Miao ZM. Prevalence and characterization of extended-spectrum beta-lactamase-producing Enterobacteriaceae in spring waters. Lett Appl Microbiol. 2015 Dec;61(6):544-8. [ Links ]
Cómo citar este artículo / How to cite this article: Martins A, Silva RA, Ferreira LO, Licate MM, Delafiori CR, Pôrto SF. Resistencia a antimicrobianos de enterobacterias aisladas de aguas destinadas al abastecimiento público en la región centro-oeste del estado de São Paulo, Brasil. Rev Pan Amaz Saude. 2019;10:e201900065. Doi: http://dx.doi.org/10.5123/S2176-6223201900065
Recibido: 25 de Junio de 2018; Aprobado: 10 de Enero de 2019










 texto em
texto em